ИИ в обработке изображений микроскопа
ИИ революционизирует обработку изображений микроскопа, предлагая мощные возможности, такие как точная сегментация, снижение шума, сверхразрешение и автоматизированное получение изображений. В статье освещаются ключевые инструменты ИИ и новые тенденции в научных исследованиях.
Методы ИИ революционизируют микроскопию, оптимизируя получение изображений и автоматизируя анализ. В современных умных микроскопах модули ИИ могут в реальном времени настраивать параметры съемки (например, фокус, освещение), чтобы минимизировать фотоблеклость и улучшить сигнал. Тем временем алгоритмы глубокого обучения способны анализировать сложные данные изображений, извлекая скрытые биологические сведения и даже связывая изображения с другими данными (например, геномикой).
Методы ИИ: машинное обучение против глубокого обучения
Методы ИИ варьируются от классического машинного обучения (МО) до современного глубокого обучения (ГО). Каждый подход имеет свои сильные и слабые стороны:
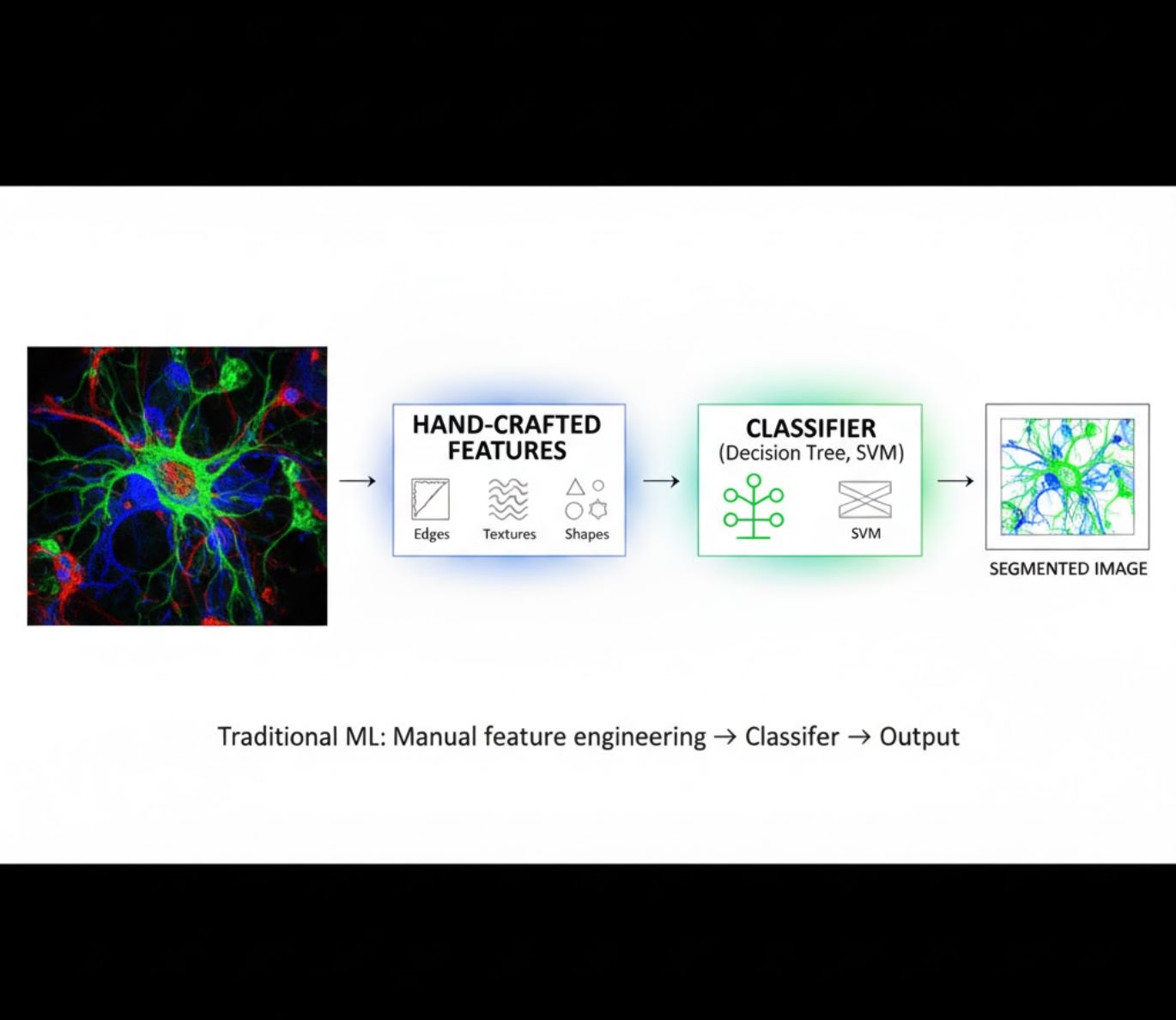
Ручное создание признаков
- Исследователи вручную выделяют признаки изображения (грани, текстуры, формы)
- Признаки подаются в классификаторы (деревья решений, SVM)
- Быстрое обучение
- Сложности с комплексными или шумными изображениями
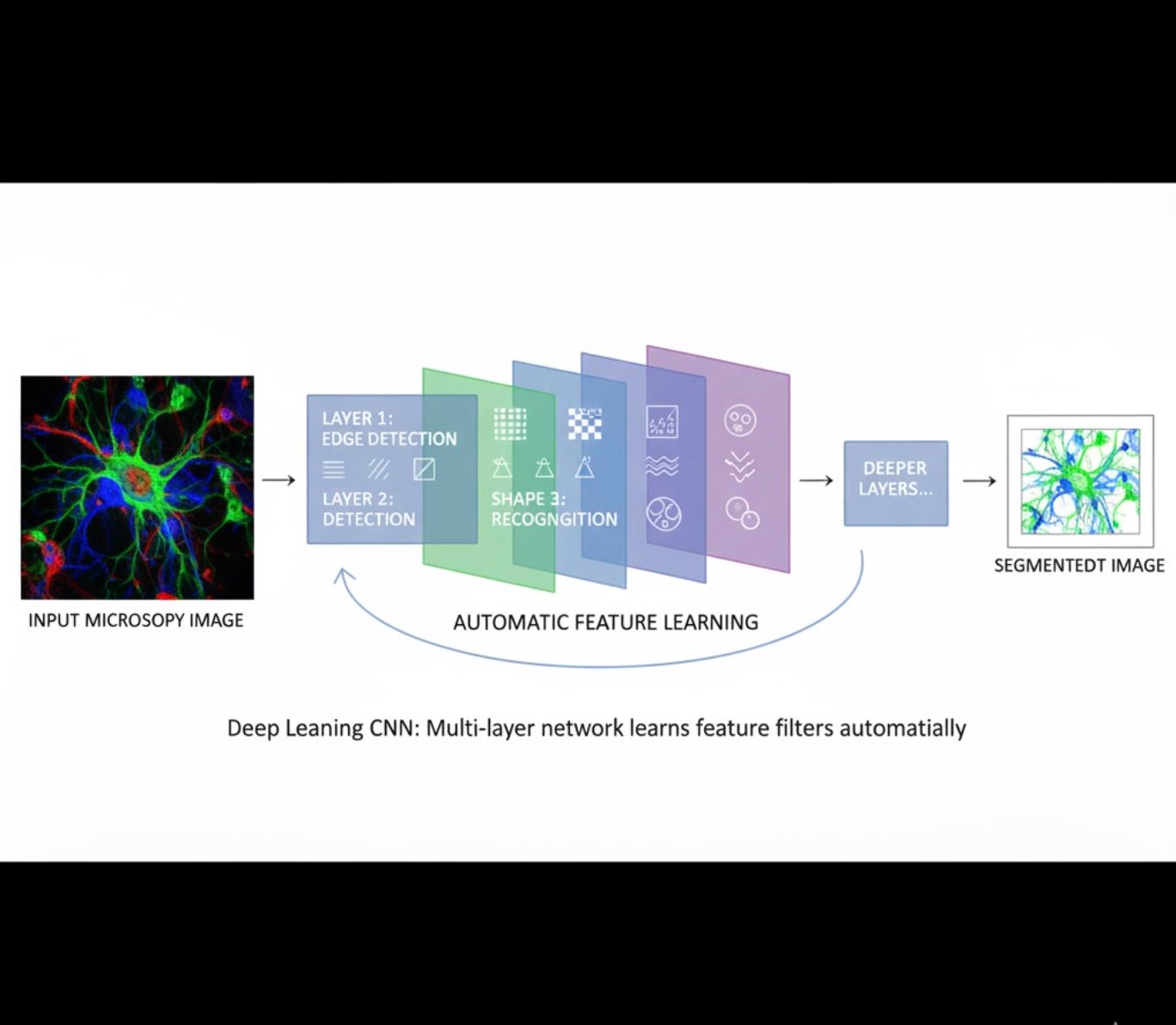
Автоматическое обучение признаков
- Многослойные нейронные сети (CNN) автоматически обучаются признакам
- Обучение от сырых пикселей «от конца до конца»
- Гораздо более устойчиво к вариациям
- Надежно захватывает сложные текстуры и структуры
Как работают CNN: Свёрточные нейронные сети применяют последовательные фильтры к микроскопическим изображениям, обучаясь распознавать простые паттерны (грани) на ранних слоях и сложные структуры (формы клеток, текстуры) на глубоких слоях. Такая иерархическая обучаемость делает глубокое обучение особенно устойчивым даже при значительных изменениях интенсивности.
Визуальное сравнение: конвейеры МО и ГО
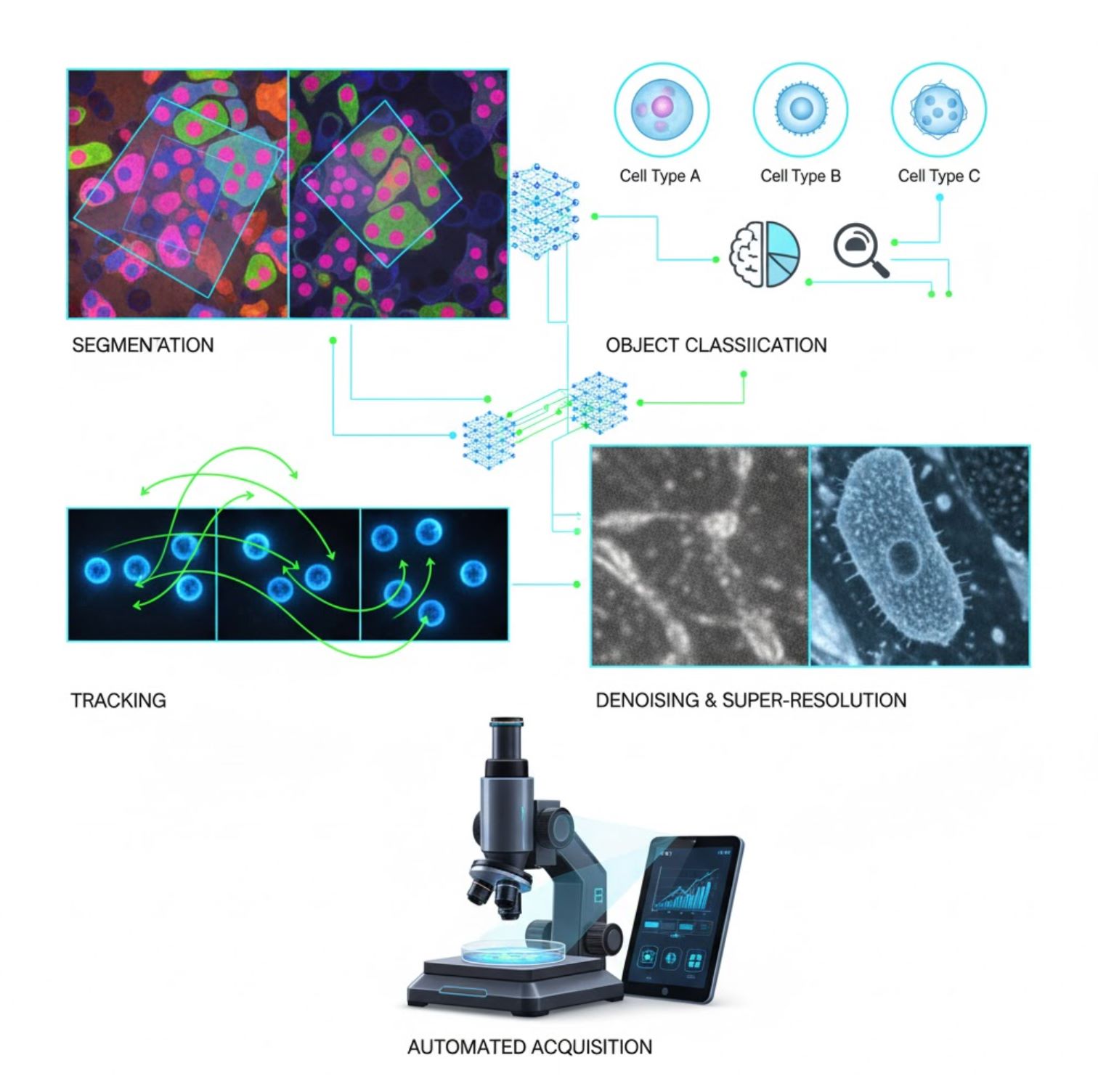
Ключевые приложения ИИ в микроскопии
ИИ сейчас встроен во многие задачи обработки изображений на всех этапах микроскопии:
Сегментация
Разделение изображений на области (например, идентификация каждой клетки или ядра). Глубокие сети, такие как U-Net, отлично справляются с этой задачей.
- Семантическая сегментация: метки классов для каждого пикселя
- Инстанс-сегментация: разделение отдельных объектов
- Высокая точность на плотных или тусклых изображениях
- Модели фундаментального зрения (например, μSAM) теперь адаптированы для микроскопии
Классификация объектов
После сегментации ИИ классифицирует каждый объект с высокой точностью.
- Определение типа клеток
- Определение стадии митоза
- Обнаружение патологических индикаторов
- Выделение тонких фенотипов, трудно поддающихся ручной оценке
Трекинг
В микроскопии с временной съемкой ИИ отслеживает клетки или частицы между кадрами с беспрецедентной точностью.
- Глубокое обучение значительно улучшает точность трекинга
- Обеспечивает надежный анализ движущихся клеток
- Фиксирует динамические биологические процессы
Снижение шума и сверхразрешение
Модели ИИ улучшают качество изображений, удаляя шум и размытость.
- Глубокие модели с учетом физики изучают оптику микроскопа
- Восстанавливают более четкие, безартефактные изображения
- Более высокое разрешение с меньшим количеством артефактов по сравнению с традиционными методами
Автоматизированное получение изображений
ИИ управляет самим микроскопом в реальном времени.
- Анализирует живые изображения для принятия интеллектуальных решений
- Автоматически настраивает фокус и сканирует интересующие области
- Снижает фототоксичность и экономит время
- Обеспечивает высокопроизводительные и адаптивные эксперименты
Популярные инструменты ИИ для обработки изображений микроскопа
Богатая экосистема инструментов поддерживает ИИ в микроскопии. Исследователи создали как универсальное, так и специализированное программное обеспечение, многие из которых являются открытым исходным кодом:
Cellpose
| Разработчик | Карсен Стрингер и Мариус Пачитариу (исследовательская группа MouseLand) |
| Поддерживаемые платформы |
Требуется Python (установка через pip/conda). Графический интерфейс доступен только на настольных платформах. |
| Поддержка языков | Документация на английском; широко используется в исследовательских лабораториях по всему миру |
| Модель ценообразования | Бесплатно и с открытым исходным кодом по лицензии BSD-3-Clause |
Обзор
Cellpose — это продвинутый инструмент сегментации на основе глубокого обучения, предназначенный для микроскопических изображений. Как универсальный алгоритм, он точно сегментирует разнообразные типы клеток (ядра, цитоплазма и др.) в различных режимах визуализации без необходимости переобучения модели. Благодаря функциям с участием пользователя исследователи могут уточнять результаты, адаптировать модель под свои данные и применять систему как для 2D, так и для 3D изображений.
Ключевые особенности
Работают «из коробки» для широкого спектра типов клеток, красителей и методов визуализации без дополнительного обучения.
Поддержка полноценных 3D стеков с использованием подхода «2.5D», который повторно использует 2D модели для объемных данных.
Ручная корректировка результатов сегментации и переобучение модели на собственных данных для повышения точности.
Доступ через Python API, командную строку или графический интерфейс для гибких рабочих процессов.
Функции шумоподавления, устранения размытия и повышения разрешения для улучшения качества изображений перед сегментацией.
Скачать или получить доступ
Техническая информация
Cellpose был представлен в ключевом исследовании Стрингера, Ванга, Майклоса и Пачитариу, обучен на большом и очень разнообразном наборе данных, содержащем более 70 000 сегментированных объектов. Такое разнообразие позволяет модели обобщать информацию о формах, размерах клеток и условиях микроскопии, значительно снижая необходимость в дополнительном обучении для большинства случаев. Для 3D данных Cellpose умело повторно использует свою 2D модель в режиме «2.5D», избегая необходимости в полностью 3D размеченных данных, при этом обеспечивая объемную сегментацию. В версии Cellpose 2.0 была введена функция переобучения с участием пользователя, позволяющая вручную корректировать предсказания и переобучать модель на собственных изображениях для улучшения результатов на конкретных наборах данных.
Установка и настройка
Настройте Python-окружение с помощью conda:
conda create -n cellpose python=3.10Активируйте окружение и установите Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeНачало работы
Режим GUI
- Запустите графический интерфейс, выполнив:
python -m cellpose - Перетащите файлы изображений (
.tif,.pngи др.) в окно программы - Выберите тип модели (например, «cyto» для цитоплазмы или «nuclei» для ядер)
- Установите предполагаемый диаметр клетки или позвольте Cellpose выполнить автокалибровку
- Нажмите для начала сегментации и просмотра результатов
Режим Python API
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Уточнение и переобучение
- После генерации масок вручную исправьте сегментацию в GUI, объединяя или удаляя маски
- Используйте встроенные функции обучения для переобучения на исправленных примерах
- Повышение точности модели на вашем конкретном наборе данных
Обработка 3D данных
- Загрузите многоуровневый TIFF или объемный стек
- Используйте флаг
--Zstackв GUI или API для обработки как 3D - При необходимости уточните 3D потоки с помощью сглаживания или специальных параметров для улучшения сегментации
Ограничения и особенности
- Компромисс универсальности модели: Несмотря на широкую применимость универсальной модели, для очень необычных форм клеток или условий визуализации может потребоваться переобучение.
- Затраты на разметку: Обучение с участием пользователя требует ручных исправлений, что может занимать много времени при больших наборах данных.
- Сложность установки: Установка GUI может потребовать работы с командной строкой, conda-окружениями и управлением зависимостями Python — не всегда просто для непрофессионалов.
- Только для настольных ПК: Cellpose предназначен для использования на настольных компьютерах; нативных приложений для Android или iOS нет.
Часто задаваемые вопросы
Нет — Cellpose предоставляет предобученные универсальные модели, которые часто хорошо работают без переобучения. Однако для оптимальных результатов на специальных или необычных данных вы можете разметить и переобучить модель с помощью функций с участием пользователя.
Да — он поддерживает 3D, повторно используя свою 2D модель (так называемый «2.5D»), и вы можете обрабатывать объемные стеки через GUI или API.
GPU настоятельно рекомендуется для ускорения вывода и обучения, особенно на больших или 3D наборах данных, но Cellpose может работать и на машинах с только CPU, хотя и с меньшей производительностью.
В GUI установите предполагаемый диаметр клетки вручную или позвольте Cellpose выполнить автоматическую калибровку. Вы можете уточнять результаты и переобучать модель, если сегментация недостаточно точна.
Да — в новых версиях (Cellpose 3) включены модели восстановления изображений для шумоподавления, устранения размытия и повышения разрешения, чтобы улучшить качество сегментации перед обработкой.
StarDist
| Разработчики | Уве Шмидт, Мартин Вайгерт, Коулман Броаддус и Джин Майерс |
| Поддерживаемые платформы |
|
| Поддержка языков | Проект с открытым исходным кодом, документация и сообщество преимущественно на английском языке |
| Модель лицензирования | Бесплатно и с открытым исходным кодом. Лицензия BSD-3-Clause |
Обзор
StarDist — это инструмент глубокого обучения для сегментации отдельных объектов на микроскопических изображениях. Он представляет каждый объект (например, ядра клеток) в виде звездообразного выпуклого многоугольника в 2D или многогранника в 3D, что позволяет точно обнаруживать и разделять плотно расположенные или перекрывающиеся объекты. Благодаря своей надёжной архитектуре StarDist широко используется для автоматической сегментации клеток и ядер в флуоресцентной микроскопии, гистопатологии и других приложениях биомедицинского анализа изображений.
Основные возможности
Высокоточная сегментация отдельных объектов с использованием звездообразных выпуклых многоугольников (2D) и многогранников (3D) для надёжного обнаружения объектов.
Специализированные модели для 2D изображений и 3D объемных данных для комплексного анализа микроскопии.
Готовые к использованию модели для флуоресцентных ядер, гистологии с окраской H&E и других распространённых сценариев визуализации.
Классификация обнаруженных объектов по различным классам (например, разные типы клеток) в одном запуске сегментации.
Бесшовная интеграция с ImageJ/Fiji, QuPath и napari для удобных рабочих процессов с графическим интерфейсом.
Полный набор метрик для оценки сегментации объектов, включая точность, полноту, F1-меру и паноптическое качество.
Технические детали
Первоначально представленная в статье MICCAI 2018 года, основная инновация StarDist — предсказание радиальных расстояний вдоль фиксированных лучей в сочетании с вероятностью объекта для каждого пикселя, что позволяет точно восстанавливать звездообразные выпуклые формы. Такой подход надёжно сегментирует плотно прилегающие объекты, которые сложно разделить традиционными методами на основе пикселей или ограничивающих рамок.
Недавние разработки расширили применение StarDist на гистопатологические изображения, позволяя не только сегментировать ядра, но и выполнять мультиклассовую классификацию обнаруженных объектов. Метод показал высокие результаты в конкурсах, таких как CoNIC (Colon Nuclei Identification and Counting).
Скачать или получить доступ
Установка и настройка
Установите TensorFlow (версии 1.x или 2.x) как предварительное условие для StarDist.
Используйте pip для установки Python-пакета StarDist:
pip install stardistДля napari:
pip install stardist-napariДля QuPath: Установите расширение StarDist, перетащив файл .jar в QuPath.
Для ImageJ/Fiji: Используйте встроенный менеджер плагинов или установку вручную через меню плагинов.
Запуск сегментации
Загрузите предварительно обученную модель, нормализуйте изображение и выполните предсказание:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Откройте изображение в napari, выберите плагин StarDist, выберите предварительно обученную или пользовательскую модель и запустите предсказание прямо из графического интерфейса.
Используйте плагин StarDist из меню Плагины для применения модели к стеку изображений с интуитивно понятным интерфейсом.
После установки расширения запустите обнаружение StarDist через консоль скриптов или графический интерфейс QuPath для анализа гистопатологии.
Обучение и донастройка
Создайте изображения с истинными метками, где каждый объект размечен уникальной маской. Используйте инструменты аннотации, такие как LabKit, QuPath или Fiji, для подготовки набора данных.
Используйте Python API StarDist для обучения новой модели или донастройки существующей на ваших собственных размеченных данных.
Опции постобработки
- Применение подавления немаксимумов (NMS) для устранения избыточных кандидатов
- Использование StarDist OPP (постобработка объектов) для объединения масок для форм, не являющихся звездообразно выпуклыми
Ограничения и особенности
- Предположение о звездообразной выпуклости может не идеально моделировать сильно невыпуклые или очень неправильные формы
- Сложность установки: для пользовательских сборок требуется совместимый компилятор C++ для сборки расширений
- Аппаратное ускорение на GPU зависит от совместимых версий TensorFlow, CUDA и cuDNN
- Некоторые пользователи сообщают о проблемах с запуском плагина ImageJ из-за конфигурации Java
Часто задаваемые вопросы
StarDist работает с различными типами изображений, включая флуоресцентные, светлопольные и гистопатологические (например, H&E), благодаря гибким предварительно обученным моделям и адаптивности к разным методам визуализации.
Да — StarDist поддерживает 3D сегментацию отдельных объектов с использованием звездообразных выпуклых многогранников для объёмных данных, расширяя возможности 2D до полноценного 3D анализа.
Не обязательно. Предварительно обученные модели доступны и часто хорошо работают из коробки. Однако для специализированных или новых данных аннотирование и обучение собственных моделей значительно повышает точность.
StarDist интегрируется с napari, ImageJ/Fiji и QuPath, позволяя запускать сегментацию через графический интерфейс без программирования. Также поддерживается прямое использование Python API для продвинутых рабочих процессов.
StarDist предоставляет встроенные функции для вычисления распространённых метрик сегментации объектов, включая точность, полноту, F1-меру и паноптическое качество, для оценки производительности сегментации.
SAM
Информация о приложении
| Разработчик | Meta AI Research (FAIR) |
| Поддерживаемые устройства |
|
| Язык и доступность | Открытая базовая модель, доступная по всему миру; документация на английском языке |
| Стоимость | Бесплатно — с открытым исходным кодом по лицензии Meta через GitHub и интеграцию в MIB |
Общий обзор
SAM (Segment Anything Model) — мощная базовая модель ИИ, созданная Meta, которая обеспечивает интерактивную и автоматическую сегментацию практически любого объекта на изображениях. Используя подсказки, такие как точки, ограничивающие рамки или грубые маски, SAM генерирует маски сегментации без необходимости переобучения для конкретных задач. В микроскопических исследованиях гибкость SAM была адаптирована для сегментации клеток, обнаружения органелл и анализа гистопатологии, предлагая масштабируемое решение для исследователей, которым нужен универсальный инструмент сегментации с поддержкой подсказок.
Подробное введение
Изначально обученная Meta на более чем 1 миллиарде масок на 11 миллионах изображений, SAM была разработана как базовая модель с поддержкой подсказок и «zero-shot» производительностью на новых доменах. В исследованиях медицинской визуализации SAM оценивалась для сегментации целых слайдов патологии, обнаружения опухолей и идентификации ядер клеток. Однако её производительность на плотно расположенных объектах — таких как ядра клеток — неоднозначна: даже при обширных подсказках (например, 20 кликов или рамок) сегментация zero-shot может испытывать трудности на сложных микроскопических изображениях.
Для решения этой проблемы появились адаптации, специфичные для области:
- SAMCell — дообучена на больших микроскопических наборах данных для надёжной сегментации zero-shot различных типов клеток без необходимости переобучения для каждого эксперимента
- μSAM — переобучена на более чем 17 000 вручную аннотированных микроскопических изображениях для повышения точности на мелких клеточных структурах
Ключевые особенности
Гибкое взаимодействие с использованием точек, рамок и масок для точного управления.
Выполняет сегментацию без дообучения на новых доменах изображений.
Адаптируется для микроскопии и гистопатологии с помощью few-shot или подсказочного переобучения.
Доступна в Microscopy Image Browser (MIB) с поддержкой 3D и интерполированной сегментации.
IDCC-SAM обеспечивает автоматический подсчёт клеток в иммунноцитохимии без ручной аннотации.
Загрузка или доступ
Руководство пользователя
- Откройте Microscopy Image Browser и перейдите в панель сегментации SAM
- Настройте интерпретатор Python и выберите модели SAM-1 или SAM-2
- Для ускорения на GPU выберите «cuda» в среде выполнения (рекомендуется для оптимальной производительности)
- Подсказки точками: Кликните по объекту для определения положительной точки; используйте Shift + клик для расширения и Ctrl + клик для отрицательных точек
- 3D стеки: Используйте интерактивный 3D режим — кликните по одному срезу, прокрутите с Shift и интерполируйте точки между срезами
- Режим корректировки: Заменяйте, добавляйте, вычитайте маски или создавайте новый слой по необходимости
- Используйте опцию «Автоматически всё» в панели SAM-2 MIB для сегментации всех видимых объектов в области
- Просматривайте и при необходимости уточняйте маски после сегментации
- Используйте подсказочные пайплайны дообучения (например, «All-in-SAM») для генерации аннотаций на уровне пикселей из разреженных пользовательских подсказок
- Для подсчёта клеток применяйте IDCC-SAM, который использует SAM в zero-shot режиме с последующей обработкой
- Для высокоточной сегментации клеток используйте SAMCell, дообученную на микроскопических изображениях клеток
Ограничения и особенности
- Zero-shot производительность нестабильна на плотных или перекрывающихся структурах без дообучения
- Качество сегментации сильно зависит от стратегии и типа подсказок
- Настоятельно рекомендуется GPU; работа на CPU очень медленная
- Испытывает трудности с очень высокоразрешёнными изображениями целых слайдов и многоуровневыми тканевыми структурами
- Дообучение или адаптация SAM для микроскопии может требовать навыков машинного обучения
Часто задаваемые вопросы
Да — через адаптации, такие как SAMCell, которая дообучает SAM на микроскопических наборах данных специально для задач сегментации клеток.
Не всегда. С помощью IDCC-SAM можно выполнять подсчёт клеток в режиме zero-shot без ручных аннотаций.
Используйте подсказочное дообучение (например, «All-in-SAM») или предобученные микроскопические версии, такие как μSAM, обученную на более чем 17 000 аннотированных микроскопических изображениях.
Хотя возможно использование на CPU, GPU настоятельно рекомендуется для практической скорости вывода и интерактивной сегментации в реальном времени.
Да — интеграция SAM-2 в MIB поддерживает 3D сегментацию с интерполяцией точек между срезами для объемного анализа.
AxonDeepSeg
| Разработчик | Лаборатория NeuroPoly при Polytechnique Montréal и Университете Монреаля |
| Поддерживаемые платформы |
|
| Язык | Документация на английском; инструмент с открытым исходным кодом, используемый по всему миру |
| Стоимость | Бесплатно и с открытым исходным кодом |
Обзор
AxonDeepSeg — это инструмент на базе ИИ для автоматической сегментации аксонов и миелина на микроскопических изображениях. Используя сверточные нейронные сети, он обеспечивает точную сегментацию по трем классам (аксон, миелин, фон) для различных методов визуализации, включая TEM, SEM и светлопольную микроскопию. Автоматизация морфометрических измерений, таких как диаметр аксона, g-коэффициент и толщина миелина, упрощает количественный анализ в нейронауках, значительно сокращая время ручной разметки и повышая воспроизводимость.
Ключевые особенности
Готовые к использованию модели, оптимизированные для TEM, SEM и светлопольной микроскопии.
Точное определение областей аксона, миелина и фона на микроскопических изображениях.
Автоматический расчет диаметра аксона, g-коэффициента, толщины миелина и показателей плотности.
Интеграция с Napari GUI позволяет вручную уточнять маски сегментации для повышения точности.
Бесшовная интеграция в пользовательские конвейеры для масштабного анализа нервной ткани.
Полный набор тестов обеспечивает воспроизводимость и надежность результатов сегментации.
Технические детали
Разработанный лабораторией NeuroPoly, AxonDeepSeg использует глубокое обучение для высокоточной сегментации в нейронаучных приложениях. Предварительно обученные модели доступны для различных методов микроскопии, обеспечивая универсальность при работе с разными техниками визуализации. Инструмент интегрируется с Napari, что позволяет интерактивно корректировать маски сегментации и повышать точность на сложных наборах данных. AxonDeepSeg вычисляет ключевые морфометрические показатели, поддерживая масштабные исследования структуры и патологии нервной ткани. Его Python-фреймворк позволяет интегрировать инструмент в пользовательские конвейеры для анализа морфологии аксонов и миелина.
Скачать или получить доступ
Установка и настройка
Убедитесь, что установлен Python версии 3.8 или выше, затем установите AxonDeepSeg и Napari с помощью pip:
pip install axondeepseg napariЗапустите предоставленные тестовые скрипты, чтобы убедиться, что все компоненты установлены и работают корректно.
Импортируйте микроскопические изображения (TEM, SEM или светлопольные) в Napari или вашу среду Python.
Выберите подходящую предварительно обученную модель для вашего типа микроскопии и запустите сегментацию для создания масок аксонов и миелина.
Автоматически вычислите морфометрические параметры, включая диаметр аксона, g-коэффициент, плотность и толщину миелина, затем экспортируйте результаты в формате CSV.
Используйте графический интерфейс Napari для ручной корректировки масок сегментации, объединяя или удаляя маски для повышения точности.
Важные замечания
- Производительность может снижаться на новых или не обученных методах визуализации
- Для сложных или проблемных областей может потребоваться ручная коррекция
- Для ускорения обработки больших наборов данных рекомендуется GPU; поддерживается также обработка на CPU
Часто задаваемые вопросы
AxonDeepSeg поддерживает TEM (просвечивающая электронная микроскопия), SEM (сканирующая электронная микроскопия) и светлопольную микроскопию с предварительно обученными моделями, оптимизированными для каждого метода.
Да, AxonDeepSeg полностью бесплатен и с открытым исходным кодом, доступен для академического и коммерческого использования.
Да, AxonDeepSeg автоматически рассчитывает диаметр аксона, g-коэффициент, толщину миелина и показатели плотности на основе сегментированных изображений.
GPU рекомендуется для ускорения сегментации больших наборов данных, но обработка на CPU также поддерживается для небольших анализов.
Да, интеграция с графическим интерфейсом Napari позволяет интерактивно корректировать и уточнять маски сегментации для повышения точности на сложных участках.
Ilastik
| Разработчик | Команда Ilastik при Европейской лаборатории молекулярной биологии (EMBL) и связанные академические партнеры |
| Поддерживаемые платформы |
|
| Язык | Английский |
| Стоимость | Бесплатно и с открытым исходным кодом |
Обзор
Ilastik — мощный инструмент на базе ИИ для интерактивной сегментации, классификации и анализа микроскопических изображений. Используя методы машинного обучения, такие как классификаторы Random Forest, он позволяет исследователям сегментировать пиксели, классифицировать объекты, отслеживать клетки во времени и выполнять подсчет плотности как в 2D, так и в 3D наборах данных. Благодаря интуитивно понятному интерфейсу и обратной связи в реальном времени Ilastik доступен учёным без навыков программирования и широко используется в клеточной биологии, нейронауках и биомедицинской визуализации.
Ключевые возможности
Обратная связь в реальном времени при аннотировании представительных областей для мгновенных результатов сегментации.
Категоризация сегментированных структур на основе морфологических и интенсивностных признаков.
Отслеживание движения и деления клеток в 2D и 3D временных микроскопических экспериментах.
Квантование густонаселённых областей без явной сегментации отдельных объектов.
Полуавтоматическая сегментация сложных 3D объемов с интуитивным взаимодействием.
Автоматическая обработка множества изображений с использованием командного режима без графического интерфейса.
Загрузка
Руководство по началу работы
Скачайте Ilastik для вашей операционной системы с официального сайта. Пакет включает все необходимые зависимости Python, следуйте инструкциям по установке для вашей платформы.
Откройте Ilastik и выберите нужный рабочий процесс анализа: классификация пикселей, классификация объектов, отслеживание или подсчет плотности. Загрузите ваш набор изображений, который может включать многоканальные, 3D или временные изображения.
Отметьте несколько представительных пикселей или объектов на изображениях. Классификатор Random Forest Ilastik обучается на этих аннотациях и автоматически предсказывает метки для всего набора данных.
Примените обученную модель для сегментации или классификации всего набора данных. Экспортируйте результаты в виде размеченных изображений, карт вероятностей или количественных таблиц для дальнейшего анализа и визуализации.
Используйте безголовый режим Ilastik для автоматической обработки множества изображений без ручного вмешательства, что идеально подходит для масштабных аналитических конвейеров.
Ограничения и особенности
- Интерактивная разметка может занимать много времени при работе с очень большими наборами данных
- Точность зависит от качества и репрезентативности аннотаций пользователя
- Требования к памяти — очень высокое разрешение или многогигабайтные наборы данных могут требовать значительного объема оперативной памяти
- Сложные данные — классификаторы Random Forest могут уступать глубоким нейронным сетям при работе с сильно вариабельными или сложными изображениями
Часто задаваемые вопросы
Да, Ilastik полностью поддерживает 3D объемы и временные эксперименты для сегментации, отслеживания и количественного анализа на нескольких временных точках.
Да, Ilastik полностью бесплатен и с открытым исходным кодом, доступен для всех пользователей без лицензионных ограничений.
Нет, Ilastik предоставляет интуитивно понятный графический интерфейс с обратной связью в реальном времени, что делает его доступным для исследователей без навыков программирования. Продвинутые пользователи также могут использовать пакетную обработку через командную строку.
Да, специализированный рабочий процесс отслеживания позволяет анализировать движение и деление клеток как в 2D, так и в 3D временных наборах данных с автоматическим отслеживанием родословных.
Результаты сегментации можно экспортировать в виде размеченных изображений, карт вероятностей или количественных таблиц, что обеспечивает бесшовную интеграцию с инструментами для дальнейшего анализа и визуализации.
Эти инструменты подходят как новичкам, так и экспертам. Многие из них бесплатны и с открытым исходным кодом, что облегчает воспроизводимость и совместное использование рабочих процессов ИИ в научном сообществе.
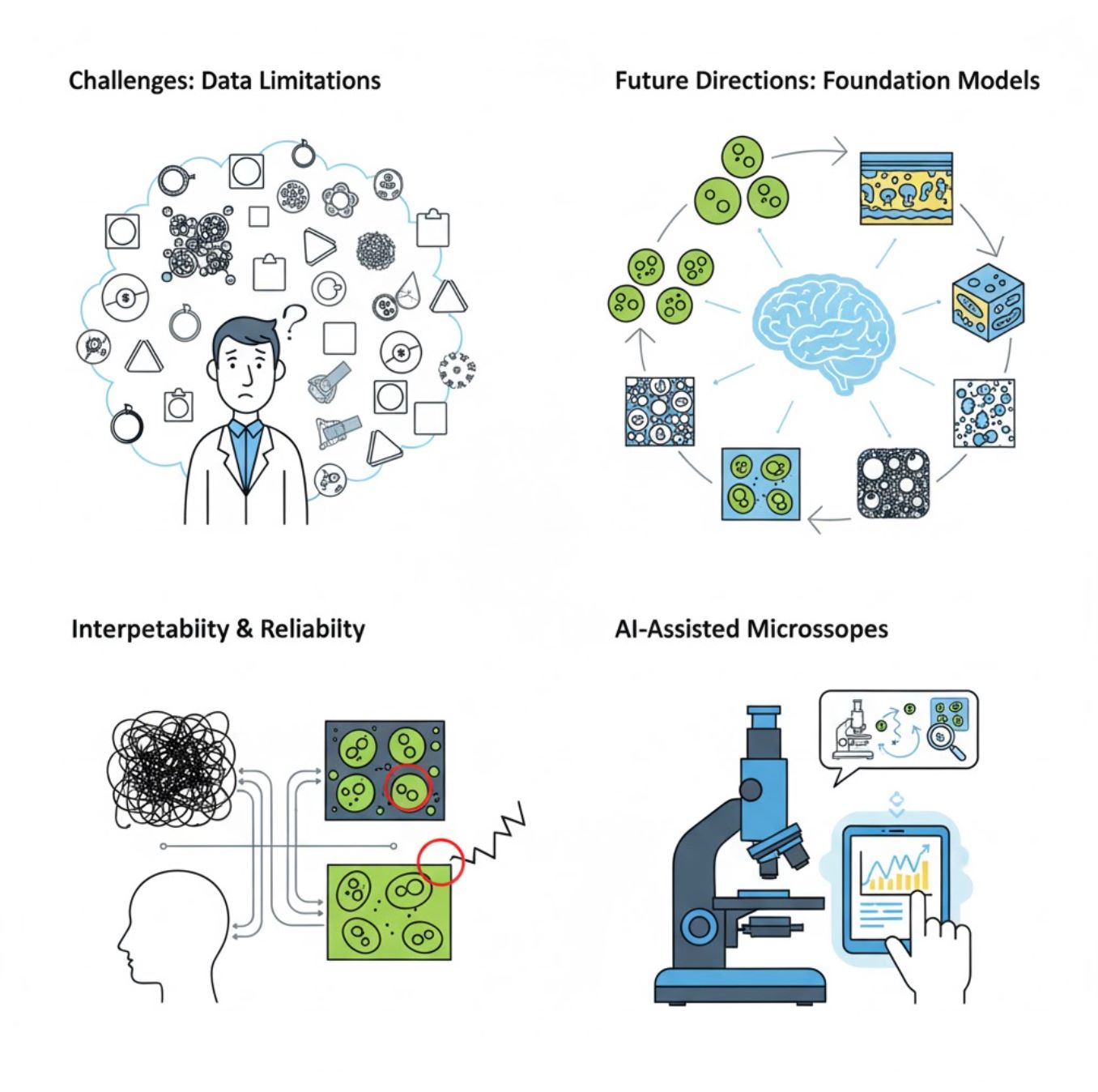
Проблемы и перспективы
Текущие проблемы
Новые тенденции
Модели фундаментального зрения
Системы ИИ следующего поколения обещают снизить потребность в обучении для каждой конкретной задачи.
- Модели, такие как SAM и системы на базе CLIP
- Один ИИ для множества задач микроскопии
- Быстрая развертка и адаптация
ИИ-поддерживаемые микроскопы
Полностью автономные и интеллектуальные системы микроскопии становятся реальностью.
- Управление на естественном языке через большие языковые модели (LLM)
- Полностью автоматизированные циклы обратной связи
- Демократизация доступа к передовой микроскопии
Основные выводы
- ИИ быстро трансформирует обработку изображений микроскопа, повышая точность и автоматизацию
- Глубокое обучение превосходит традиционное машинное обучение на сложных и вариабельных микроскопических изображениях
- CNN автоматически обучаются иерархическим признакам из сырых пикселей для надежного анализа
- Ключевые приложения включают сегментацию, классификацию, трекинг, снижение шума и автоматизированное получение изображений
- Успех зависит от качества данных и тщательной проверки экспертами
- Модели фундаментального зрения и ИИ-поддерживаемые микроскопы — будущее области
С дальнейшим развитием и усилиями сообщества (инструменты с открытым исходным кодом, общие наборы данных) ИИ все больше станет «глазом» микроскопа, помогая ученым видеть невидимое.







No comments yet. Be the first to comment!