L'IA dans le traitement des images au microscope
L'IA révolutionne le traitement des images au microscope grâce à des capacités puissantes telles que la segmentation précise, la réduction du bruit, la super-résolution et l'acquisition automatisée d'images. Cet article met en lumière les outils essentiels d'IA et les tendances émergentes en recherche scientifique.
Les techniques d'IA révolutionnent la microscopie en optimisant l'acquisition d'images et en automatisant l'analyse. Dans les microscopes intelligents modernes, les modules d'IA peuvent ajuster les paramètres d'imagerie en temps réel (par exemple, la mise au point, l'illumination) pour minimiser la photoblanchiment et améliorer le signal. Parallèlement, les algorithmes d'apprentissage profond peuvent analyser des données d'images complexes pour extraire des informations biologiques cachées et même relier les images à d'autres données (par exemple, la génomique).
Méthodes d'IA : apprentissage automatique vs apprentissage profond
Les méthodes d'IA vont de l'apprentissage automatique (ML) classique à l'apprentissage profond (DL) moderne. Chaque approche présente des forces et des limites distinctes :
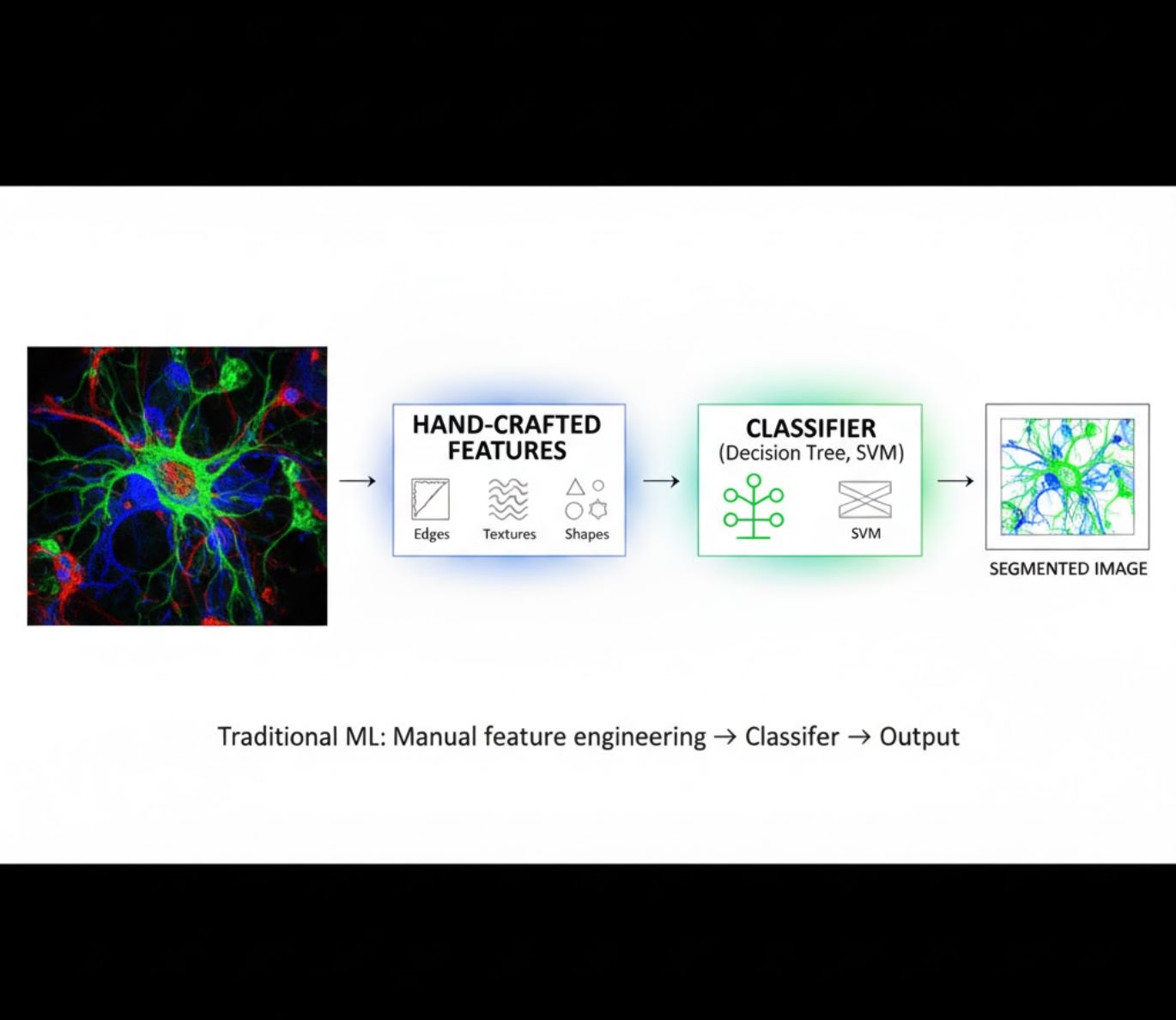
Caractéristiques conçues manuellement
- Les chercheurs conçoivent manuellement des caractéristiques d'image (contours, textures, formes)
- Les caractéristiques sont fournies à des classificateurs (arbres de décision, SVM)
- Rapide à entraîner
- Peine à gérer des images complexes ou bruitées
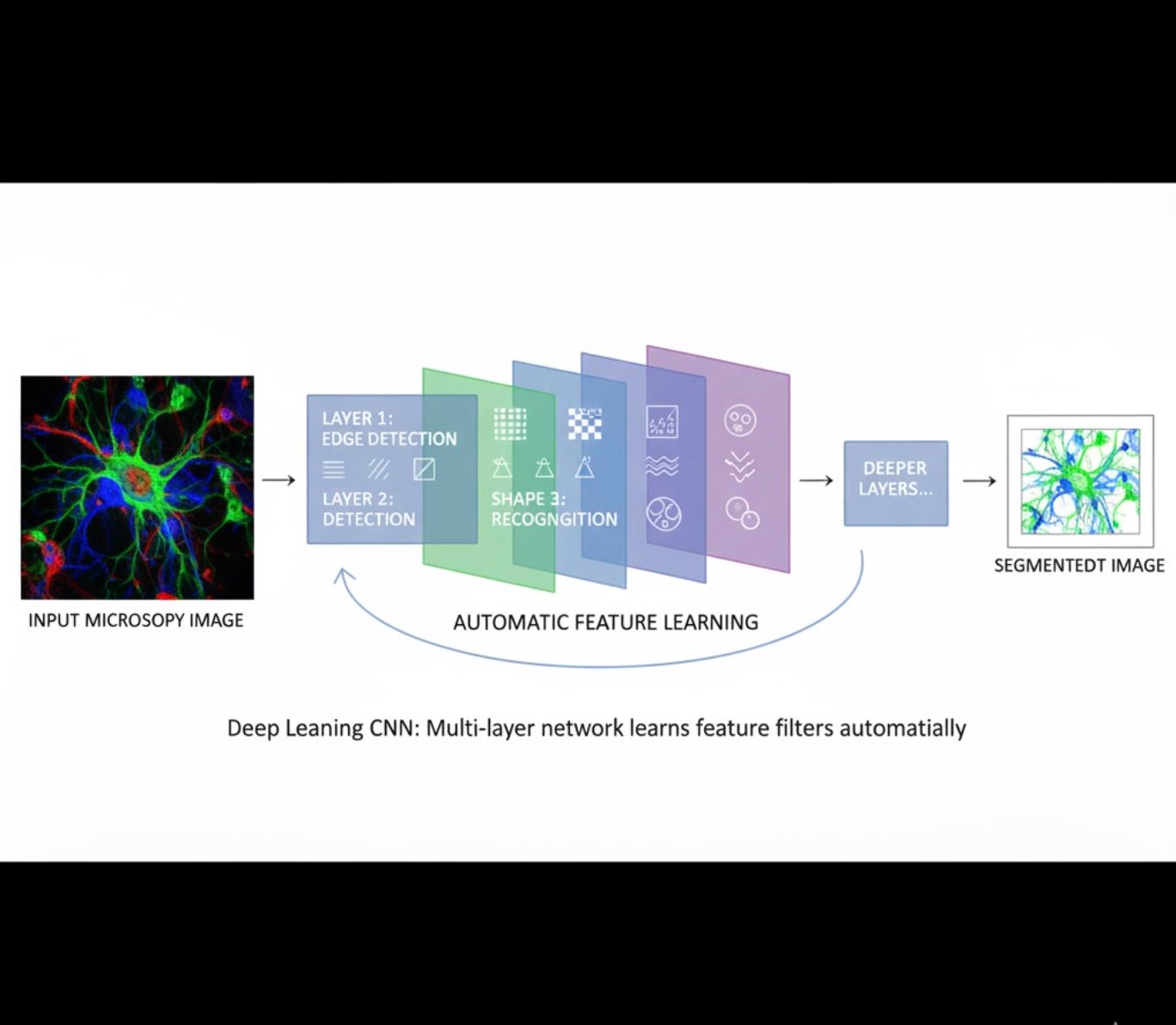
Apprentissage automatique des caractéristiques
- Les réseaux neuronaux multicouches (CNN) apprennent automatiquement les caractéristiques
- Apprentissage de bout en bout à partir des pixels bruts
- Beaucoup plus robuste aux variations
- Capture de manière fiable des textures et structures complexes
Fonctionnement des CNN : Les réseaux neuronaux convolutionnels appliquent des filtres successifs aux images de microscopie, apprenant à détecter des motifs simples (contours) dans les couches initiales et des structures complexes (formes cellulaires, textures) dans les couches profondes. Cet apprentissage hiérarchique rend le DL exceptionnellement robuste même lorsque les profils d'intensité varient fortement.
Comparaison visuelle : pipelines ML vs DL
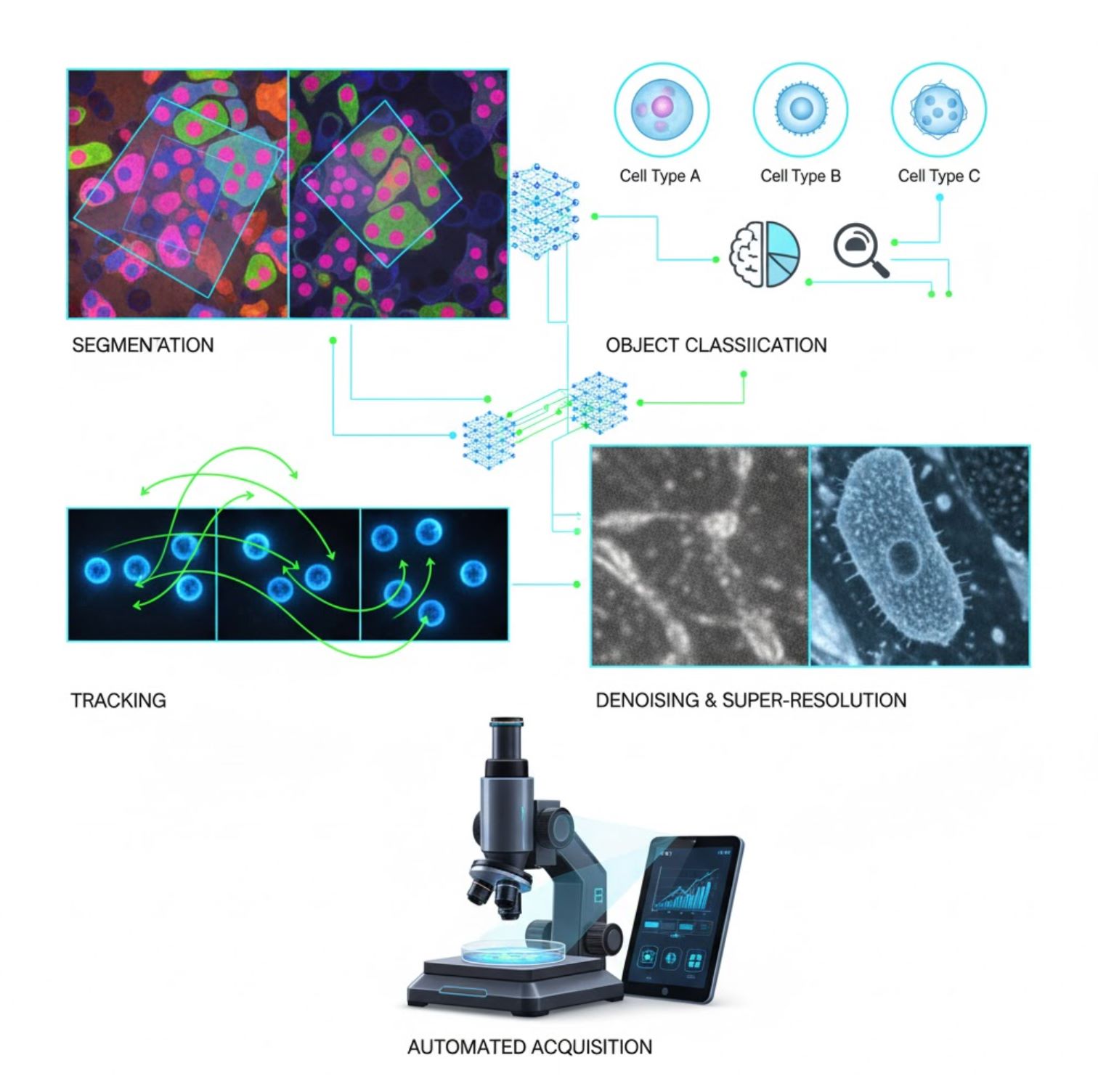
Applications clés de l'IA en microscopie
L'IA est désormais intégrée dans de nombreuses tâches de traitement d'images tout au long du flux de travail en microscopie :
Segmentation
Partition des images en régions (par exemple, identification de chaque cellule ou noyau). Les réseaux profonds comme U-Net excellent dans cette tâche.
- Segmentation sémantique : étiquettes de classe par pixel
- Segmentation d'instance : séparation des objets individuels
- Haute précision sur des images denses ou peu lumineuses
- Modèles fondamentaux de vision (ex. μSAM) désormais adaptés à la microscopie
Classification d'objets
Après segmentation, l'IA classe chaque objet avec une grande précision.
- Identification du type cellulaire
- Détermination du stade mitotique
- Détection d'indicateurs pathologiques
- Distingue des phénotypes subtils difficiles à quantifier manuellement
Suivi
En microscopie en temps réel, l'IA suit les cellules ou particules à travers les images avec une précision sans précédent.
- L'apprentissage profond améliore considérablement la précision du suivi
- Permet une analyse fiable des cellules en mouvement
- Capture les processus biologiques dynamiques
Dénombrement du bruit & super-résolution
Les modèles d'IA améliorent la qualité des images en supprimant le bruit et le flou.
- Modèles profonds informés par la physique apprennent l'optique du microscope
- Reconstruction d'images plus nettes et sans artefacts
- Résolution supérieure avec moins d'artefacts comparé aux méthodes traditionnelles
Acquisition automatisée
L'IA guide le microscope lui-même en temps réel.
- Analyse les images en direct pour prendre des décisions intelligentes
- Ajuste automatiquement la mise au point et scanne les zones d'intérêt
- Réduit la phototoxicité et fait gagner du temps
- Permet des expériences d'imagerie à haut débit et adaptatives
Outils d'IA populaires dans le traitement des images au microscope
Un écosystème riche en outils soutient l'IA en microscopie. Les chercheurs ont développé à la fois des logiciels polyvalents et spécialisés, dont beaucoup sont open source :
Cellpose
| Développeurs | Carsen Stringer et Marius Pachitariu (groupe de recherche MouseLand) |
| Plateformes supportées |
Nécessite Python (installation via pip/conda). Interface graphique disponible uniquement sur bureau. |
| Support linguistique | Documentation en anglais ; largement adopté dans les laboratoires de recherche du monde entier |
| Modèle tarifaire | Gratuit et open-source sous licence BSD-3-Clause |
Présentation
Cellpose est un outil avancé de segmentation basé sur l’apprentissage profond, conçu pour les images de microscopie. En tant qu’algorithme généraliste, il segmente avec précision divers types cellulaires (noyaux, cytoplasme, etc.) à travers différentes modalités d’imagerie sans nécessiter de réentraînement du modèle. Grâce à ses capacités d’intervention humaine, les chercheurs peuvent affiner les résultats, adapter le modèle à leurs données et appliquer le système aux flux d’imagerie 2D et 3D.
Fonctionnalités clés
Fonctionne immédiatement pour une grande variété de types cellulaires, colorations et modalités d’imagerie sans entraînement personnalisé.
Prend en charge les piles 3D complètes via une approche « 2,5D » qui réutilise les modèles 2D pour les données volumétriques.
Corrigez manuellement les résultats de segmentation et réentraînez le modèle sur vos données personnalisées pour une meilleure précision.
Accès via API Python, interface en ligne de commande ou interface graphique pour des flux de travail flexibles.
Fonctions de débruitage, de déconvolution et de suréchantillonnage pour améliorer la qualité des images avant segmentation.
Télécharger ou accéder
Contexte technique
Cellpose a été présenté dans une étude majeure par Stringer, Wang, Michaelos et Pachitariu, entraîné sur un jeu de données large et très varié contenant plus de 70 000 objets segmentés. Cette diversité permet au modèle de généraliser à travers les formes, tailles cellulaires et configurations de microscopie, réduisant significativement le besoin d’entraînement personnalisé dans la plupart des cas. Pour les données 3D, Cellpose réutilise astucieusement son modèle 2D selon une approche « 2,5D », évitant ainsi la nécessité de données d’entraînement annotées en 3D tout en fournissant une segmentation volumétrique. Cellpose 2.0 a introduit le réentraînement avec intervention humaine, permettant aux utilisateurs de corriger manuellement les prédictions et de réentraîner sur leurs propres images pour améliorer les performances sur des jeux de données spécifiques.
Installation & configuration
Configurez un environnement Python avec conda :
conda create -n cellpose python=3.10Activez l’environnement et installez Cellpose :
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposePremiers pas
Mode interface graphique
- Lancez l’interface graphique en exécutant :
python -m cellpose - Glissez-déposez des fichiers image (
.tif,.png, etc.) dans l’interface - Sélectionnez le type de modèle (par exemple, « cyto » pour le cytoplasme ou « nuclei » pour les noyaux)
- Définissez le diamètre cellulaire estimé ou laissez Cellpose s’auto-calibrer
- Cliquez pour démarrer la segmentation et visualiser les résultats
Mode API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Affiner & réentraîner
- Après génération des masques, corrigez la segmentation dans l’interface graphique en fusionnant ou supprimant manuellement des masques
- Utilisez les fonctions d’entraînement intégrées pour réentraîner sur les exemples corrigés
- Amélioration des performances du modèle sur votre jeu de données spécifique
Traiter des données 3D
- Chargez un TIFF multi-Z ou une pile volumétrique
- Utilisez le paramètre
--Zstackdans l’interface graphique ou l’API pour traiter en 3D - Affinez éventuellement les flux 3D via un lissage ou des paramètres spécialisés pour une meilleure segmentation
Limitations & considérations
- Compromis sur la généralité du modèle : Bien que le modèle généraliste fonctionne largement, des formes cellulaires très inhabituelles ou des conditions d’imagerie spécifiques peuvent nécessiter un réentraînement.
- Effort d’annotation : Le réentraînement avec intervention humaine demande des corrections manuelles, ce qui peut être long pour de grands jeux de données.
- Complexité d’installation : L’installation de l’interface graphique peut nécessiter l’usage de la ligne de commande, des environnements conda et la gestion des dépendances Python — ce qui n’est pas toujours simple pour les non-programmeurs.
- Usage bureau uniquement : Cellpose est conçu pour une utilisation sur ordinateur de bureau ; aucune application native Android ou iOS n’est disponible.
Questions fréquentes
Non — Cellpose fournit des modèles généralistes pré-entraînés qui fonctionnent souvent bien sans réentraînement. Cependant, pour des résultats optimaux sur des données spéciales ou inhabituelles, vous pouvez annoter et réentraîner en utilisant les fonctionnalités avec intervention humaine.
Oui — il prend en charge la 3D en réutilisant son modèle 2D (approche dite « 2,5D »), et vous pouvez traiter des piles volumétriques via l’interface graphique ou l’API.
Un GPU est fortement recommandé pour accélérer l’inférence et l’entraînement, surtout sur des jeux de données volumineux ou 3D, mais Cellpose peut fonctionner sur des machines CPU uniquement avec des performances réduites.
Dans l’interface graphique, définissez manuellement le diamètre cellulaire estimé ou laissez Cellpose le calibrer automatiquement. Vous pouvez affiner les résultats et réentraîner si la segmentation n’est pas optimale.
Oui — les versions récentes (Cellpose 3) incluent des modèles de restauration d’images pour le débruitage, la déconvolution et le suréchantillonnage afin d’améliorer la qualité de la segmentation avant traitement.
StarDist
| Développeurs | Uwe Schmidt, Martin Weigert, Coleman Broaddus et Gene Myers |
| Plateformes supportées |
|
| Support linguistique | Projet open source avec documentation et communauté principalement en anglais |
| Modèle tarifaire | Gratuit et open source. Sous licence BSD-3-Clause |
Présentation
StarDist est un outil d’apprentissage profond pour la segmentation d’instances dans les images de microscopie. Il représente chaque objet (comme les noyaux cellulaires) sous forme de polygone étoile-convexe en 2D ou de polyèdre en 3D, permettant une détection et une séparation précises d’objets densément regroupés ou chevauchants. Grâce à son architecture robuste, StarDist est largement utilisé pour la segmentation automatisée de cellules et noyaux en microscopie à fluorescence, histopathologie et autres applications d’analyse bio-imagerie.
Fonctionnalités clés
Segmentation d’instances très précise utilisant des polygones étoile-convexes (2D) et polyèdres (3D) pour une détection fiable des objets.
Modèles dédiés pour images 2D et données volumétriques 3D pour une analyse microscopique complète.
Modèles prêts à l’emploi pour noyaux fluorescents, histologie colorée H&E et autres scénarios d’imagerie courants.
Classification des objets détectés en classes distinctes (par ex. différents types cellulaires) en une seule exécution de segmentation.
Intégration fluide avec ImageJ/Fiji, QuPath et napari pour des flux de travail accessibles via interface graphique.
Évaluation complète de la segmentation d’instances incluant précision, rappel, score F1 et qualité panoptique.
Contexte technique
Initialement présenté dans un article MICCAI 2018, l’innovation principale de StarDist est la prédiction des distances radiales le long de rayons fixes combinée à la probabilité d’objet pour chaque pixel, permettant une reconstruction précise des formes étoile-convexes. Cette approche segmente de manière fiable des objets très proches difficiles à séparer avec les méthodes traditionnelles basées sur les pixels ou les boîtes englobantes.
Les développements récents ont étendu StarDist aux images d’histopathologie, permettant non seulement la segmentation des noyaux mais aussi la classification multi-classes des objets détectés. La méthode a obtenu des performances de pointe dans des challenges tels que CoNIC (Colon Nuclei Identification and Counting).
Télécharger ou accéder
Installation & configuration
Installez TensorFlow (version 1.x ou 2.x) comme prérequis pour StarDist.
Utilisez pip pour installer le package Python StarDist :
pip install stardistPour napari :
pip install stardist-napariPour QuPath : Installez l’extension StarDist en glissant le fichier .jar dans QuPath.
Pour ImageJ/Fiji : Utilisez le gestionnaire de plugins intégré ou l’installation manuelle via le menu plugins.
Exécution de la segmentation
Chargez un modèle pré-entraîné, normalisez votre image et lancez la prédiction :
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Ouvrez votre image dans napari, sélectionnez le plugin StarDist, choisissez un modèle pré-entraîné ou personnalisé, et lancez la prédiction directement depuis l’interface graphique.
Utilisez le plugin StarDist depuis le menu Plugins pour appliquer un modèle sur votre pile d’images avec une interface intuitive.
Après installation de l’extension, lancez la détection StarDist via la console de scripts ou l’interface graphique de QuPath pour l’analyse histopathologique.
Entraînement & ajustement fin
Créez des images d’étiquettes de vérité terrain où chaque objet est étiqueté de manière unique. Utilisez des outils d’annotation comme LabKit, QuPath ou Fiji pour préparer votre jeu de données.
Utilisez l’API Python de StarDist pour entraîner un nouveau modèle ou affiner un modèle existant avec vos données annotées personnalisées.
Options de post-traitement
- Appliquer la suppression des maxima non locaux (NMS) pour éliminer les formes candidates redondantes
- Utiliser StarDist OPP (post-traitement d’objets) pour fusionner les masques des formes non étoile-convexes
Limitations & considérations
- L’hypothèse étoile-convexe peut ne pas modéliser parfaitement les formes très non convexes ou très irrégulières
- Complexité d’installation : les installations personnalisées requièrent un compilateur C++ compatible pour construire les extensions
- L’accélération GPU dépend des versions compatibles de TensorFlow, CUDA et cuDNN
- Certains utilisateurs rapportent des problèmes lors de l’exécution du plugin ImageJ à cause de la configuration Java
Questions fréquentes
StarDist fonctionne avec une variété de types d’images incluant fluorescence, champ clair et histopathologie (par ex. H&E), grâce à ses modèles pré-entraînés flexibles et son adaptabilité aux différentes modalités d’imagerie.
Oui — StarDist supporte la segmentation d’instances 3D en utilisant des polyèdres étoile-convexes pour les données volumétriques, étendant les capacités 2D à une analyse complète en 3D.
Pas nécessairement. Des modèles pré-entraînés sont disponibles et fonctionnent souvent bien immédiatement. Cependant, pour des données spécialisées ou inédites, annoter et entraîner des modèles personnalisés améliore significativement la précision.
StarDist s’intègre avec napari, ImageJ/Fiji et QuPath, vous permettant d’exécuter la segmentation via une interface graphique sans coder. Il supporte également l’utilisation directe de l’API Python pour des flux de travail avancés.
StarDist fournit des fonctions intégrées pour calculer les métriques courantes de segmentation d’instances incluant précision, rappel, score F1 et qualité panoptique afin d’évaluer la performance de la segmentation.
SAM
Informations sur l’application
| Développeur | Meta AI Research (FAIR) |
| Appareils supportés |
|
| Langue & Disponibilité | Modèle fondamental open-source disponible mondialement ; documentation en anglais |
| Tarification | Gratuit — open-source sous licence Meta via GitHub et intégration MIB |
Vue d’ensemble générale
SAM (Segment Anything Model) est un puissant modèle fondamental d’IA créé par Meta qui permet la segmentation interactive et automatique de pratiquement n’importe quel objet dans les images. En utilisant des invites telles que des points, des cadres englobants ou des masques approximatifs, SAM génère des masques de segmentation sans nécessiter de réentraînement spécifique à la tâche. En recherche en microscopie, la flexibilité de SAM a été adaptée pour la segmentation cellulaire, la détection d’organites et l’analyse histopathologique, offrant une solution évolutive pour les chercheurs ayant besoin d’un outil de segmentation généraliste et promptable.
Introduction détaillée
Initialement entraîné par Meta sur plus de 1 milliard de masques répartis sur 11 millions d’images, SAM a été conçu comme un modèle fondamental promptable pour la segmentation avec des performances « zéro-shot » sur des domaines nouveaux. En recherche en imagerie médicale, SAM a été évalué pour la segmentation de lames entières en pathologie, la détection de tumeurs et l’identification des noyaux cellulaires. Cependant, ses performances sur des instances très denses — comme les noyaux cellulaires — sont mitigées : même avec de nombreuses invites (par exemple, 20 clics ou cadres), la segmentation zéro-shot peut être difficile dans des images complexes de microscopie.
Pour pallier cette limitation, des adaptations spécifiques au domaine ont émergé :
- SAMCell — Fine-tuné sur de larges jeux de données de microscopie pour une segmentation zéro-shot performante sur divers types cellulaires sans réentraînement par expérience
- μSAM — Réentraîné sur plus de 17 000 images de microscopie annotées manuellement pour améliorer la précision sur les petites structures cellulaires
Fonctionnalités clés
Interaction flexible utilisant points, cadres et masques pour un contrôle précis.
Effectue la segmentation sans fine-tuning sur de nouveaux domaines d’images.
Adaptable pour la microscopie et l’histopathologie via un réentraînement few-shot ou basé sur des invites.
Disponible dans Microscopy Image Browser (MIB) avec prise en charge de la segmentation 3D et interpolée.
IDCC-SAM permet le comptage automatique des cellules en immunocytochimie sans annotation manuelle.
Télécharger ou accéder
Guide utilisateur
- Ouvrez Microscopy Image Browser et accédez au panneau de segmentation SAM
- Configurez l’interpréteur Python et choisissez entre les modèles SAM-1 ou SAM-2
- Pour l’accélération GPU, sélectionnez « cuda » dans l’environnement d’exécution (recommandé pour des performances optimales)
- Invites par points : Cliquez sur un objet pour définir une graine positive ; utilisez Shift + clic pour étendre et Ctrl + clic pour des graines négatives
- Piles 3D : Utilisez le mode interactif 3D — cliquez sur une tranche, faites défiler avec Shift, et interpolez les graines entre les tranches
- Mode ajustement : Remplacez, ajoutez, soustrayez des masques ou créez un nouveau calque selon les besoins
- Utilisez l’option « Tout automatique » de MIB dans le panneau SAM-2 pour segmenter tous les objets visibles dans une région
- Vérifiez et affinez les masques après segmentation si nécessaire
- Utilisez les pipelines de fine-tuning basés sur des invites (par exemple, « All-in-SAM ») pour générer des annotations au niveau pixel à partir d’invites utilisateur éparses
- Pour le comptage cellulaire, appliquez IDCC-SAM, qui utilise SAM dans un pipeline zéro-shot avec post-traitement
- Pour une segmentation cellulaire très précise, utilisez SAMCell, fine-tuné sur des images cellulaires de microscopie
Limitations & considérations
- Performances zéro-shot inconstantes sur des structures denses ou chevauchantes sans ajustement au domaine
- Qualité de segmentation fortement dépendante de la conception et de la stratégie des invites
- GPU fortement recommandé ; l’inférence CPU est très lente
- Difficultés avec les images entières très haute résolution et les structures tissulaires multi-échelles
- Le fine-tuning ou l’adaptation de SAM pour la microscopie peut nécessiter des compétences en apprentissage automatique
Questions fréquentes
Oui — grâce à des adaptations comme SAMCell, qui fine-tune SAM sur des jeux de données de microscopie spécifiquement pour les tâches de segmentation cellulaire.
Pas toujours. Avec IDCC-SAM, vous pouvez effectuer un comptage cellulaire zéro-shot sans annotations manuelles.
Utilisez le fine-tuning basé sur des invites (par exemple, « All-in-SAM ») ou des versions préentraînées pour la microscopie comme μSAM, entraîné sur plus de 17 000 images de microscopie annotées.
Bien que possible sur CPU, le GPU est fortement recommandé pour une vitesse d’inférence pratique et une segmentation interactive en temps réel.
Oui — l’intégration SAM-2 de MIB prend en charge la segmentation 3D avec interpolation des graines entre les tranches pour l’analyse volumétrique.
AxonDeepSeg
| Développeur | NeuroPoly Lab à Polytechnique Montréal et Université de Montréal |
| Plateformes supportées |
|
| Langue | Documentation en anglais ; outil open source utilisé mondialement |
| Tarification | Gratuit et open source |
Présentation
AxonDeepSeg est un outil propulsé par l’IA pour la segmentation automatique des axones et de la myéline dans les images de microscopie. Utilisant des réseaux de neurones convolutionnels, il fournit une segmentation précise à trois classes (axone, myéline, fond) sur plusieurs modalités d’imagerie, notamment TEM, SEM et microscopie en champ clair. En automatisant les mesures morphométriques telles que le diamètre des axones, le rapport g et l’épaisseur de la myéline, AxonDeepSeg facilite l’analyse quantitative en recherche neuroscientifique, réduisant significativement le temps d’annotation manuelle et améliorant la reproductibilité.
Fonctionnalités clés
Modèles prêts à l’emploi optimisés pour les modalités de microscopie TEM, SEM et en champ clair.
Classification précise des régions axone, myéline et fond dans les images de microscopie.
Calcul automatique du diamètre d’axone, du rapport g, de l’épaisseur de myéline et des métriques de densité.
L’intégration avec l’interface Napari permet un affinage manuel des masques de segmentation pour une meilleure précision.
S’intègre parfaitement dans des pipelines personnalisés pour l’analyse à grande échelle des tissus neuronaux.
Des scripts de test complets garantissent la reproductibilité et la fiabilité des résultats de segmentation.
Détails techniques
Développé par le NeuroPoly Lab, AxonDeepSeg exploite le deep learning pour offrir une segmentation de haute précision destinée aux applications neuroscientifiques. Des modèles pré-entraînés sont disponibles pour différentes modalités de microscopie, assurant une grande polyvalence selon les techniques d’imagerie. L’outil s’intègre avec Napari, permettant des corrections interactives des masques de segmentation, ce qui améliore la précision sur des jeux de données complexes. AxonDeepSeg calcule des métriques morphométriques clés, soutenant les études à haut débit sur la structure et la pathologie des tissus neuronaux. Son framework basé sur Python facilite l’intégration dans des pipelines personnalisés pour l’analyse à grande échelle de la morphologie des axones et de la myéline.
Téléchargement ou accès
Installation & configuration
Assurez-vous que Python 3.8 ou une version ultérieure est installé, puis installez AxonDeepSeg et Napari via pip :
pip install axondeepseg napariExécutez les scripts de test fournis pour confirmer que tous les composants sont correctement installés et fonctionnels.
Importez vos images de microscopie (TEM, SEM ou champ clair) dans Napari ou votre environnement Python.
Choisissez le modèle pré-entraîné adapté à votre modalité d’imagerie et lancez la segmentation pour générer les masques d’axone et de myéline.
Calculez automatiquement les mesures morphométriques telles que le diamètre d’axone, le rapport g, la densité et l’épaisseur de myéline, puis exportez les résultats au format CSV.
Utilisez l’interface Napari pour ajuster manuellement les masques de segmentation si nécessaire, en fusionnant ou supprimant des masques pour améliorer la précision.
Considérations importantes
- Les performances peuvent diminuer sur des modalités d’imagerie nouvelles ou non entraînées
- Des corrections manuelles peuvent être nécessaires pour des régions complexes ou difficiles
- Le GPU est recommandé pour un traitement plus rapide des grands ensembles de données ; le CPU est également supporté
Questions fréquentes
AxonDeepSeg supporte la microscopie TEM (microscopie électronique en transmission), SEM (microscopie électronique à balayage) et la microscopie en champ clair avec des modèles pré-entraînés optimisés pour chaque modalité.
Oui, AxonDeepSeg est entièrement gratuit et open source, disponible pour un usage académique et commercial.
Oui, AxonDeepSeg calcule automatiquement le diamètre des axones, le rapport g, l’épaisseur de la myéline et les métriques de densité à partir des images segmentées.
Le GPU est recommandé pour une segmentation plus rapide des grands ensembles de données, mais le traitement sur CPU est également possible pour des analyses plus petites.
Oui, l’intégration avec l’interface Napari permet des corrections interactives et un affinage des masques de segmentation pour une meilleure précision sur des régions complexes.
Ilastik
| Développeur | Équipe Ilastik au Laboratoire européen de biologie moléculaire (EMBL) et partenaires académiques associés |
| Plateformes supportées |
|
| Langue | Anglais |
| Tarification | Gratuit et open source |
Présentation
Ilastik est un outil puissant, piloté par l’IA, pour la segmentation interactive, la classification et l’analyse d’images de microscopie. Utilisant des techniques d’apprentissage automatique telles que les classificateurs Random Forest, il permet aux chercheurs de segmenter les pixels, classifier les objets, suivre les cellules dans le temps et effectuer le comptage de densité sur des ensembles de données 2D et 3D. Avec son interface intuitive et son retour en temps réel, Ilastik est accessible aux scientifiques sans compétences en programmation et est largement adopté en biologie cellulaire, neurosciences et imagerie biomédicale.
Fonctionnalités clés
Retour en temps réel lors de l’annotation de régions représentatives pour des résultats de segmentation instantanés.
Catégorisez les structures segmentées selon leurs caractéristiques morphologiques et d’intensité.
Suivez le mouvement et la division cellulaire dans des expériences de microscopie time-lapse 2D et 3D.
Quantifiez les régions denses sans segmentation explicite des objets individuels.
Segmentation semi-automatique pour des volumes 3D complexes avec interaction intuitive.
Traitez automatiquement plusieurs images en mode ligne de commande sans interface graphique.
Téléchargement
Guide de démarrage
Téléchargez Ilastik pour votre système d’exploitation depuis le site officiel. Le package inclut toutes les dépendances Python nécessaires, suivez donc les instructions d’installation spécifiques à votre plateforme.
Ouvrez Ilastik et choisissez votre flux d’analyse : Classification de pixels, Classification d’objets, Suivi ou Comptage de densité. Chargez votre jeu d’images, qui peut inclure des images multi-canaux, 3D ou en time-lapse.
Étiquetez quelques pixels ou objets représentatifs dans vos images. Le classificateur Random Forest d’Ilastik apprend à partir de ces annotations et prédit automatiquement les étiquettes sur l’ensemble de votre jeu de données.
Appliquez le modèle entraîné pour segmenter ou classifier l’ensemble de vos données. Exportez les résultats sous forme d’images étiquetées, de cartes de probabilité ou de tableaux quantitatifs pour analyse et visualisation ultérieures.
Utilisez le mode sans interface graphique d’Ilastik pour traiter automatiquement plusieurs images sans intervention manuelle, idéal pour les pipelines d’analyse à grande échelle.
Limitations et considérations
- L’étiquetage interactif peut être chronophage pour des ensembles de données très volumineux
- La précision dépend de la qualité et de la représentativité des annotations utilisateur
- Exigences mémoire — les ensembles de données très haute résolution ou de plusieurs gigaoctets peuvent nécessiter une RAM importante
- Données complexes — les classificateurs Random Forest peuvent être moins performants que les réseaux neuronaux profonds sur des données d’imagerie très variables ou complexes
Questions fréquentes
Oui, Ilastik prend en charge les volumes 3D et les expériences en time-lapse pour la segmentation, le suivi et l’analyse quantitative sur plusieurs points temporels.
Oui, Ilastik est entièrement gratuit et open source, disponible pour tous les utilisateurs sans restrictions de licence.
Non, Ilastik offre une interface graphique intuitive avec un retour en temps réel, le rendant accessible aux chercheurs sans compétences en programmation. Les utilisateurs avancés peuvent également utiliser le traitement par lots en ligne de commande.
Oui, le flux de travail dédié au suivi permet d’analyser le mouvement et la division cellulaire dans des ensembles de données time-lapse 2D et 3D avec suivi automatique des lignées.
Les résultats de segmentation peuvent être exportés sous forme d’images étiquetées, de cartes de probabilité ou de tableaux quantitatifs, permettant une intégration fluide avec les outils d’analyse et de visualisation en aval.
Ces outils couvrent des niveaux du novice à l'expert. Beaucoup sont gratuits et open-source, facilitant des flux de travail d'IA reproductibles et partageables au sein de la communauté scientifique.
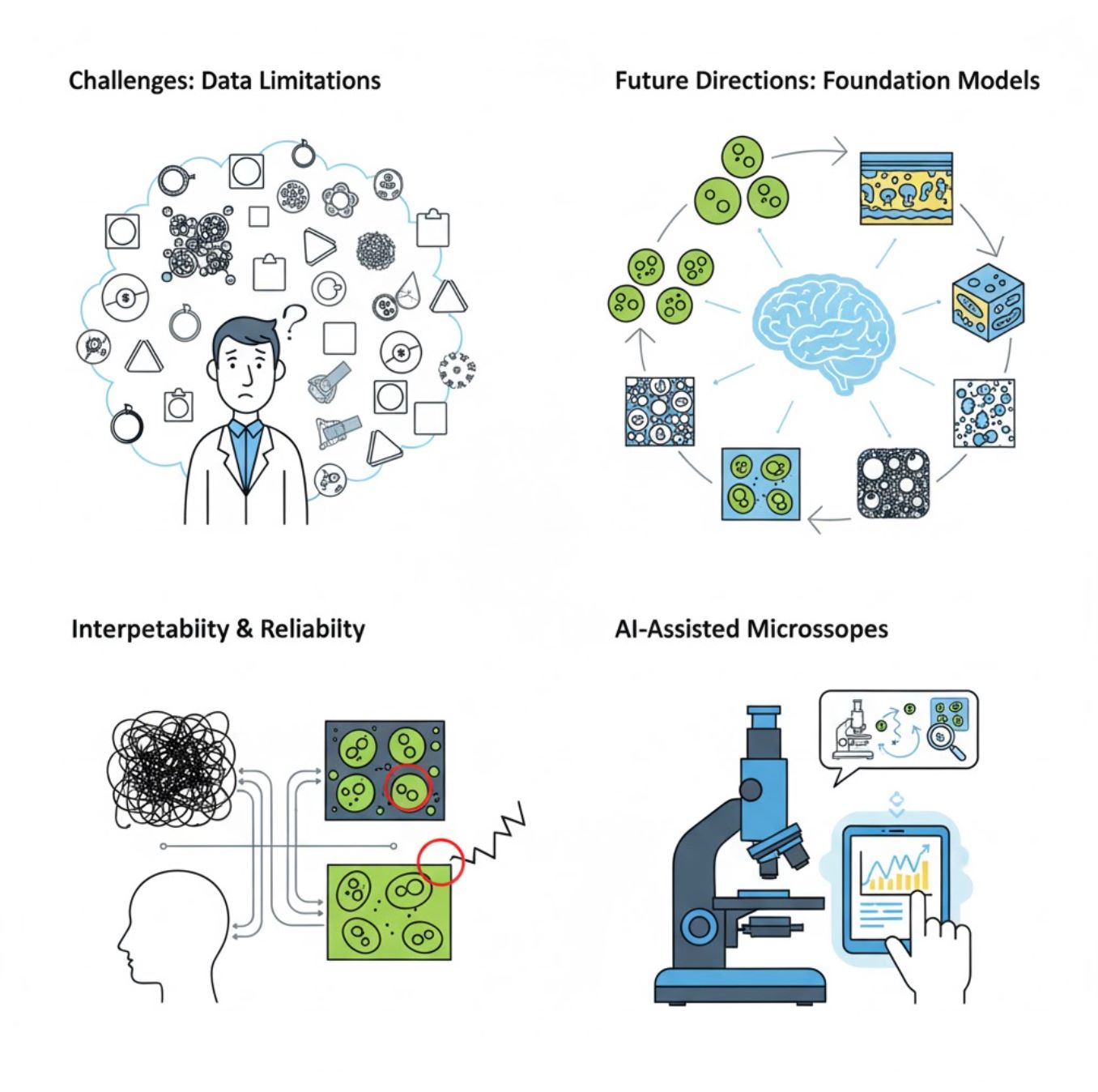
Défis et perspectives futures
Défis actuels
Tendances émergentes
Modèles fondamentaux de vision
Les systèmes d'IA de nouvelle génération promettent de réduire le besoin d'entraînement spécifique à chaque tâche.
- Modèles comme SAM et systèmes basés sur CLIP
- Une IA pour gérer de nombreuses tâches en microscopie
- Déploiement et adaptation plus rapides
Microscopes assistés par IA
Les systèmes de microscopie entièrement autonomes et intelligents deviennent réalité.
- Contrôle en langage naturel via des LLM
- Boucles de rétroaction entièrement automatisées
- Démocratise l'accès à la microscopie avancée
Points clés à retenir
- L'IA transforme rapidement le traitement des images au microscope avec une meilleure précision et automatisation
- L'apprentissage profond surpasse l'apprentissage automatique traditionnel sur des images complexes et variables
- Les CNN apprennent automatiquement des caractéristiques hiérarchiques à partir des pixels bruts pour une analyse robuste
- Les applications clés incluent la segmentation, la classification, le suivi, la réduction du bruit et l'acquisition automatisée
- Le succès dépend de la qualité des données et d'une validation rigoureuse par des experts
- Les modèles fondamentaux de vision et les microscopes assistés par IA représentent l'avenir du domaine
Avec les progrès continus et les efforts communautaires (outils open-source, jeux de données partagés), l'IA deviendra de plus en plus une partie intégrante de « l'œil » du microscope, aidant les scientifiques à voir l'invisible.







No comments yet. Be the first to comment!