Mikroskop Görüntü İşlemede Yapay Zeka
Yapay zeka, hassas segmentasyon, gürültü azaltma, süper çözünürlük ve otomatik görüntü edinimi gibi güçlü yeteneklerle mikroskop görüntü işlemede devrim yaratıyor. Bu makale, bilimsel araştırmalarda temel yapay zeka araçları ve ortaya çıkan trendleri vurgulamaktadır.
Yapay zeka teknikleri, görüntü edinimini optimize ederek ve analizi otomatikleştirerek mikroskopiyi dönüştürüyor. Modern akıllı mikroskoplarda, yapay zeka modülleri odaklama, aydınlatma gibi görüntüleme parametrelerini anlık olarak ayarlayarak fotobleklenmeyi en aza indirir ve sinyali artırır. Bu arada, derin öğrenme algoritmaları karmaşık görüntü verilerini tarayarak gizli biyolojik bilgileri çıkarabilir ve görüntüleri genomik gibi diğer verilerle ilişkilendirebilir.
Yapay Zeka Yöntemleri: Makine Öğrenimi ve Derin Öğrenme
Yapay zeka yöntemleri klasik makine öğrenimi (ML) ile modern derin öğrenme (DL) arasında değişir. Her yaklaşımın kendine özgü güçlü ve zayıf yönleri vardır:
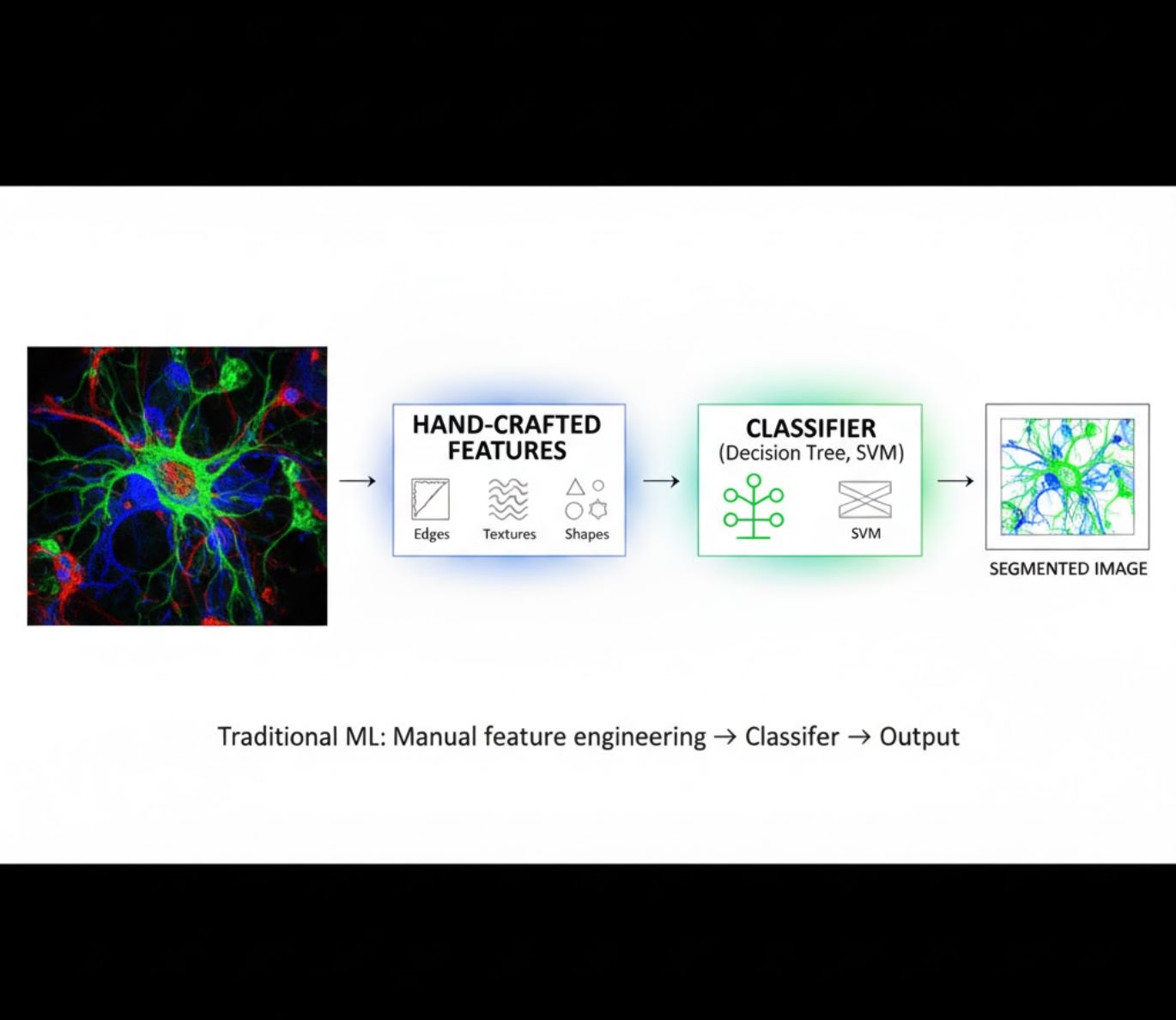
Elle Tasarlanmış Özellikler
- Araştırmacılar görüntü özelliklerini (kenarlar, dokular, şekiller) manuel olarak oluşturur
- Özellikler sınıflandırıcılara (karar ağaçları, SVM) beslenir
- Eğitimi hızlıdır
- Karmaşık veya gürültülü görüntülerde zorlanır
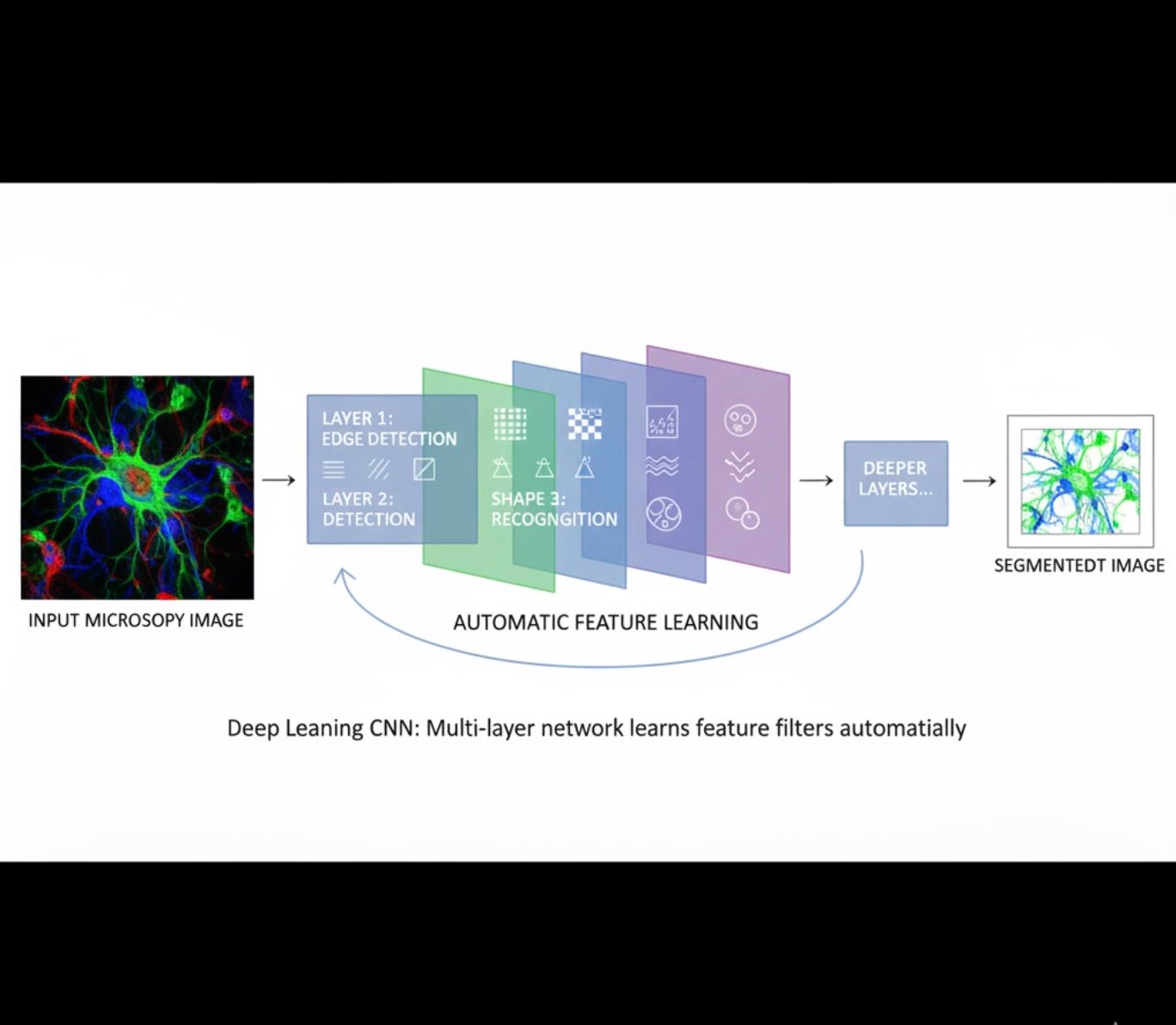
Otomatik Özellik Öğrenimi
- Çok katmanlı sinir ağları (CNN’ler) özellikleri otomatik öğrenir
- Ham piksellerden uçtan uca öğrenme
- Çeşitliliklere karşı çok daha dayanıklıdır
- İnce dokuları ve yapıları güvenilir şekilde yakalar
CNN’ler nasıl çalışır: Konvolüsyonel sinir ağları mikroskop görüntülerine ardışık filtreler uygular, erken katmanlarda basit desenleri (kenarlar) ve derin katmanlarda karmaşık yapıları (hücre şekilleri, dokular) öğrenir. Bu hiyerarşik öğrenme, yoğunluk profilleri önemli ölçüde değişse bile DL’yi son derece dayanıklı kılar.
Görsel Karşılaştırma: ML ve DL İş Akışları
Mikroskopide Önemli Yapay Zeka Uygulamaları
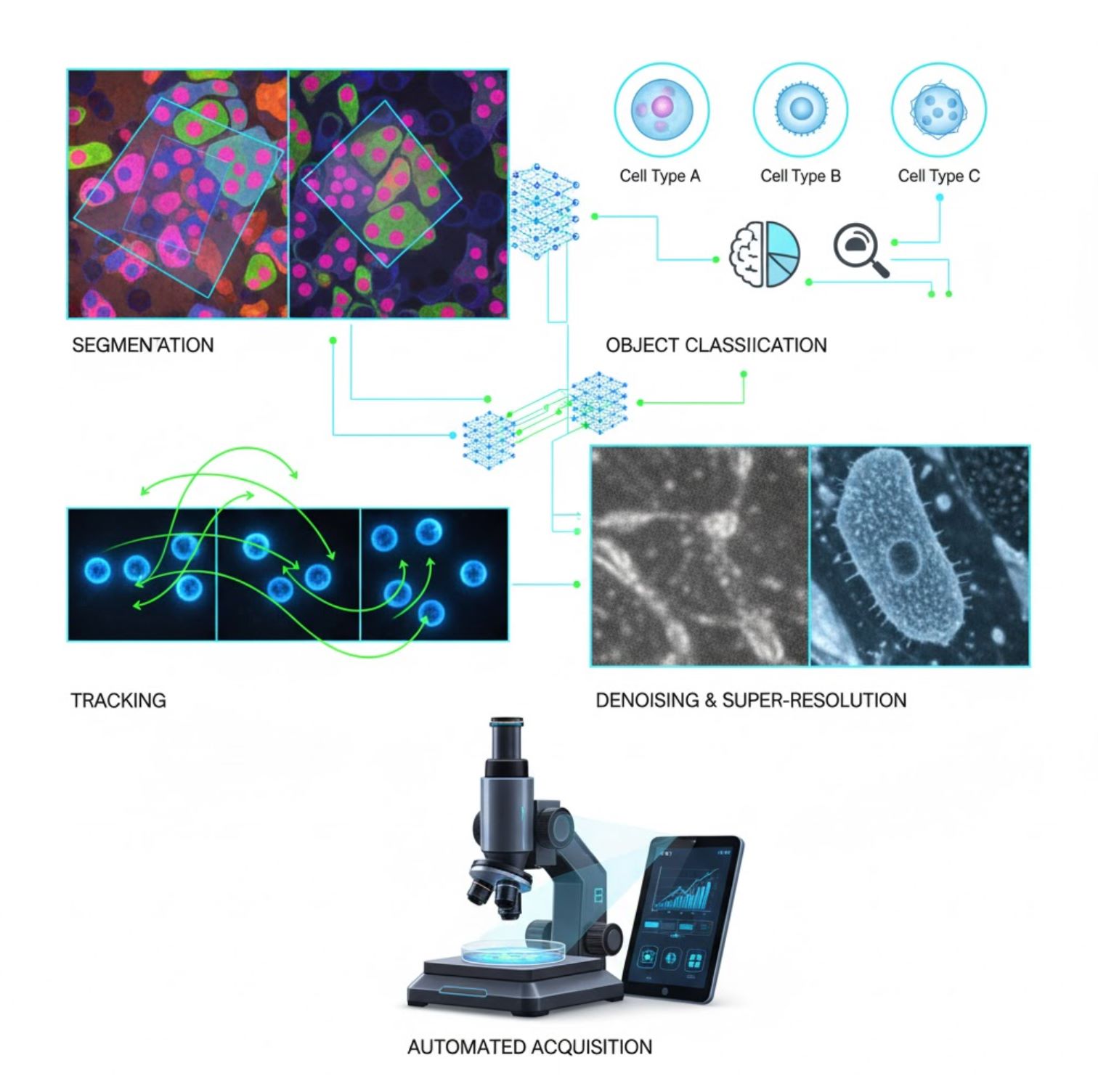
Yapay zeka artık mikroskop iş akışındaki birçok görüntü işleme görevine entegre edilmiştir:
Segmentasyon
Görüntüleri bölgelere ayırma (örneğin, her hücre veya çekirdeği tanımlama). U-Net gibi derin ağlar bu görevde üstündür.
- Semantik segmentasyon: Piksel başına sınıf etiketleri
- Örnek segmentasyonu: Bireysel nesnelerin ayrılması
- Kalabalık veya loş görüntülerde yüksek doğruluk
- Görsel temel modeller (örneğin, μSAM) artık mikroskopiye uyarlanmıştır
Nesne Sınıflandırması
Segmentasyondan sonra, yapay zeka her nesneyi yüksek hassasiyetle sınıflandırır.
- Hücre tipi tanımlama
- Mitotik evre belirleme
- Patoloji göstergesi tespiti
- Elle ölçülmesi zor ince fenotipleri ayırt eder
Takip
Zaman atlamalı mikroskopide, yapay zeka hücreleri veya partikülleri kareler arasında benzersiz doğrulukla takip eder.
- Derin öğrenme takip doğruluğunu önemli ölçüde artırır
- Hareket eden hücrelerin güvenilir analizi mümkün olur
- Dinamik biyolojik süreçleri yakalar
Gürültü Azaltma ve Süper Çözünürlük
Yapay zeka modelleri, gürültü ve bulanıklığı gidererek görüntü kalitesini artırır.
- Fizik bilgisi içeren derin modeller mikroskop optiklerini öğrenir
- Daha keskin, artefaktsız görüntüler yeniden oluşturulur
- Geleneksel yöntemlere kıyasla daha yüksek çözünürlük ve azalmış artefaktlar
Otomatik Edinim
Yapay zeka mikroskobu gerçek zamanlı olarak yönlendirir.
- Canlı görüntüleri analiz ederek akıllı kararlar verir
- Otomatik olarak odak ayarı yapar ve ilgi alanlarını tarar
- Fototoksisiteyi azaltır ve zaman kazandırır
- Yüksek verimli ve uyarlanabilir görüntüleme deneylerini mümkün kılar
Mikroskop Görüntü İşlemede Popüler Yapay Zeka Araçları
Mikroskopide yapay zekayı destekleyen zengin bir araç ekosistemi bulunmaktadır. Araştırmacılar hem genel amaçlı hem de özel amaçlı yazılımlar geliştirmiştir; bunların birçoğu açık kaynaklıdır:
Cellpose
| Geliştirici | Carsen Stringer ve Marius Pachitariu (MouseLand araştırma grubu) |
| Desteklenen Platformlar |
Python (pip/conda kurulumu) gerektirir. GUI sadece masaüstünde mevcuttur. |
| Dil Desteği | İngilizce dokümantasyon; dünya çapında araştırma laboratuvarlarında yaygın kullanım |
| Fiyatlandırma Modeli | BSD-3-Clause lisansı altında ücretsiz ve açık kaynak |
Genel Bakış
Cellpose, mikroskopi görüntüleri için tasarlanmış gelişmiş, derin öğrenme tabanlı bir segmentasyon aracıdır. Genel amaçlı bir algoritma olarak, model yeniden eğitimi gerektirmeden farklı görüntüleme modlarında çeşitli hücre tiplerini (çekirdekler, sitoplazma vb.) doğru şekilde segmentler. İnsan müdahalesi özellikleri sayesinde araştırmacılar sonuçları iyileştirebilir, modeli kendi verilerine uyarlayabilir ve sistemi hem 2D hem de 3D görüntü iş akışlarında kullanabilir.
Temel Özellikler
Özel eğitim gerektirmeden çok çeşitli hücre tipleri, boyalar ve görüntüleme modlarında kutudan çıkar çıkmaz çalışır.
Volümetrik veriler için 2D modelleri yeniden kullanan "2.5D" yaklaşımıyla tam 3D yığınları destekler.
Segmentasyon sonuçlarını manuel olarak düzeltin ve özel verilerinizde modeli yeniden eğiterek doğruluğu artırın.
Esnek iş akışları için Python API, komut satırı arayüzü veya grafiksel kullanıcı arayüzü üzerinden erişim.
Segmentasyon öncesinde görüntü kalitesini artırmak için gürültü giderme, bulanıklık giderme ve yükseltme yetenekleri.
İndir veya Erişim
Teknik Arka Plan
Cellpose, Stringer, Wang, Michaelos ve Pachitariu tarafından sunulan önemli bir çalışmada tanıtıldı ve 70.000’den fazla segmentlenmiş nesne içeren büyük ve çok çeşitli bir veri seti üzerinde eğitildi. Bu çeşitlilik, modelin hücre şekilleri, boyutları ve mikroskopi ayarları arasında genelleme yapmasını sağlar ve çoğu kullanım durumunda özel eğitim ihtiyacını önemli ölçüde azaltır. 3D veriler için Cellpose, tamamen 3D etiketli eğitim verisi gerektirmeden 2D modelini "2.5D" biçiminde yeniden kullanarak volümetrik segmentasyon sunar. Cellpose 2.0, kullanıcıların tahminleri manuel olarak düzeltebilmesi ve kendi görüntüleri üzerinde yeniden eğitim yapabilmesi için insan müdahaleli yeniden eğitim özelliğini getirdi.
Kurulum ve Ayarlar
Conda kullanarak bir Python ortamı kurun:
conda create -n cellpose python=3.10Ortamı etkinleştirin ve Cellpose’u kurun:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeBaşlarken
GUI Modu
- Şu komutu çalıştırarak GUI’yi başlatın:
python -m cellpose - Görüntü dosyalarını (
.tif,.pngvb.) arayüze sürükleyip bırakın - Model türünü seçin (örneğin, sitoplazma için "cyto" veya çekirdekler için "nuclei")
- Tahmini hücre çapını ayarlayın veya Cellpose’un otomatik kalibrasyon yapmasına izin verin
- Segmentasyonu başlatmak ve sonuçları görüntülemek için tıklayın
Python API Modu
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)İyileştir ve Yeniden Eğit
- Maskeler oluşturulduktan sonra, GUI’de segmentasyonu manuel olarak birleştirerek veya silerek düzeltin
- Dahili eğitim fonksiyonlarını kullanarak düzeltilmiş örnekler üzerinde yeniden eğitim yapın
- Özel veri setinizde model performansını artırın
3D Verileri İşleyin
- Çoklu Z TIFF veya volümetrik yığın yükleyin
- GUI veya API’de
--Zstackbayrağını kullanarak 3D olarak işleyin - Daha iyi segmentasyon için isteğe bağlı olarak 3D akışları yumuşatma veya özel parametrelerle iyileştirin
Sınırlamalar ve Dikkat Edilmesi Gerekenler
- Model Genelliği Takası: Genel amaçlı model geniş kapsamda çalışsa da, çok sıra dışı hücre şekilleri veya görüntüleme koşulları yeniden eğitim gerektirebilir.
- Anotasyon Çabası: İnsan müdahaleli eğitim manuel düzeltmeler gerektirir, bu da büyük veri setlerinde zaman alıcı olabilir.
- Kurulum Karmaşıklığı: GUI kurulumu komut satırı kullanımı, conda ortamları ve Python bağımlılıklarının yönetimini gerektirebilir — programlama bilgisi olmayanlar için her zaman kolay değildir.
- Sadece Masaüstü: Cellpose masaüstü kullanımı için tasarlanmıştır; yerel Android veya iOS uygulaması mevcut değildir.
Sıkça Sorulan Sorular
Hayır — Cellpose, çoğu durumda yeniden eğitim gerektirmeden iyi çalışan önceden eğitilmiş genel amaçlı modeller sunar. Ancak, özel veya sıra dışı verilerde en iyi sonuçlar için insan müdahaleli özellikleri kullanarak anotasyon yapabilir ve yeniden eğitim gerçekleştirebilirsiniz.
Evet — 2D modelini yeniden kullanarak ("2.5D" olarak adlandırılır) 3D desteği sağlar ve GUI veya API üzerinden volümetrik yığınları işleyebilirsiniz.
GPU, özellikle büyük veya 3D veri setlerinde daha hızlı çıkarım ve eğitim için şiddetle önerilir, ancak Cellpose sadece CPU ile de çalışabilir; performans daha düşük olur.
GUI’de tahmini hücre çapını manuel olarak ayarlayabilir veya Cellpose’un otomatik kalibrasyon yapmasına izin verebilirsiniz. Segmentasyon optimal değilse sonuçları iyileştirebilir ve yeniden eğitim yapabilirsiniz.
Evet — yeni sürümler (Cellpose 3) segmentasyon kalitesini artırmak için gürültü giderme, bulanıklık giderme ve yükseltme modelleri içerir.
StarDist
| Geliştirici | Uwe Schmidt, Martin Weigert, Coleman Broaddus ve Gene Myers |
| Desteklenen Platformlar |
|
| Dil Desteği | Ağırlıklı olarak İngilizce dokümantasyon ve topluluğa sahip açık kaynak proje |
| Fiyatlandırma Modeli | Ücretsiz ve açık kaynak. BSD-3-Clause lisansı altında |
Genel Bakış
StarDist, mikroskopi görüntülerinde örnek segmentasyonu için derin öğrenme aracıdır. Her nesneyi (örneğin hücre çekirdekleri) 2B’de yıldız-konveks çokgen, 3B’de polihedron olarak temsil ederek sıkışık veya üst üste binen nesnelerin doğru tespiti ve ayrımını sağlar. Sağlam mimarisiyle StarDist, floresan mikroskopi, histopatoloji ve diğer biyogörüntü analiz uygulamalarında otomatik hücre ve çekirdek segmentasyonu için yaygın olarak kullanılır.
Temel Özellikler
Güvenilir nesne tespiti için yıldız-konveks çokgenler (2B) ve polihedronlar (3B) kullanarak yüksek doğrulukta örnek segmentasyonu.
Kapsamlı mikroskopi analizi için hem 2B görüntüler hem de 3B hacim verileri için özel modeller.
Floresan çekirdekler, H&E boyalı histoloji ve diğer yaygın görüntüleme senaryoları için kullanıma hazır modeller.
Tek segmentasyon çalıştırmasında tespit edilen nesneleri farklı sınıflara (örneğin farklı hücre tipleri) ayırma.
Kullanıcı dostu GUI tabanlı iş akışları için ImageJ/Fiji, QuPath ve napari ile sorunsuz entegrasyon.
Hassasiyet, geri çağırma, F1 skoru ve panoptik kalite dahil kapsamlı örnek segmentasyon değerlendirmesi.
Teknik Arka Plan
StarDist, MICCAI 2018 makalesinde tanıtılmış olup, temel yeniliği sabit ışınlar boyunca radyal mesafelerin nesne olasılığı ile birlikte tahmin edilmesi ve böylece yıldız-konveks şekillerin doğru şekilde yeniden oluşturulmasıdır. Bu yöntem, geleneksel piksel tabanlı veya sınırlayıcı kutu yöntemleriyle ayrılması zor olan birbirine temas eden nesneleri güvenilir şekilde segmentler.
Son gelişmeler StarDist’ı histopatoloji görüntülerine genişletmiş, sadece çekirdek segmentasyonu değil aynı zamanda tespit edilen nesnelerin çoklu sınıf sınıflandırmasını da mümkün kılmıştır. Yöntem, CoNIC (Kolon Çekirdek Tanımlama ve Sayma) gibi yarışmalarda üst düzey performans göstermiştir.
İndir veya Erişim
Kurulum ve Ayarlar
StarDist için ön koşul olarak TensorFlow (sürüm 1.x veya 2.x) yükleyin.
StarDist Python paketini pip ile yükleyin:
pip install stardistnapari için:
pip install stardist-napariQuPath için: StarDist uzantısını .jar dosyasını QuPath’a sürükleyerek yükleyin.
ImageJ/Fiji için: Dahili eklenti yöneticisini veya eklentiler menüsünden manuel kurulumu kullanın.
Segmentasyonu Çalıştırma
Önceden eğitilmiş modeli yükleyin, görüntünüzü normalize edin ve tahmin çalıştırın:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Görüntünüzü napari’da açın, StarDist eklentisini seçin, önceden eğitilmiş veya özel modeli seçin ve GUI üzerinden doğrudan tahmin yapın.
Eklentiler menüsünden StarDist eklentisini kullanarak görüntü yığını üzerinde sezgisel arayüzle model uygulayın.
Uzantıyı yükledikten sonra, histopatoloji analizi için QuPath’ın betik konsolu veya grafik arayüzü üzerinden StarDist tespiti yapın.
Eğitim ve İnce Ayar
Her nesnenin benzersiz şekilde etiketlendiği gerçek etiketli görüntüler oluşturun. Veri setinizi hazırlamak için LabKit, QuPath veya Fiji gibi anotasyon araçlarını kullanın.
StarDist Python API’sini kullanarak yeni model eğitin veya özel anotasyonlu verilerle mevcut modeli ince ayar yapın.
Son İşleme Seçenekleri
- Yinelenen aday şekilleri elemek için maksimum olmayan baskılama (NMS) uygulayın
- Yıldız-konveks olmayan şekiller için maskeleri birleştirmek üzere StarDist OPP (Nesne Son İşleme) kullanın
Sınırlamalar ve Dikkat Edilmesi Gerekenler
- Yıldız-konveks varsayımı çok konveks veya düzensiz şekilleri mükemmel modellemeyebilir
- Kurulum karmaşıklığı: özel kurulumlar uzantılar için uyumlu C++ derleyicisi gerektirir
- GPU hızlandırması uyumlu TensorFlow, CUDA ve cuDNN sürümlerine bağlıdır
- Bazı kullanıcılar Java yapılandırması nedeniyle ImageJ eklentisi çalıştırmada sorun bildirmiştir
Sıkça Sorulan Sorular
StarDist, esnek önceden eğitilmiş modelleri ve farklı görüntüleme modlarına uyumu sayesinde floresan, parlak alan ve histopatoloji (örneğin H&E) gibi çeşitli görüntü türleriyle çalışır.
Evet — StarDist, hacimsel veriler için yıldız-konveks polihedronlar kullanarak 3B örnek segmentasyonunu destekler ve 2B yeteneklerini tam 3B analize genişletir.
Gerekli değil. Önceden eğitilmiş modeller hazır olarak sunulur ve çoğu durumda doğrudan iyi sonuç verir. Ancak özel veya yeni veriler için anotasyon yapıp özel modeller eğitmek doğruluğu önemli ölçüde artırır.
StarDist, napari, ImageJ/Fiji ve QuPath ile entegre olur; böylece kodlama yapmadan GUI üzerinden segmentasyon yapabilirsiniz. Ayrıca gelişmiş iş akışları için doğrudan Python API kullanımı da desteklenir.
StarDist, segmentasyon performansını değerlendirmek için hassasiyet, geri çağırma, F1 skoru ve panoptik kalite gibi yaygın örnek segmentasyon ölçütlerini hesaplayan yerleşik fonksiyonlar sağlar.
SAM
Uygulama Bilgileri
| Geliştirici | Meta AI Araştırma (FAIR) |
| Desteklenen Cihazlar |
|
| Dil ve Erişilebilirlik | Küresel olarak erişilebilir açık kaynak temel model; dokümantasyon İngilizce |
| Fiyatlandırma | Ücretsiz — Meta lisansı altında GitHub ve MIB entegrasyonu ile açık kaynak |
Genel Bakış
SAM (Segment Anything Model), Meta tarafından oluşturulan güçlü bir yapay zeka temel modelidir ve görüntülerde neredeyse her nesnenin etkileşimli ve otomatik segmentasyonunu sağlar. Noktalar, sınırlayıcı kutular veya kaba maskeler gibi istemler kullanarak, SAM görev odaklı yeniden eğitim gerektirmeden segmentasyon maskeleri üretir. Mikroskopi araştırmalarında, SAM'ın esnekliği hücre segmentasyonu, organel tespiti ve histopatoloji analizine uyarlanmış olup, araştırmacılara istem tabanlı, genel amaçlı bir segmentasyon aracı sunar.
Ayrıntılı Tanıtım
Meta tarafından 1 milyardan fazla maske ve 11 milyon görüntü üzerinde eğitilen SAM, yeni alanlarda "sıfır atış" performansıyla segmentasyon için istemlenebilir temel model olarak tasarlanmıştır. Tıbbi görüntüleme araştırmalarında, SAM tüm slayt patoloji segmentasyonu, tümör tespiti ve hücre çekirdeği tanımlaması için değerlendirilmiştir. Ancak, yoğun paketlenmiş örneklerde—örneğin hücre çekirdeklerinde—performansı karışıktır: kapsamlı istemlere (örneğin, 20 tıklama veya kutu) rağmen, sıfır atış segmentasyon karmaşık mikroskopi görüntülerinde zorlanabilir.
Bu sınırlamayı aşmak için alan‑özgü uyarlamalar geliştirilmiştir:
- SAMCell — Çeşitli hücre tiplerinde güçlü sıfır atış segmentasyon için büyük mikroskopi veri setlerinde ince ayar yapılmıştır, deney başına yeniden eğitim gerektirmez
- μSAM — Küçük hücresel yapılar üzerinde doğruluğu artırmak için 17.000'den fazla elle anotasyonlu mikroskopi görüntüsü üzerinde yeniden eğitilmiştir
Temel Özellikler
Nokta, kutu ve maskelerle esnek etkileşim ve hassas kontrol.
Yeni görüntü alanlarında ince ayar yapmadan segmentasyon gerçekleştirir.
Mikroskopi ve histopatoloji için az sayıda örnek veya istem tabanlı yeniden eğitimle uyarlanabilir.
Mikroskopi Görüntü Tarayıcısı (MIB) içinde 3D ve interpolasyonlu segmentasyon desteğiyle kullanılabilir.
IDCC-SAM, immünositokimya verilerinde manuel anotasyon olmadan otomatik hücre sayımı sağlar.
İndirme veya Erişim
Kullanıcı Kılavuzu
- Mikroskopi Görüntü Tarayıcısını açın ve SAM segmentasyon paneline gidin
- Python yorumlayıcısını yapılandırın ve SAM-1 veya SAM-2 modellerinden birini seçin
- GPU hızlandırması için yürütme ortamında "cuda" seçin (optimum performans için önerilir)
- Nokta istemleri: Pozitif tohum tanımlamak için nesneye tıklayın; genişletmek için Shift + tıklama, negatif tohumlar için Ctrl + tıklama kullanın
- 3D yığınlar: Etkileşimli 3D modunu kullanın—bir dilime tıklayın, shift ile kaydırın ve dilimler arasında tohumları enterpolayın
- Ayarlama modu: Maskeleri değiştirin, ekleyin, çıkarın veya gerektiğinde yeni katman oluşturun
- SAM-2 panelindeki MIB'in "Her şeyi otomatik" seçeneğini kullanarak bir bölgedeki tüm görünür nesneleri segmentleyin
- Segmentasyon sonrası maskeleri gözden geçirin ve gerektiğinde iyileştirin
- Az sayıda kullanıcı isteminden piksel düzeyinde anotasyonlar oluşturmak için istem tabanlı ince ayar iş akışlarını (örneğin, "All-in-SAM") kullanın
- Hücre sayımı için, SAM'ı sıfır atış iş akışı ve son işlemle kullanan IDCC-SAM'ı uygulayın
- Yüksek doğruluklu hücre segmentasyonu için mikroskopi hücre görüntülerinde ince ayar yapılmış SAMCell'i kullanın
Sınırlamalar ve Dikkat Edilmesi Gerekenler
- Alan ince ayarı olmadan yoğun veya üst üste binen yapılarda sıfır atış performans tutarsızdır
- Segmentasyon kalitesi istem tasarımı ve stratejisine çok bağlıdır
- GPU şiddetle tavsiye edilir; CPU ile çıkarım çok yavaştır
- Çok yüksek çözünürlüklü tüm slayt görüntüleri ve çok ölçekli doku yapılarında zorlanabilir
- Mikroskopi için SAM'ı ince ayar yapmak veya uyarlamak makine öğrenimi bilgisi gerektirebilir
Sıkça Sorulan Sorular
Evet—özellikle hücre segmentasyonu görevleri için mikroskopi veri setlerinde SAM'ı ince ayar yapan SAMCell gibi uyarlamalarla.
Her zaman değil. IDCC-SAM ile manuel anotasyon olmadan sıfır atış hücre sayımı yapabilirsiniz.
İstem tabanlı ince ayar (örneğin, "All-in-SAM") veya 17.000'den fazla anotasyonlu mikroskopi görüntüsü üzerinde eğitilmiş μSAM gibi önceden eğitilmiş mikroskopi versiyonlarını kullanın.
CPU üzerinde mümkün olsa da, pratik çıkarım hızı ve gerçek zamanlı etkileşimli segmentasyon için GPU şiddetle tavsiye edilir.
Evet—MIB'in SAM-2 entegrasyonu, dilimler arasında tohum enterpolasyonu ile 3D segmentasyon ve hacimsel analiz desteği sunar.
AxonDeepSeg
| Geliştirici | Polytechnique Montréal ve Université de Montréal bünyesindeki NeuroPoly Lab |
| Desteklenen Platformlar |
|
| Dil | İngilizce dokümantasyon; dünya çapında kullanılan açık kaynak araç |
| Fiyatlandırma | Ücretsiz ve açık kaynaklı |
Genel Bakış
AxonDeepSeg, mikroskopi görüntülerinde aksonlar ve myelin kılıflarının otomatik segmentasyonu için yapay zeka destekli bir araçtır. Konvolüsyonel sinir ağları kullanarak TEM, SEM ve parlak alan mikroskopisi gibi çeşitli görüntüleme modlarında doğru üç sınıflı segmentasyon (akson, myelin, arka plan) sağlar. Akson çapı, g-oranı ve myelin kalınlığı gibi morfometrik ölçümleri otomatikleştirerek, AxonDeepSeg sinirbilim araştırmalarında nicel analizleri kolaylaştırır, manuel anotasyon süresini önemli ölçüde azaltır ve tekrarlanabilirliği artırır.
Temel Özellikler
TEM, SEM ve parlak alan mikroskopisi modları için optimize edilmiş kullanıma hazır modeller.
Mikroskopi görüntülerinde akson, myelin ve arka plan bölgelerinin hassas sınıflandırması.
Akson çapı, g-oranı, myelin kalınlığı ve yoğunluk metriklerinin otomatik hesaplanması.
Napari GUI entegrasyonu, segmentasyon maskelerinin manuel olarak düzeltilmesine olanak tanır ve doğruluğu artırır.
Büyük ölçekli sinir dokusu analizleri için özel iş akışlarına sorunsuz entegrasyon sağlar.
Kapsamlı test betikleri, tekrarlanabilirlik ve güvenilir segmentasyon sonuçları sağlar.
Teknik Detaylar
NeuroPoly Lab tarafından geliştirilen AxonDeepSeg, sinirbilim uygulamaları için yüksek hassasiyetli segmentasyon sunmak üzere derin öğrenmeyi kullanır. Farklı mikroskopi modları için önceden eğitilmiş modeller mevcuttur ve bu sayede görüntüleme teknikleri arasında çok yönlülük sağlar. Araç, Napari ile entegre olarak segmentasyon maskelerinin etkileşimli olarak düzeltilmesine imkan verir; bu da zorlu veri setlerinde doğruluğu artırır. AxonDeepSeg, sinir dokusu yapısı ve patolojisinin yüksek verimli çalışmaları için temel morfometrik metrikleri hesaplar. Python tabanlı yapısı, akson ve myelin morfolojisinin büyük ölçekli analizlerine yönelik özel iş akışlarına entegrasyonu kolaylaştırır.
İndirme veya Erişim
Kurulum ve Ayarlar
Python 3.8 veya daha yeni sürümün yüklü olduğundan emin olun, ardından pip ile AxonDeepSeg ve Napari’yi yükleyin:
pip install axondeepseg napariTüm bileşenlerin doğru yüklendiğini ve çalıştığını doğrulamak için sağlanan test betiklerini çalıştırın.
Mikroskopi görüntülerinizi (TEM, SEM veya parlak alan) Napari veya Python ortamınıza aktarın.
Görüntüleme modunuza uygun önceden eğitilmiş modeli seçin ve akson ile myelin maskelerini oluşturmak için segmentasyonu çalıştırın.
Akson çapı, g-oranı, yoğunluk ve myelin kalınlığı gibi morfometrik ölçümleri otomatik olarak hesaplayın ve sonuçları CSV formatında dışa aktarın.
Gerekli durumlarda Napari GUI kullanarak segmentasyon maskelerini manuel olarak düzenleyin, maskeleri birleştirin veya silin, böylece doğruluğu artırın.
Önemli Hususlar
- Yeni veya eğitilmemiş görüntüleme modlarında performans düşebilir
- Zorlu veya karmaşık bölgeler için manuel düzeltmeler gerekebilir
- Büyük veri setlerinin daha hızlı işlenmesi için GPU önerilir; CPU ile de işlem yapılabilir
Sıkça Sorulan Sorular
AxonDeepSeg, TEM (Geçirimli Elektron Mikroskobu), SEM (Taramalı Elektron Mikroskobu) ve parlak alan mikroskopisi için her modaya optimize edilmiş önceden eğitilmiş modellerle destek sağlar.
Evet, AxonDeepSeg tamamen ücretsiz ve açık kaynaklıdır; akademik ve ticari kullanıma açıktır.
Evet, AxonDeepSeg segmentasyonlu görüntülerden akson çapı, g-oranı, myelin kalınlığı ve yoğunluk metriklerini otomatik olarak hesaplar.
Büyük veri setlerinin daha hızlı segmentasyonu için GPU önerilir, ancak küçük ölçekli analizlerde CPU ile de çalıştırılabilir.
Evet, Napari GUI entegrasyonu, zorlu bölgelerde segmentasyon maskelerinin etkileşimli olarak düzeltilmesine ve iyileştirilmesine olanak tanır.
Ilastik
| Geliştirici | Avrupa Moleküler Biyoloji Laboratuvarı (EMBL) Ilastik Ekibi ve ilişkili akademik ortaklar |
| Desteklenen Platformlar |
|
| Dil | İngilizce |
| Fiyatlandırma | Ücretsiz ve açık kaynaklı |
Genel Bakış
Ilastik, mikroskopi verilerinin etkileşimli görüntü segmentasyonu, sınıflandırması ve analizi için güçlü, yapay zeka destekli bir araçtır. Random Forest sınıflandırıcıları gibi makine öğrenimi tekniklerini kullanarak araştırmacıların pikselleri segmentlemesine, nesneleri sınıflandırmasına, hücreleri zaman içinde izlemesine ve hem 2B hem de 3B veri setlerinde yoğunluk sayımı yapmasına olanak tanır. Sezgisel arayüzü ve gerçek zamanlı geri bildirimi sayesinde Ilastik, programlama bilgisi olmayan bilim insanları tarafından erişilebilir olup hücre biyolojisi, sinirbilim ve biyomedikal görüntülemede yaygın olarak kullanılmaktadır.
Temel Özellikler
Temsilci bölgeleri not ederken anlık segmentasyon sonuçları için gerçek zamanlı geri bildirim.
Segmentlenen yapıları morfolojik ve yoğunluk özelliklerine göre kategorize edin.
2B ve 3B zaman serisi mikroskopi deneylerinde hücre hareketi ve bölünmesini izleyin.
Bireysel nesnelerin açık segmentasyonu olmadan kalabalık bölgeleri sayısal olarak değerlendirin.
Karmaşık 3B hacimler için sezgisel etkileşimle yarı otomatik segmentasyon.
Komut satırı modunda çoklu görüntüleri otomatik olarak işleyin.
İndir
Başlangıç Kılavuzu
Resmi web sitesinden işletim sisteminize uygun Ilastik sürümünü indirin. Paket, gerekli tüm Python bağımlılıklarını içerir; platformunuza uygun kurulum talimatlarını izleyin.
Ilastik’i açın ve analiz iş akışınızı seçin: Piksel Sınıflandırması, Nesne Sınıflandırması, İzleme veya Yoğunluk Sayımı. Çok kanallı, 3B veya zaman serisi görüntülerinizi yükleyin.
Görüntülerinizde birkaç temsilci piksel veya nesneyi etiketleyin. Ilastik’in Random Forest sınıflandırıcısı bu notlardan öğrenir ve tüm veri setinizde otomatik olarak etiket tahmini yapar.
Eğitilmiş modeli kullanarak tüm veri setinizi segmentleyin veya sınıflandırın. Sonuçları etiketli görüntüler, olasılık haritaları veya nicel tablolar olarak dışa aktararak sonraki analiz ve görselleştirme için kullanın.
Büyük ölçekli analiz süreçleri için Ilastik’in komut satırı modunu kullanarak çoklu görüntüleri manuel müdahale olmadan otomatik işleyin.
Sınırlamalar ve Dikkat Edilmesi Gerekenler
- Etkileşimli etiketleme çok büyük veri setlerinde zaman alıcı olabilir
- Doğruluk kullanıcı notlarının kalitesi ve temsil yeteneğine bağlıdır
- Bellek gereksinimleri — çok yüksek çözünürlüklü veya çok gigabaytlık veri setleri önemli RAM gerektirebilir
- Karmaşık veriler — Random Forest sınıflandırıcılar, çok değişken veya karmaşık görüntü verilerinde derin sinir ağlarına kıyasla daha düşük performans gösterebilir
Sıkça Sorulan Sorular
Evet, Ilastik segmentasyon, izleme ve çoklu zaman noktalarında nicel analiz için 3B hacimleri ve zaman serisi deneylerini tam olarak destekler.
Evet, Ilastik tamamen ücretsiz ve açık kaynaklıdır, tüm kullanıcılar için lisans kısıtlaması olmadan sunulmaktadır.
Hayır, Ilastik sezgisel grafik arayüzü ve gerçek zamanlı geri bildirimi ile programlama bilgisi olmayan araştırmacılar için erişilebilirdir. İleri düzey kullanıcılar komut satırı toplu işleme modunu da kullanabilir.
Evet, özel izleme iş akışı 2B ve 3B zaman serisi veri setlerinde hücre hareketi ve bölünmesini otomatik soy takibi ile analiz etmeye olanak tanır.
Segmentasyon çıktıları etiketli görüntüler, olasılık haritaları veya nicel tablolar olarak dışa aktarılabilir; böylece sonraki analiz araçları ve görselleştirme yazılımlarıyla sorunsuz entegrasyon sağlanır.
Bu araçlar acemiden uzmana kadar geniş bir yelpazeyi kapsar. Birçoğu ücretsiz ve açık kaynaklıdır, böylece araştırma topluluğunda tekrarlanabilir ve paylaşılabilir yapay zeka iş akışlarını kolaylaştırır.
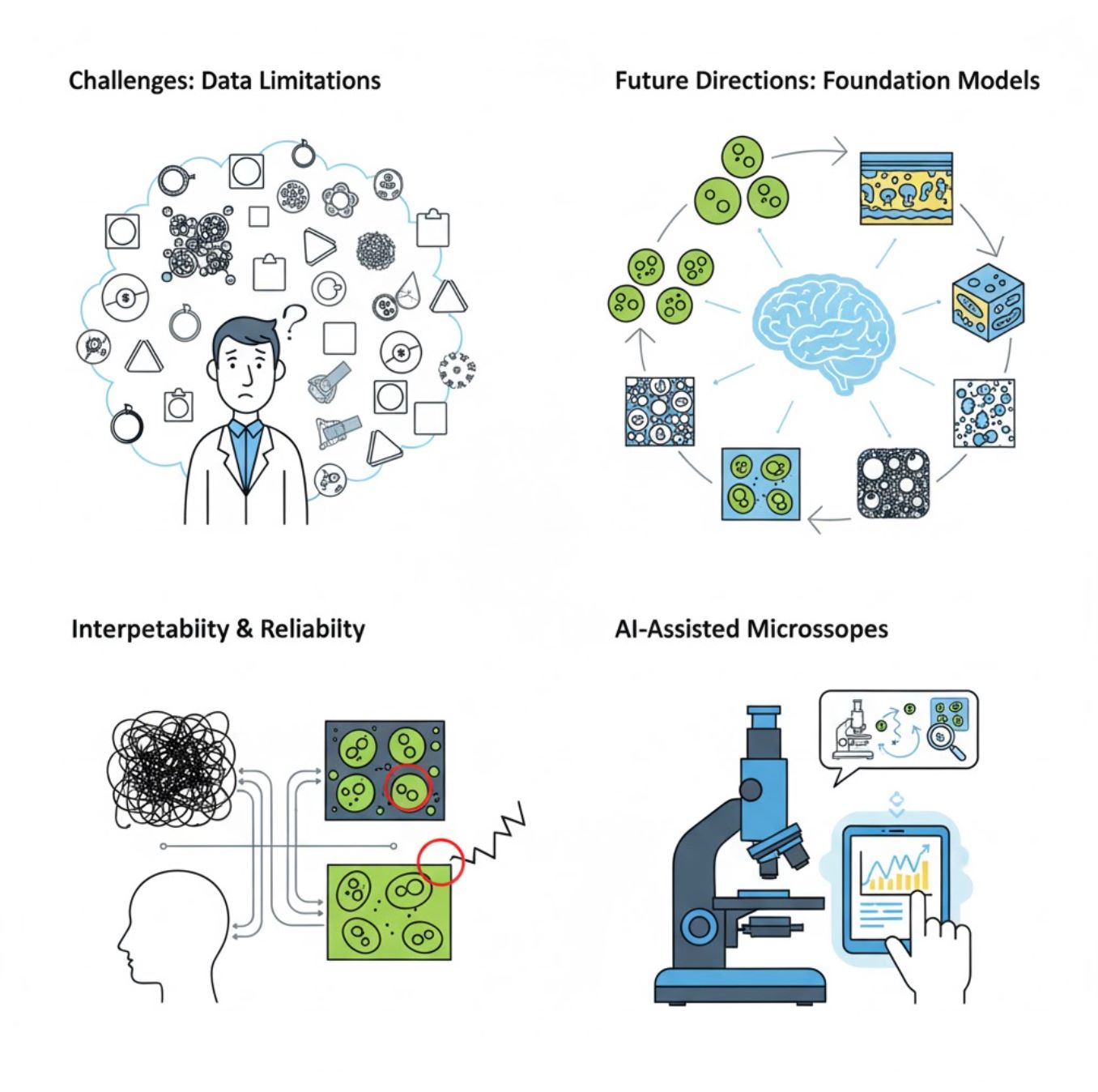
Zorluklar ve Gelecek Yönelimleri
Mevcut Zorluklar
Ortaya Çıkan Trendler
Görsel Temel Modeller
Yeni nesil yapay zeka sistemleri, görev bazlı eğitime olan ihtiyacı azaltmayı vaat ediyor.
- SAM ve CLIP tabanlı sistemler gibi modeller
- Tek bir yapay zeka birçok mikroskopi görevini yönetir
- Daha hızlı dağıtım ve uyarlama
Yapay Zeka Destekli Mikroskoplar
Tam otonom ve akıllı mikroskopi sistemleri gerçeğe dönüşüyor.
- LLM’ler aracılığıyla doğal dil kontrolü
- Tam otomatik geri bildirim döngüleri
- Gelişmiş mikroskopi erişimini demokratikleştirir
Temel Çıkarımlar
- Yapay zeka, mikroskop görüntü işlemede doğruluk ve otomasyonu artırarak hızla dönüşüm yaratıyor
- Derin öğrenme, karmaşık ve değişken mikroskopi görüntülerinde geleneksel makine öğrenimini geride bırakıyor
- CNN’ler ham piksellerden hiyerarşik özellikleri otomatik öğrenerek sağlam analiz sağlar
- Ana uygulamalar segmentasyon, sınıflandırma, takip, gürültü azaltma ve otomatik edinimi içerir
- Başarı, kaliteli veri ve uzman doğrulamasına bağlıdır
- Görsel temel modeller ve yapay zeka destekli mikroskoplar alanın geleceğini temsil eder
Sürekli ilerlemeler ve topluluk çabaları (açık kaynak araçlar, paylaşılan veri setleri) ile yapay zeka, mikroskobun "gözü"nün temel bir parçası haline gelerek bilim insanlarının görünmeyeni görmesine yardımcı olacak.







No comments yet. Be the first to comment!