MI a mikroszkópos képfeldolgozásban
A mesterséges intelligencia forradalmasítja a mikroszkópos képfeldolgozást olyan erőteljes képességekkel, mint a precíz szegmentáció, zajcsökkentés, szuperfelbontás és az automatikus képrögzítés. Ez a cikk kiemeli az alapvető MI-eszközöket és a tudományos kutatásban megjelenő trendeket.
A mesterséges intelligencia technikák forradalmasítják a mikroszkópiát az képrögzítés optimalizálásával és az elemzés automatizálásával. A modern okosmikrószkópokban az MI modulok képesek valós időben állítani a képalkotási paramétereket (pl. fókusz, megvilágítás), hogy minimalizálják a fotoblekhelést és javítsák a jelminőséget. Eközben a mélytanuló algoritmusok képesek átvizsgálni a komplex képadatokat, hogy rejtett biológiai információkat tárjanak fel, sőt összekapcsolják a képeket más adatokkal (pl. genomika).
MI módszerek: gépi tanulás vs mélytanulás
Az MI módszerek a klasszikus gépi tanulást (ML)ól a modern mélytanulásig (DL) terjednek. Mindkét megközelítésnek megvannak a maga erősségei és korlátai:
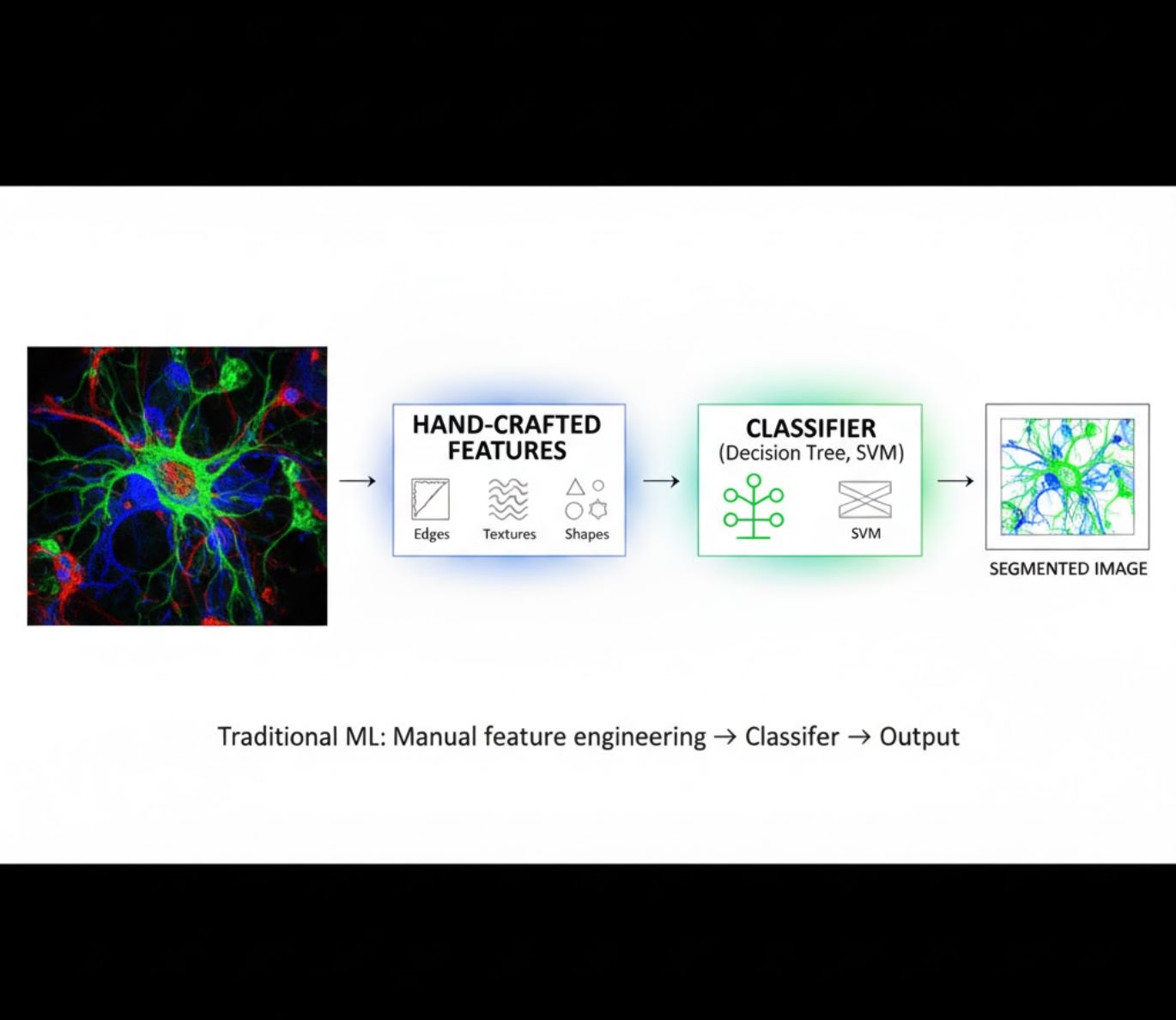
Kézzel készített jellemzők
- A kutatók manuálisan alkotják meg a képi jellemzőket (élek, textúrák, formák)
- A jellemzőket osztályozóknak adják át (döntési fák, SVM)
- Gyors tanítás
- Nehezen boldogul komplex vagy zajos képekkel
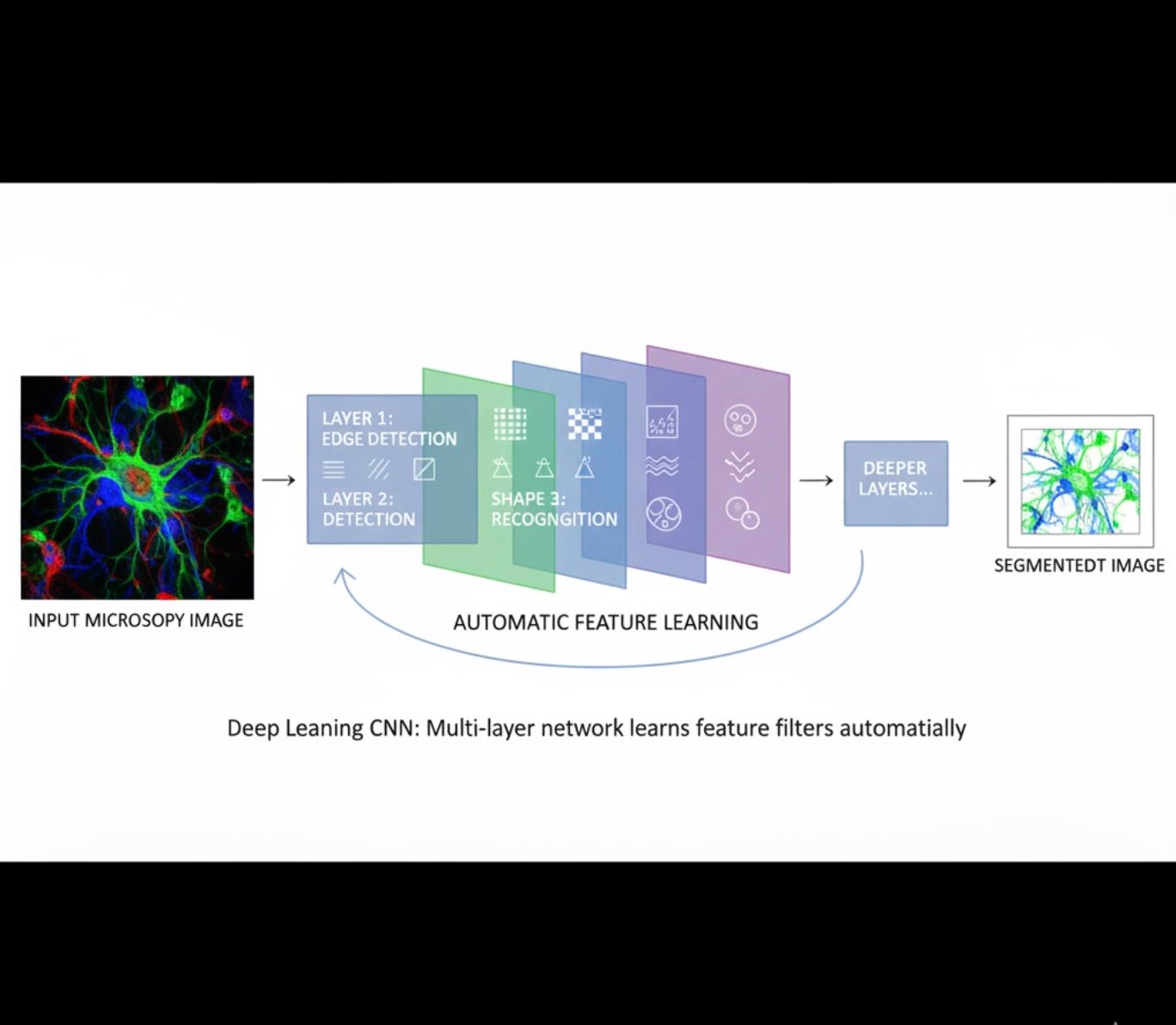
Automatikus jellemzőtanulás
- Többrétegű neurális hálózatok (CNN-ek) automatikusan tanulják a jellemzőket
- Végponttól végpontig tanulás nyers pixelekből
- Sokkal robusztusabb a változásokkal szemben
- Megbízhatóan megragadja az összetett textúrákat és struktúrákat
Hogyan működnek a CNN-ek: A konvolúciós neurális hálózatok egymást követő szűrőket alkalmaznak a mikroszkópos képeken, megtanulva az egyszerű mintázatok (élek) felismerését az első rétegekben, és az összetett struktúrák (sejtformák, textúrák) felismerését a mélyebb rétegekben. Ez a hierarchikus tanulás teszi a mélytanulást rendkívül robusztussá, még akkor is, ha az intenzitásprofilok jelentősen változnak.
Vizuális összehasonlítás: ML vs DL folyamatok
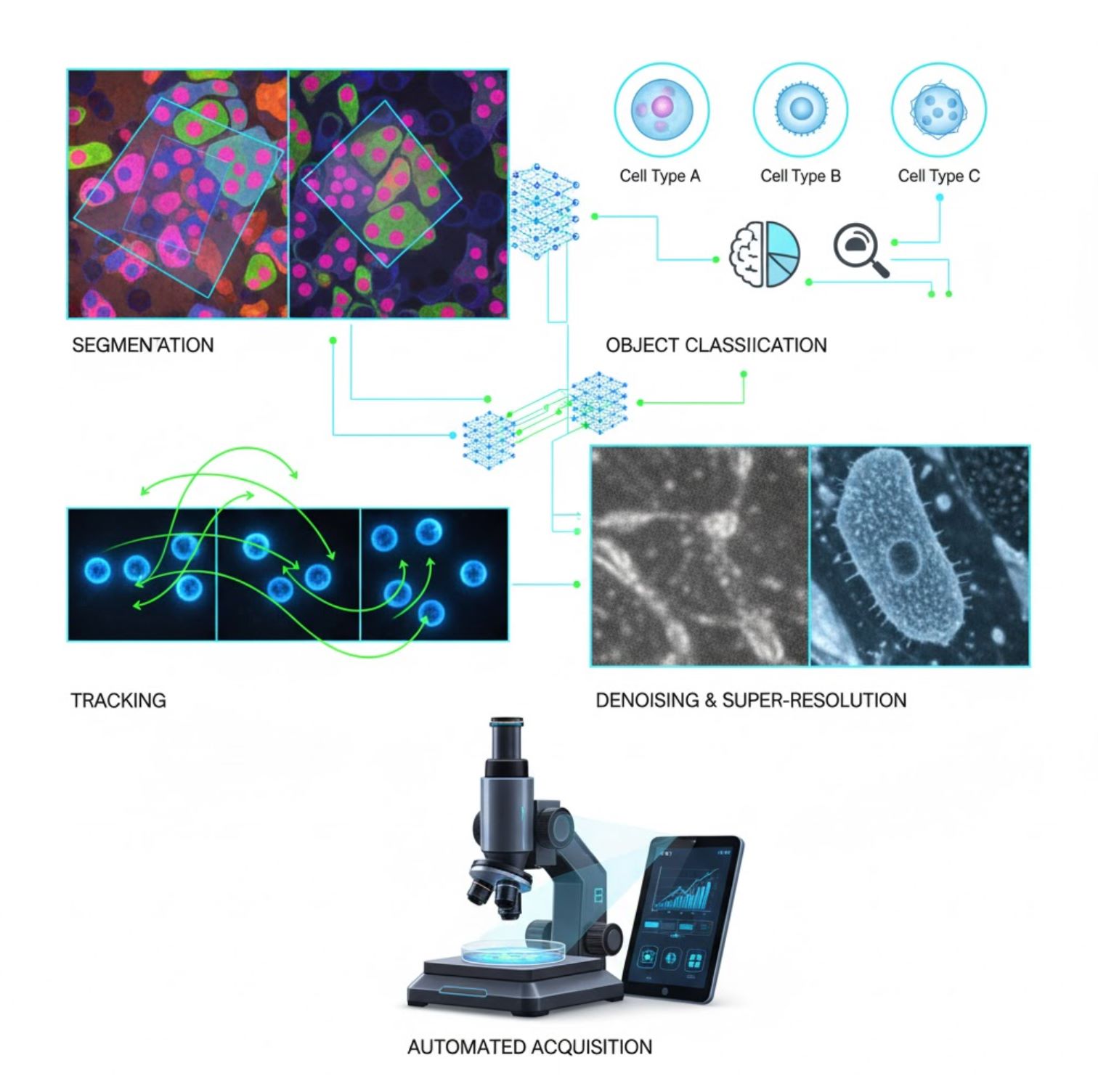
Kulcsfontosságú MI alkalmazások a mikroszkópiában
Az MI ma már számos képfeldolgozási feladatban jelen van a mikroszkópos munkafolyamat során:
Szegmentáció
Képek régiókra bontása (pl. minden sejt vagy sejtmag azonosítása). Mély hálózatok, mint az U-Net, kiválóak ebben a feladatban.
- Szemiantikus szegmentáció: pixelenkénti osztálycímkék
- Objektum szegmentáció: egyedi objektumok elkülönítése
- Magas pontosság zsúfolt vagy gyenge fényviszonyok között
- Vision alapú modellek (pl. μSAM) már mikroszkópiára adaptálva
Objektumosztályozás
A szegmentáció után az MI nagy pontossággal osztályozza az egyes objektumokat.
- Sejttípus azonosítás
- Mitotikus fázis meghatározás
- Patológiai jelzők felismerése
- Finom fenotípusok megkülönböztetése, melyeket nehéz manuálisan mérni
Követés
Időbeli mikroszkópiában az MI páratlan pontossággal követi a sejteket vagy részecskéket a képkockák között.
- A mélytanulás drámaian javítja a követés pontosságát
- Megbízható elemzést tesz lehetővé mozgó sejtek esetén
- Dinamikus biológiai folyamatokat rögzít
Zajcsökkentés és szuperfelbontás
Az MI modellek javítják a képminőséget zaj és elmosódás eltávolításával.
- Fizikailag tájékozott mély modellek tanulják a mikroszkóp optikáját
- Élesebb, műtermékmentes képek rekonstruálása
- Magasabb felbontás kevesebb műtermékkel a hagyományos módszerekhez képest
Automatikus képrögzítés
Az MI valós időben irányítja magát a mikroszkópot.
- Élő képek elemzése intelligens döntésekhez
- Automatikusan állítja a fókuszt és pásztázza az érdekes területeket
- Csökkenti a fototoxicitást és időt takarít meg
- Lehetővé teszi a nagy áteresztőképességű és adaptív képalkotó kísérleteket
Népszerű MI eszközök a mikroszkópos képfeldolgozásban
A rich ecosystem of tools supports AI in microscopy. Researchers have built both general-purpose and specialized software, many open-source:
Cellpose
| Fejlesztő | Carsen Stringer és Marius Pachitariu (MouseLand kutatócsoport) |
| Támogatott platformok |
Python szükséges (pip/conda telepítés). Grafikus felület csak asztali gépen érhető el. |
| Nyelvi támogatás | Angol dokumentáció; világszerte elfogadott kutatóintézetekben |
| Árazási modell | Ingyenes és nyílt forráskódú BSD-3-Clause licenc alatt |
Áttekintés
A Cellpose egy fejlett, mélytanuláson alapuló szegmentációs eszköz mikroszkópos képekhez. Általános algoritmusként pontosan szegmentál különféle sejttípusokat (magok, citoplazma stb.) különböző képalkotó módokon anélkül, hogy a modellt újra kellene tanítani. Az emberi beavatkozással történő finomhangolás lehetőségével a kutatók javíthatják az eredményeket, adaptálhatják a modellt saját adataikhoz, és alkalmazhatják a rendszert 2D és 3D képalkotási munkafolyamatokban egyaránt.
Főbb jellemzők
Széles körű sejttípusokra, festésekre és képalkotó módokra „dobozból kivéve” működik, egyedi tanítás nélkül.
Teljes 3D adathalmazokat támogat a „2,5D” megközelítéssel, amely újrahasznosítja a 2D modelleket térfogati adatokhoz.
Kézi korrekcióval javíthatja a szegmentációs eredményeket, és újrataníthatja a modellt saját adataira a pontosság növelése érdekében.
Python API, parancssori vagy grafikus felületen keresztül érhető el a rugalmas munkafolyamatokhoz.
Zajcsökkentés, elmosódás eltávolítása és felbontásnövelés a szegmentáció előtti képminőség javítására.
Letöltés vagy hozzáférés
Műszaki háttér
A Cellpose-t Stringer, Wang, Michaelos és Pachitariu egy alapvető tanulmányban mutatta be, amelyben egy nagy és rendkívül változatos adatbázison tanították, több mint 70 000 szegmentált objektummal. Ez a sokszínűség lehetővé teszi, hogy a modell általánosítani tudjon sejttípusok, méretek és mikroszkópos beállítások között, jelentősen csökkentve az egyedi tanítás szükségességét a legtöbb esetben. 3D adatok esetén a Cellpose ügyesen újrahasznosítja a 2D modellt „2,5D” módon, így nem kell teljesen 3D címkézett tanítóadatokat használni, miközben térfogati szegmentációt biztosít. A Cellpose 2.0 bevezette az emberi beavatkozással történő újratanítást, amely lehetővé teszi a felhasználók számára, hogy kézzel korrigálják az előrejelzéseket és saját képeiken újratanítsák a modellt a jobb teljesítmény érdekében.
Telepítés és beállítás
Állítson be egy Python környezetet conda használatával:
conda create -n cellpose python=3.10Aktiválja a környezetet és telepítse a Cellpose-t:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeElső lépések
Grafikus felület (GUI) mód
- Indítsa el a GUI-t a következő parancs futtatásával:
python -m cellpose - Húzza be a képfájlokat (
.tif,.pngstb.) az interfészbe - Válassza ki a modell típusát (pl. „cyto” a citoplazmához vagy „nuclei” a magokhoz)
- Állítsa be a becsült sejtméretet, vagy hagyja, hogy a Cellpose automatikusan kalibráljon
- Kattintson a szegmentáció indításához és az eredmények megtekintéséhez
Python API mód
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Finomhangolás és újratanítás
- A maszkolás után kézzel javítsa a szegmentációt a GUI-ban maszkolatok egyesítésével vagy törlésével
- Használja a beépített tanítási funkciókat a javított példákon történő újratanításhoz
- Javított modell teljesítmény az adott adathalmazon
3D adatok feldolgozása
- Töltsön be több-Z TIFF vagy térfogati adathalmazt
- Használja a
--Zstackkapcsolót a GUI-ban vagy API-ban 3D feldolgozáshoz - Opcionálisan finomítsa a 3D folyamatokat simítással vagy speciális paraméterekkel a jobb szegmentáció érdekében
Korlátozások és megfontolások
- Modell általánosságának kompromisszuma: Bár az általános modell széles körben működik, nagyon szokatlan sejttípusok vagy képalkotási körülmények esetén újratanítás szükséges lehet.
- Annotációs munka: Az emberi beavatkozással történő tanítás kézi korrekciókat igényel, ami nagy adathalmazok esetén időigényes lehet.
- Telepítés összetettsége: A GUI telepítése parancssori használatot, conda környezeteket és Python függőségek kezelését igényli — nem mindig egyszerű nem programozók számára.
- Csak asztali használat: A Cellpose asztali használatra készült; nincs natív Android vagy iOS alkalmazás.
Gyakran ismételt kérdések
Nem — a Cellpose előre betanított, általános modelleket kínál, amelyek gyakran jól működnek újratanítás nélkül. Azonban speciális vagy szokatlan adatok esetén az optimális eredmény érdekében annotálhat és újrataníthat a human-in-the-loop funkciókkal.
Igen — támogatja a 3D-t a 2D modell újrahasznosításával („2,5D”), és térfogati adathalmazokat is feldolgozhat a GUI-n vagy API-n keresztül.
A gyorsabb feldolgozás és tanítás érdekében erősen ajánlott GPU, különösen nagy vagy 3D adathalmazok esetén, de a Cellpose CPU-only gépeken is futtatható, lassabb teljesítménnyel.
A GUI-ban manuálisan beállíthatja a becsült sejtméretet, vagy hagyhatja, hogy a Cellpose automatikusan kalibráljon. Az eredményeket finomhangolhatja és újrataníthatja, ha a szegmentáció nem optimális.
Igen — az újabb verziók (Cellpose 3) tartalmaznak képjavító modelleket zajcsökkentésre, elmosódás eltávolítására és felbontásnövelésre, hogy javítsák a szegmentáció minőségét a feldolgozás előtt.
StarDist
| Fejlesztők | Uwe Schmidt, Martin Weigert, Coleman Broaddus és Gene Myers |
| Támogatott platformok |
|
| Nyelvi támogatás | Nyílt forráskódú projekt, dokumentációval és közösséggel elsősorban angol nyelven |
| Árazási modell | Ingyenes és nyílt forráskódú. BSD-3-Clause licenc alatt. |
Áttekintés
A StarDist egy mélytanuláson alapuló eszköz példány szegmentációra mikroszkópos képeken. Minden objektumot (például sejtmagi) csillag-konvex sokszögként (2D) vagy poliéderként (3D) ábrázol, lehetővé téve a sűrűn elhelyezkedő vagy átfedő objektumok pontos felismerését és elkülönítését. Robusztus architektúrájával a StarDist széles körben használatos automatikus sejt- és mag szegmentációra fluoreszcens mikroszkópiában, hisztopatológiában és más bio-képalkotó elemzési alkalmazásokban.
Főbb jellemzők
Nagyon pontos példány szegmentáció csillag-konvex sokszögek (2D) és poliéderek (3D) segítségével a megbízható objektumfelismerés érdekében.
Dedikált modellek 2D képekhez és 3D térfogati adatokhoz a teljes körű mikroszkópos elemzéshez.
Kész modellek fluoreszcens magvakhoz, H&E festett hisztológiához és más gyakori képalkotó helyzetekhez.
Az észlelt objektumokat különböző osztályokba sorolja (pl. különböző sejttípusok) egyetlen szegmentációs futtatás során.
Zökkenőmentes integráció az ImageJ/Fiji, QuPath és napari programokkal, elérhető GUI-alapú munkafolyamatokhoz.
Átfogó példány szegmentációs értékelés, beleértve a pontosságot, visszahívást, F1 pontszámot és panoráma minőséget.
Műszaki háttér
Eredetileg a MICCAI 2018 konferencián bemutatott StarDist alapvető újítása a sugárirányú távolságok előrejelzése fix sugarak mentén, kombinálva az objektum valószínűségével minden pixelre, ami lehetővé teszi a csillag-konvex alakzatok pontos rekonstrukcióját. Ez a megközelítés megbízhatóan szegmentálja a szorosan érintkező objektumokat, amelyeket hagyományos pixel-alapú vagy határoló dobozos módszerekkel nehéz elkülöníteni.
A közelmúltbeli fejlesztések kiterjesztették a StarDist-ot hisztopatológiai képekre is, lehetővé téve nemcsak a magok szegmentációját, hanem az észlelt objektumok többosztályos osztályozását is. A módszer kiemelkedő eredményeket ért el olyan kihívásokon, mint a CoNIC (Colon Nuclei Identification and Counting) verseny.
Letöltés vagy hozzáférés
Telepítés és beállítás
Telepítse a TensorFlow-t (1.x vagy 2.x verzió) a StarDist előfeltételeként.
Használja a pip-et a StarDist Python csomag telepítéséhez:
pip install stardistnapari esetén:
pip install stardist-napariQuPath esetén: Telepítse a StarDist kiterjesztést úgy, hogy a .jar fájlt a QuPath-ba húzza.
ImageJ/Fiji esetén: Használja a beépített plugin kezelőt vagy manuális telepítést a plugin menüből.
Szegmentáció futtatása
Töltsön be egy előre betanított modellt, normalizálja a képét, és futtassa az előrejelzést:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Nyissa meg a képét napari-ban, válassza ki a StarDist plugint, válasszon előre betanított vagy egyéni modellt, és futtassa az előrejelzést közvetlenül a GUI-ból.
Használja a StarDist plugint a Plugins menüből, hogy egy intuitív felületen alkalmazzon modellt a képsorozatára.
A kiterjesztés telepítése után futtassa a StarDist detektálást a QuPath szkript konzolján vagy grafikus felületén hisztopatológiai elemzéshez.
Tanítás és finomhangolás
Készítsen valós címkézésű képeket, ahol minden objektum egyedi címkét kap. Használjon annotációs eszközöket, mint a LabKit, QuPath vagy Fiji az adatkészlet előkészítéséhez.
Használja a StarDist Python API-t új modell tanításához vagy meglévő finomhangolásához a saját annotált adataival.
Utófeldolgozási lehetőségek
- Alkalmazzon nem maximum szűrést (NMS) a felesleges jelölt alakzatok eltávolításához
- Használja a StarDist OPP-t (Object Post-Processing) a nem csillag-konvex alakzatok maszkolásának egyesítéséhez
Korlátozások és megfontolások
- A csillag-konvex feltételezés nem modellezi tökéletesen a nagyon nem konvex vagy szabálytalan alakzatokat
- Telepítési bonyolultság: egyedi telepítésekhez kompatibilis C++ fordító szükséges a kiterjesztésekhez
- A GPU gyorsítás kompatibilis TensorFlow, CUDA és cuDNN verzióktól függ
- Néhány felhasználó Java konfigurációs problémákat jelent az ImageJ plugin futtatásakor
Gyakran ismételt kérdések
A StarDist különféle kép típusokkal működik, beleértve a fluoreszcens, fényterű és hisztopatológiai (pl. H&E) képeket, köszönhetően rugalmas előre betanított modelljeinek és a különböző képalkotó modalitásokhoz való alkalmazkodásának.
Igen — a StarDist támogatja a 3D példány szegmentációt csillag-konvex poliéderekkel térfogati adatok esetén, kiterjesztve a 2D képességeket teljes 3D elemzésre.
Nem feltétlenül. Elérhetők előre betanított modellek, amelyek gyakran jól működnek azonnal. Ugyanakkor speciális vagy új adatok esetén az annotálás és egyéni modellek tanítása jelentősen javítja a pontosságot.
A StarDist integrálható a napari, ImageJ/Fiji és QuPath programokkal, lehetővé téve a szegmentáció futtatását GUI-ból kódolás nélkül. Emellett támogatja a közvetlen Python API használatot fejlettebb munkafolyamatokhoz.
A StarDist beépített funkciókat kínál a gyakori példány szegmentációs metrikák kiszámítására, beleértve a pontosságot, visszahívást, F1 pontszámot és panoráma minőséget a szegmentáció teljesítményének értékeléséhez.
SAM
Alkalmazási információk
| Fejlesztő | Meta AI Research (FAIR) |
| Támogatott eszközök |
|
| Nyelv és elérhetőség | Nyílt forráskódú alapmodell világszerte elérhető; dokumentáció angol nyelven |
| Árazás | Ingyenes — nyílt forráskódú a Meta licenc alatt, GitHubon és MIB integráción keresztül |
Általános áttekintés
A SAM (Segment Anything Model) egy erőteljes mesterséges intelligencia alapmodell, amelyet a Meta hozott létre, és amely lehetővé teszi szinte bármilyen objektum interaktív és automatikus szegmentálását képeken. Pontok, határoló dobozok vagy durva maszkok használatával a SAM szegmentációs maszkokat generál anélkül, hogy feladatspecifikus újratanításra lenne szükség. A mikroszkópos kutatásban a SAM rugalmasságát adaptálták sejtszegmentálásra, sejtszervecske-felismerésre és hisztopatológiai elemzésre, így skálázható megoldást kínál kutatóknak, akik egy promptalapú, általános célú szegmentációs eszközt keresnek.
Részletes bemutatás
Eredetileg a Meta több mint 1 milliárd maszkon keresztül 11 millió képen tanította a SAM-ot, amelyet promptalapú alapmodellként terveztek szegmentációra, "zero-shot" teljesítménnyel új doménekben. Az orvosi képalkotó kutatásban a SAM-ot értékelték teljes metszetű patológiai szegmentációra, daganatfelismerésre és sejtmagi azonosításra. Telített példányok esetén – például sejtmagi esetén – a teljesítmény vegyes: még kiterjedt promptokkal (pl. 20 kattintás vagy doboz) is nehézségek adódhatnak összetett mikroszkópos képeken.
Ennek a korlátnak a kezelésére doménspecifikus adaptációk jelentek meg:
- SAMCell — nagy mikroszkópos adathalmazokon finomhangolva, erős zero-shot szegmentációt biztosítva különböző sejttípusokon, kísérletenkénti újratanítás nélkül
- μSAM — több mint 17 000 kézzel annotált mikroszkópos képen újratanítva a kis sejtes struktúrák pontosságának javítására
Főbb jellemzők
Rugalmas interakció pontok, dobozok és maszkok használatával a precíz vezérlés érdekében.
Új képtartományokon finomhangolás nélkül végez szegmentálást.
Alkalmazkodik mikroszkópos és hisztopatológiai feladatokhoz kevés példás vagy promptalapú újratanítással.
Elérhető a Microscopy Image Browserben (MIB) 3D és interpolált szegmentálás támogatással.
Az IDCC-SAM automatikus sejtszámlálást tesz lehetővé immuncitokémiai adatokban manuális annotáció nélkül.
Letöltés vagy hozzáférés
Felhasználói útmutató
- Nyissa meg a Microscopy Image Browser-t, és navigáljon a SAM szegmentációs panelhez
- Állítsa be a Python értelmezőt, és válasszon a SAM-1 vagy SAM-2 modellek közül
- GPU gyorsításhoz válassza a "cuda" végrehajtási környezetet (ajánlott az optimális teljesítményhez)
- Pont promptok: Kattintson egy objektumra pozitív mag definiálásához; használja a Shift + kattintás bővítéshez és a Ctrl + kattintás negatív magokhoz
- 3D rétegek: Használja az Interaktív 3D módot – kattintson egy szeletre, shift görgessen, és interpolálja a magokat a szeletek között
- Módosítási mód: Maszkok cseréje, hozzáadása, kivonása vagy új réteg létrehozása szükség szerint
- Használja a MIB "Automatikus minden" opcióját a SAM-2 panelen az adott területen látható összes objektum szegmentálásához
- Szükség szerint ellenőrizze és finomítsa a maszkokat a szegmentálás után
- Használja a promptalapú finomhangolási folyamatokat (pl. "All-in-SAM") a pixelek szintű annotációk létrehozásához ritka felhasználói promptokból
- Sejtszámláláshoz alkalmazza az IDCC-SAM-ot, amely a SAM-ot zero-shot folyamatban használja utófeldolgozással
- Magas pontosságú sejtszegmentáláshoz használja a SAMCell-t, amely mikroszkópos sejtképeken finomhangolt
Korlátozások és megfontolások
- Zero-shot teljesítmény következetlen sűrű vagy átfedő struktúrákon domain finomhangolás nélkül
- A szegmentálás minősége erősen függ a prompt tervezésétől és stratégiájától
- GPU erősen ajánlott; CPU-n az inferencia nagyon lassú
- Nehézségek nagyon nagy felbontású teljes metszetű képekkel és többskálás szöveti struktúrákkal
- A SAM mikroszkópos finomhangolása vagy adaptálása gépi tanulási jártasságot igényelhet
Gyakran ismételt kérdések
Igen – olyan adaptációkon keresztül, mint a SAMCell, amely a SAM-ot kifejezetten mikroszkópos adathalmazokon finomhangolja sejtszegmentálási feladatokra.
Nem mindig. Az IDCC-SAM segítségével zero-shot sejtszámlálás végezhető manuális annotáció nélkül.
Használjon promptalapú finomhangolást (pl. "All-in-SAM") vagy előre betanított mikroszkópos verziókat, mint a μSAM, amely több mint 17 000 annotált mikroszkópos képen tanult.
Bár CPU-n is lehetséges, a GPU erősen ajánlott a gyakorlati inferencia sebesség és a valós idejű interaktív szegmentálás érdekében.
Igen – a MIB SAM-2 integráció támogatja a 3D szegmentálást maginterpolációval a szeletek között a térfogati elemzéshez.
AxonDeepSeg
| Fejlesztő | NeuroPoly Lab a Polytechnique Montréal és az Université de Montréal intézményeiben |
| Támogatott platformok |
|
| Nyelv | Angol dokumentáció; nyílt forráskódú eszköz világszerte használva |
| Árazás | Ingyenes és nyílt forráskódú |
Áttekintés
Az AxonDeepSeg egy mesterséges intelligencia alapú eszköz az axonok és myelin automatikus szegmentálására mikroszkópos képeken. Konvolúciós neurális hálózatokat használva pontos háromosztályos szegmentációt nyújt (axon, myelin, háttér) több képalkotó modalitáson, beleértve a TEM, SEM és fényteres mikroszkópiát. Az axon átmérő, g-arány és myelin vastagság morfometriai mérések automatizálásával az AxonDeepSeg felgyorsítja a kvantitatív elemzést az idegtudományi kutatásokban, jelentősen csökkentve a manuális annotáció idejét és javítva az ismételhetőséget.
Főbb jellemzők
Kész modellek, optimalizálva TEM, SEM és fényteres mikroszkópos modalitásokhoz.
Pontosan osztályozza az axon, myelin és háttér területeket a mikroszkópos képeken.
Automatikusan számolja az axon átmérőt, g-arányt, myelin vastagságot és sűrűségi mutatókat.
A Napari GUI integráció lehetővé teszi a szegmentációs maszkok manuális finomítását a nagyobb pontosság érdekében.
Zökkenőmentesen integrálható egyedi folyamatokba nagy volumenű idegszövet elemzéshez.
Átfogó teszt szkriptek biztosítják az ismételhetőséget és a megbízható szegmentációs eredményeket.
Műszaki részletek
A NeuroPoly Lab által fejlesztett AxonDeepSeg mélytanulást alkalmaz a nagy pontosságú szegmentáció érdekében idegtudományi alkalmazásokban. Előre betanított modellek érhetők el különböző mikroszkópos modalitásokhoz, biztosítva a sokoldalúságot a képalkotó technikák között. Az eszköz integrálódik a Napari felülettel, amely lehetővé teszi a szegmentációs maszkok interaktív korrekcióját, javítva a pontosságot kihívást jelentő adathalmazokon. Az AxonDeepSeg kiszámítja a kulcsfontosságú morfometriai mutatókat, támogatva a nagy áteresztőképességű idegszöveti szerkezet- és patológia vizsgálatokat. Python alapú keretrendszere lehetővé teszi az egyedi folyamatokba való integrációt az axon és myelin morfológia nagyszabású elemzéséhez.
Letöltés vagy hozzáférés
Telepítés és beállítás
Győződjön meg róla, hogy Python 3.8 vagy újabb verzió telepítve van, majd telepítse az AxonDeepSeg és Napari csomagokat pip segítségével:
pip install axondeepseg napariFuttassa a mellékelt teszt szkripteket, hogy megerősítse, minden komponens megfelelően telepítve és működőképes.
Importálja a mikroszkópos képeket (TEM, SEM vagy fényteres) a Napari-ba vagy Python környezetébe.
Válassza ki a megfelelő előre betanított modellt az adott képalkotó modalitáshoz, majd futtassa a szegmentációt az axon és myelin maszkok létrehozásához.
Automatikusan számolja a morfometriai méréseket, beleértve az axon átmérőt, g-arányt, sűrűséget és myelin vastagságot, majd exportálja az eredményeket CSV formátumban.
A Napari GUI segítségével manuálisan módosíthatja a szegmentációs maszkokat, egyesítheti vagy törölheti azokat a pontosság javítása érdekében.
Fontos megfontolások
- A teljesítmény csökkenhet új vagy nem betanított képalkotó modalitások esetén
- Manuális korrekciók szükségesek lehetnek bonyolult vagy kihívást jelentő területeken
- GPU ajánlott a nagy adathalmazok gyorsabb feldolgozásához; CPU feldolgozás is támogatott
Gyakran ismételt kérdések
Az AxonDeepSeg támogatja a TEM (transzmissziós elektronmikroszkópia), SEM (pásztázó elektronmikroszkópia) és fényteres mikroszkópiát, előre betanított modellekkel optimalizálva mindegyik modalitáshoz.
Igen, az AxonDeepSeg teljesen ingyenes és nyílt forráskódú, akadémiai és kereskedelmi felhasználásra egyaránt elérhető.
Igen, az AxonDeepSeg automatikusan kiszámítja az axon átmérőt, g-arányt, myelin vastagságot és sűrűségi mutatókat a szegmentált képek alapján.
GPU ajánlott a nagy adathalmazok gyorsabb szegmentálásához, de kisebb elemzésekhez CPU használata is támogatott.
Igen, a Napari GUI integráció lehetővé teszi a szegmentációs maszkok interaktív korrekcióját és finomítását a nagyobb pontosság érdekében kihívást jelentő területeken.
Ilastik
| Fejlesztő | Az Ilastik csapata az Európai Molekuláris Biológiai Laboratóriumban (EMBL) és kapcsolódó akadémiai partnerek |
| Támogatott platformok |
|
| Nyelv | Angol |
| Árazás | Ingyenes és nyílt forráskódú |
Áttekintés
Az Ilastik egy erőteljes, mesterséges intelligencián alapuló eszköz interaktív képszegmentációhoz, osztályozáshoz és mikroszkópos adatok elemzéséhez. Gépi tanulási technikák, például Random Forest osztályozók segítségével lehetővé teszi a kutatók számára a pixelek szegmentálását, objektumok osztályozását, sejtek követését időben, valamint sűrűségszámlálást 2D és 3D adathalmazokban. Intuitív felületével és valós idejű visszacsatolásával az Ilastik programozói ismeretek nélkül is elérhető, és széles körben használják sejtbiológiában, idegtudományban és biomedikai képalkotásban.
Főbb jellemzők
Valós idejű visszacsatolás, miközben képviselő régiókat jelöl meg az azonnali szegmentációs eredményekért.
A szegmentált struktúrák kategorizálása morfológiai és intenzitásbeli jellemzők alapján.
Sejtek mozgásának és osztódásának követése 2D és 3D időbeli mikroszkópos kísérletekben.
Zsúfolt régiók mennyiségi meghatározása egyedi objektumok explicit szegmentációja nélkül.
Félautomatikus szegmentáció összetett 3D térfogatokhoz intuitív interakcióval.
Több kép automatikus feldolgozása parancssoros, grafikus felület nélküli módban.
Letöltés
Kezdő útmutató
Töltse le az Ilastik programot az operációs rendszerének megfelelően a hivatalos weboldalról. A csomag tartalmazza az összes szükséges Python függőséget, ezért kövesse a platformjára vonatkozó telepítési útmutatót.
Nyissa meg az Ilastikot, és válassza ki az elemzési munkafolyamatot: Pixelosztályozás, Objektumosztályozás, Követés vagy Sűrűségszámlálás. Töltse be a képadathalmazt, amely tartalmazhat többcsatornás, 3D vagy időbeli képeket.
Jelöljön meg néhány reprezentatív pixelt vagy objektumot a képeken. Az Ilastik Random Forest osztályozója ezekből az annotációkból tanul, és automatikusan előrejelzi a címkéket az egész adathalmazon.
Alkalmazza a betanított modellt az egész adathalmaz szegmentálására vagy osztályozására. Exportálja az eredményeket címkézett képek, valószínűségi térképek vagy mennyiségi táblázatok formájában további elemzéshez és megjelenítéshez.
Használja az Ilastik grafikus felület nélküli módját több kép automatikus feldolgozásához manuális beavatkozás nélkül, ami ideális nagyszabású elemzési folyamatokhoz.
Korlátozások és megfontolások
- Az interaktív címkézés időigényes lehet nagyon nagy adathalmazok esetén
- A pontosság a felhasználói annotációk minőségétől és reprezentativitásától függ
- Memóriaigény — nagyon nagy felbontású vagy több gigabájtos adathalmazok jelentős RAM-ot igényelhetnek
- Összetett adatok — a Random Forest osztályozók alulteljesíthetnek a mély neurális hálózatokhoz képest nagyon változékony vagy összetett képi adatok esetén
Gyakran Ismételt Kérdések
Igen, az Ilastik teljes mértékben támogatja a 3D térfogatok és időbeli kísérletek szegmentációját, követését és mennyiségi elemzését több időpontban.
Igen, az Ilastik teljesen ingyenes és nyílt forráskódú, minden felhasználó számára elérhető licenckorlátozások nélkül.
Nem, az Ilastik intuitív grafikus felületet biztosít valós idejű visszacsatolással, így programozói ismeretek nélkül is használható. Haladó felhasználók parancssoros kötegelt feldolgozást is végezhetnek.
Igen, a dedikált követési munkafolyamat lehetővé teszi a sejtek mozgásának és osztódásának elemzését 2D és 3D időbeli adathalmazokban automatikus leszármazási követéssel.
A szegmentációs eredmények exportálhatók címkézett képek, valószínűségi térképek vagy mennyiségi táblázatok formájában, lehetővé téve a zökkenőmentes integrációt további elemző eszközökkel és megjelenítő szoftverekkel.
Ezek az eszközök kezdőtől a szakértői szintig terjednek. Sok ingyenes és nyílt forráskódú, elősegítve az ismételhető és megosztható MI munkafolyamatokat a kutatói közösségben.
Kihívások és jövőbeli irányok
Jelenlegi kihívások
Megjelenő trendek
Vision alapmodellek
A következő generációs MI rendszerek ígérete, hogy csökkentik a feladatspecifikus tanítás szükségességét.
- Olyan modellek, mint a SAM és CLIP-alapú rendszerek
- Egy MI sok mikroszkópos feladatot kezel
- Gyorsabb bevezetés és adaptáció
MI által támogatott mikroszkópok
Teljesen autonóm és intelligens mikroszkópos rendszerek válnak valósággá.
- Természetes nyelvű vezérlés nagy nyelvi modelleken (LLM) keresztül
- Teljesen automatizált visszacsatolási hurkok
- Demokratizálja a fejlett mikroszkópia elérését
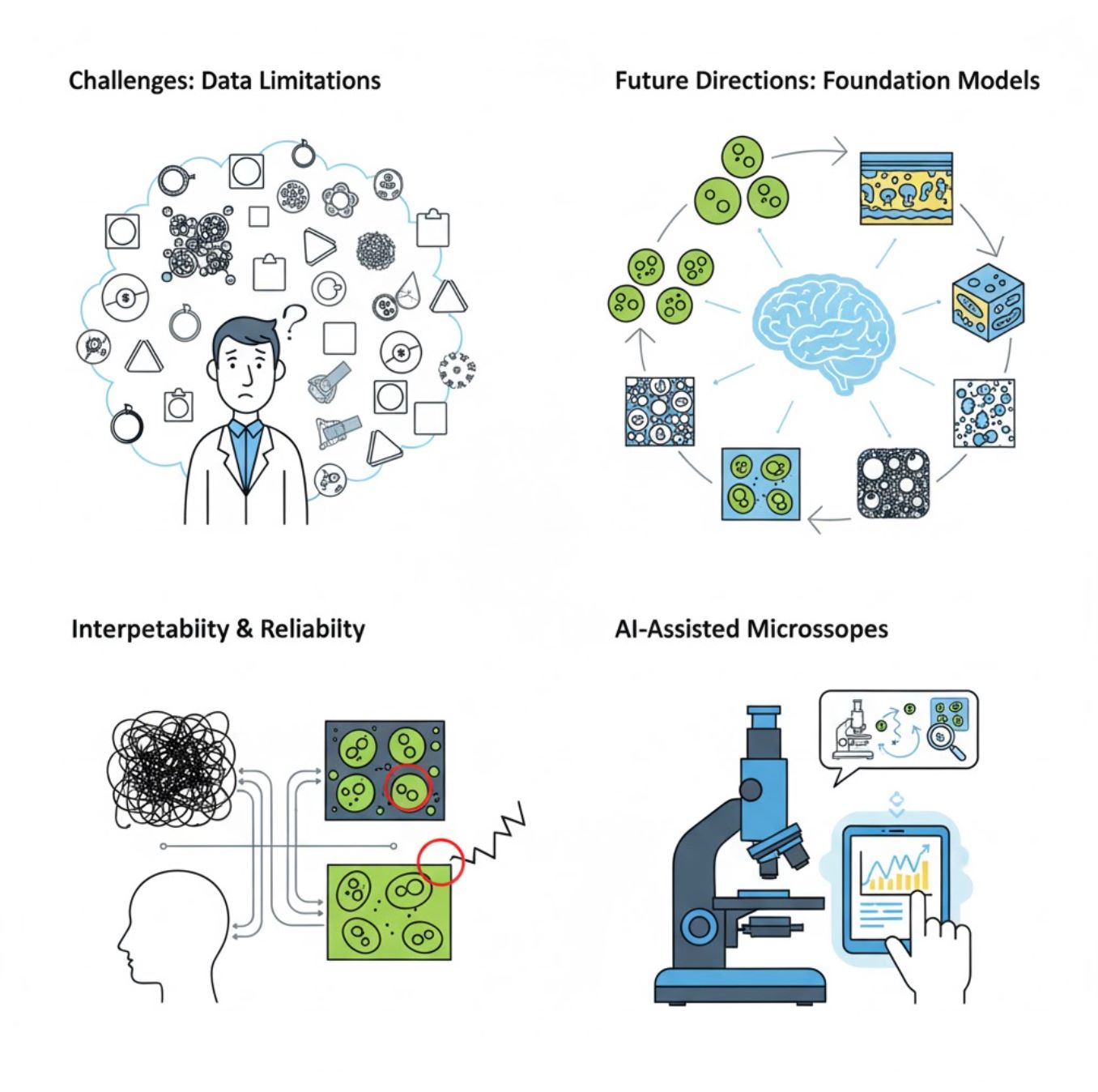
Főbb tanulságok
- Az MI gyorsan átalakítja a mikroszkópos képfeldolgozást jobb pontossággal és automatizálással
- A mélytanulás felülmúlja a hagyományos gépi tanulást összetett, változékony mikroszkópos képeken
- A CNN-ek automatikusan tanulják a hierarchikus jellemzőket nyers pixelekből a robusztus elemzéshez
- Fontos alkalmazások: szegmentáció, osztályozás, követés, zajcsökkentés és automatikus képrögzítés
- A siker a minőségi adatoktól és a szakértői validációtól függ
- A vision alapmodellek és az MI által támogatott mikroszkópok a jövőt képviselik
A folyamatos fejlődés és a közösségi erőfeszítések (nyílt forráskódú eszközök, megosztott adathalmazok) révén az MI egyre inkább a mikroszkóp „szemének” központi részévé válik, segítve a tudósokat a láthatatlan felfedezésében.







Még nincsenek kommentek. Légy te az első!