AI dalam Pemrosesan Gambar Mikroskop
AI merevolusi pemrosesan gambar mikroskop dengan kemampuan kuat seperti segmentasi presisi, pengurangan noise, super-resolusi, dan akuisisi gambar otomatis. Artikel ini menyoroti alat AI penting dan tren baru dalam penelitian ilmiah.
Teknik AI merevolusi mikroskopi dengan mengoptimalkan akuisisi gambar dan mengotomatisasi analisis. Dalam mikroskop pintar modern, modul AI dapat menyesuaikan parameter pencitraan secara langsung (misalnya, fokus, pencahayaan) untuk meminimalkan fotobleaching dan meningkatkan sinyal. Sementara itu, algoritma pembelajaran mendalam dapat menyaring data gambar kompleks untuk mengekstrak wawasan biologis tersembunyi dan bahkan menghubungkan gambar dengan data lain (misalnya, genomik).
Metode AI: Pembelajaran Mesin vs Pembelajaran Mendalam
Metode AI berkisar dari pembelajaran mesin (ML) klasik hingga pembelajaran mendalam (DL) modern. Setiap pendekatan memiliki keunggulan dan keterbatasan tersendiri:
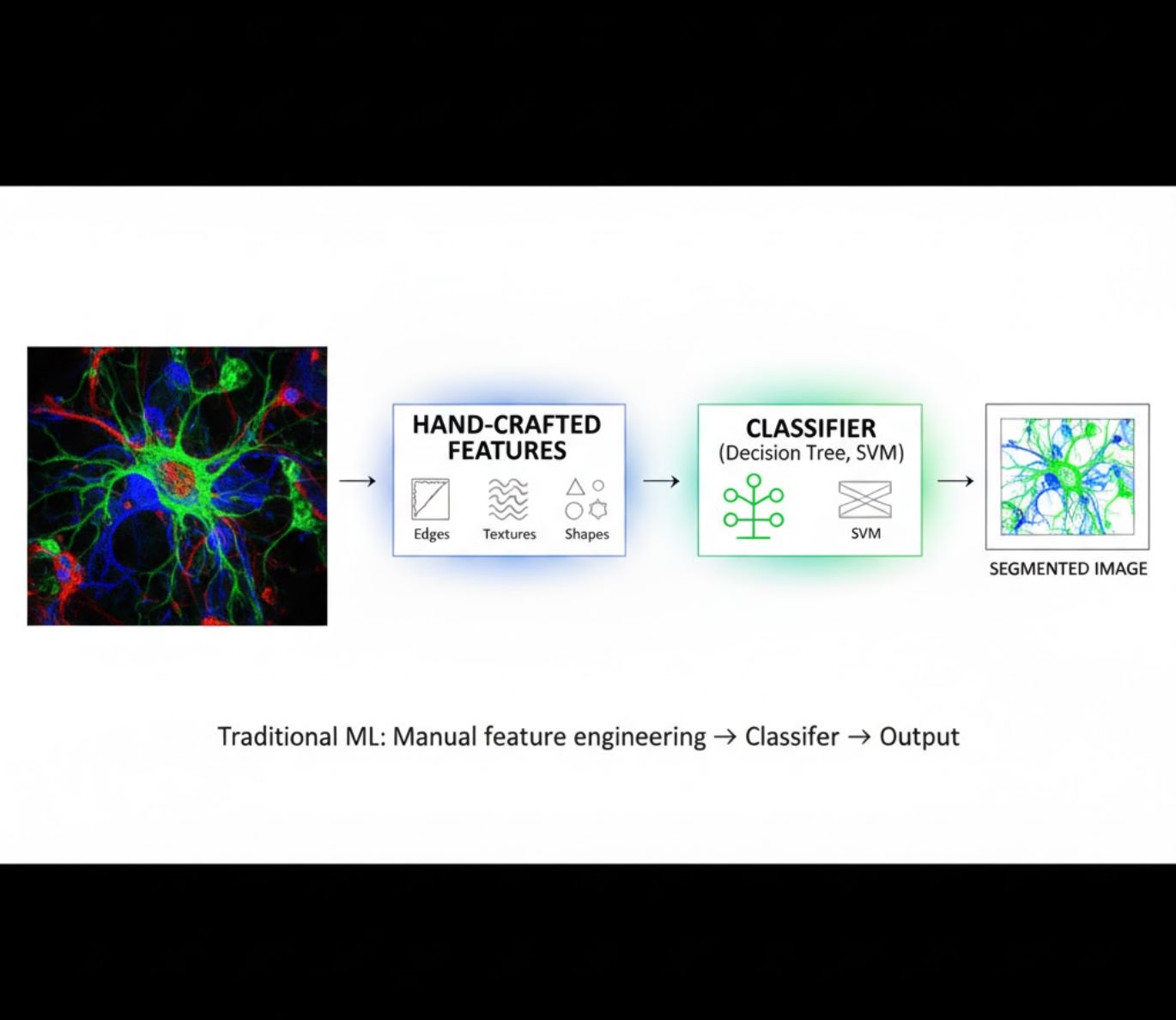
Fitur Buatan Tangan
- Peneliti secara manual membuat fitur gambar (tepi, tekstur, bentuk)
- Fitur diberikan ke pengklasifikasi (pohon keputusan, SVM)
- Cepat untuk dilatih
- Kesulitan dengan gambar kompleks atau berisik
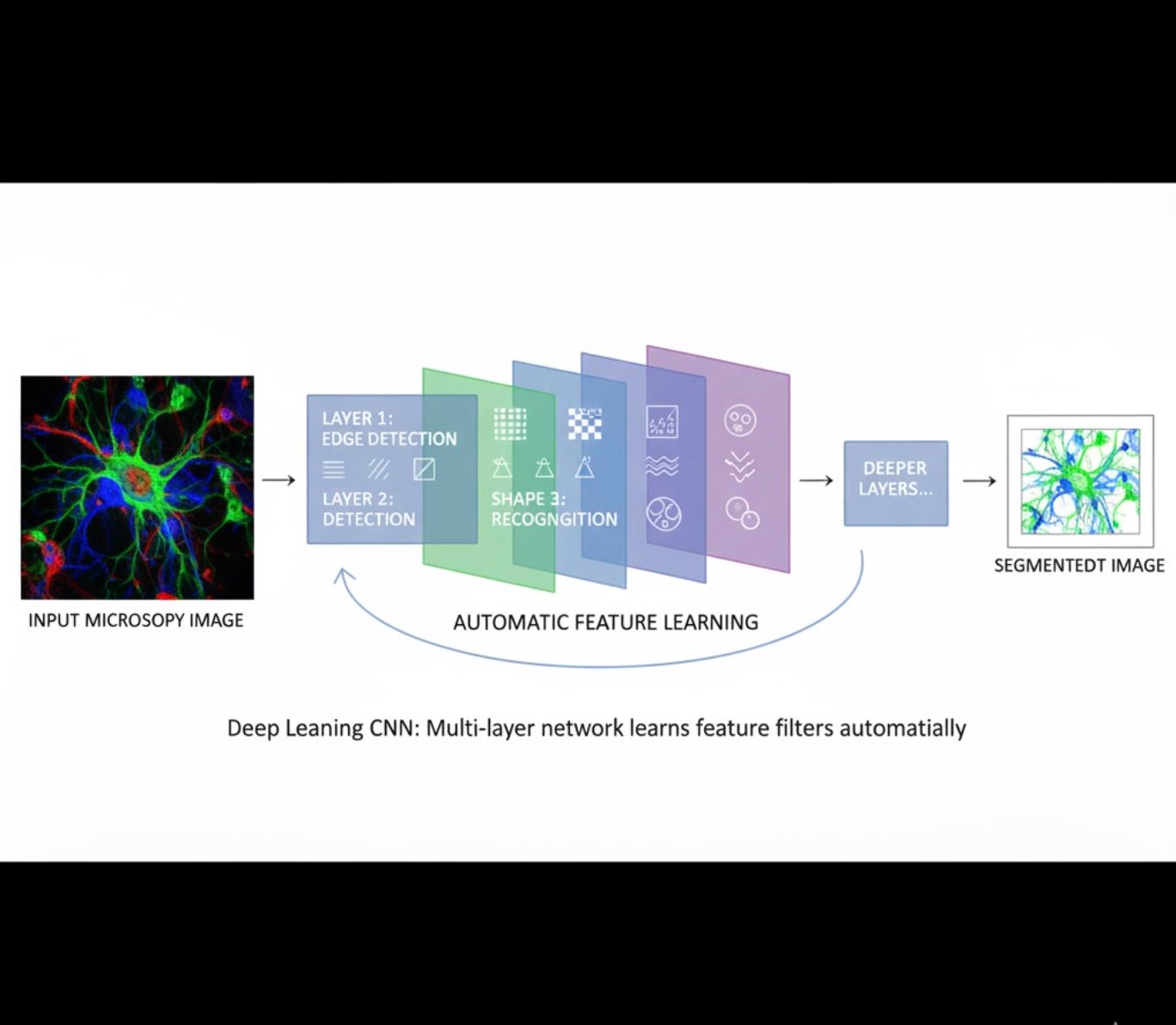
Pembelajaran Fitur Otomatis
- Jaringan saraf berlapis banyak (CNN) belajar fitur secara otomatis
- Pembelajaran ujung-ke-ujung dari piksel mentah
- Jauh lebih tangguh terhadap variasi
- Menangkap tekstur dan struktur rumit dengan andal
Cara kerja CNN: Jaringan saraf konvolusional menerapkan filter berturut-turut pada gambar mikroskopi, belajar mendeteksi pola sederhana (tepi) di lapisan awal dan struktur kompleks (bentuk sel, tekstur) di lapisan lebih dalam. Pembelajaran hierarkis ini membuat DL sangat tangguh meskipun profil intensitas sangat bervariasi.
Perbandingan Visual: Jalur ML vs DL
Aplikasi AI Utama dalam Mikroskopi
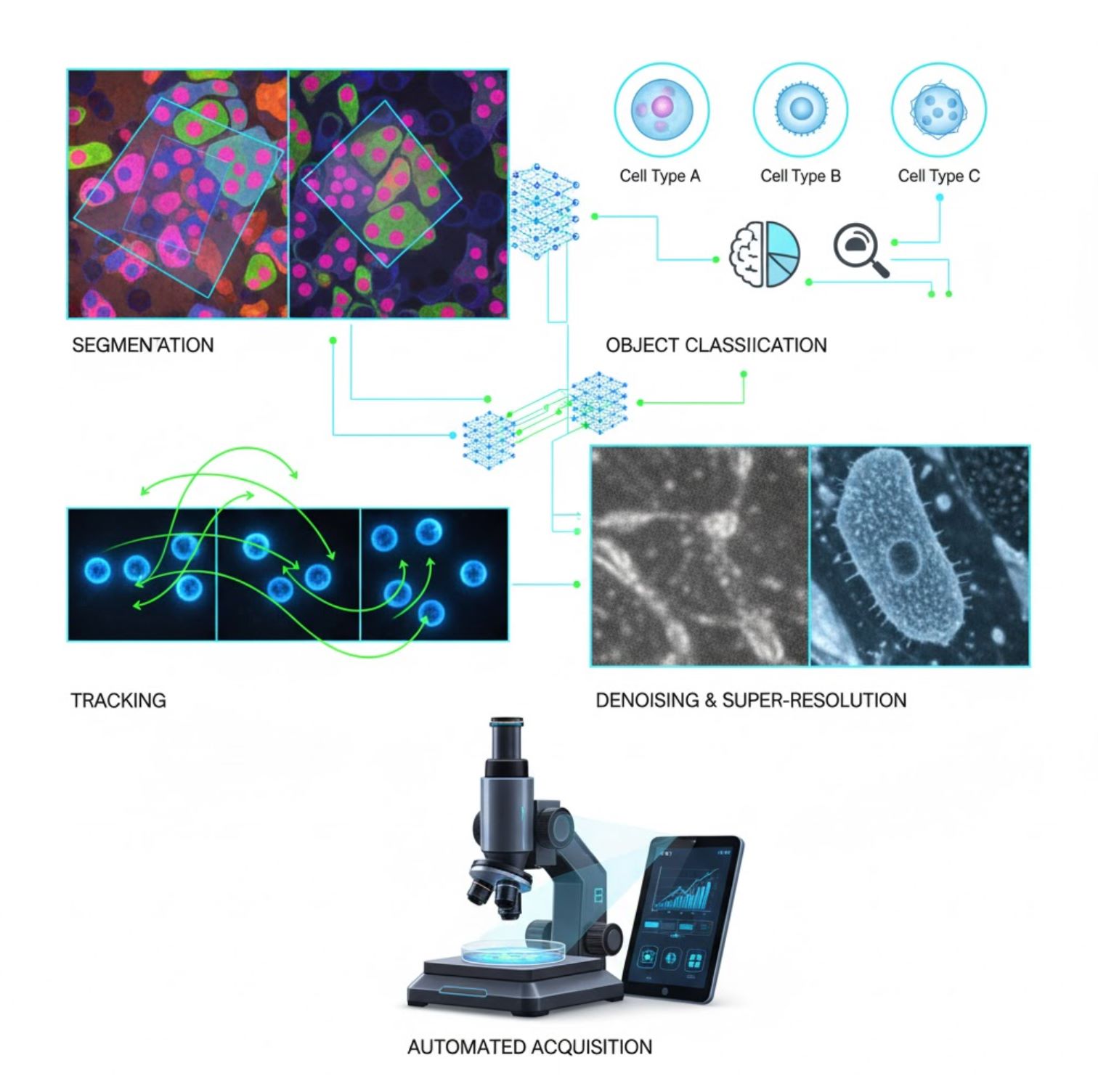
AI kini tertanam dalam banyak tugas pemrosesan gambar di seluruh alur kerja mikroskopi:
Segmentasi
Membagi gambar menjadi wilayah (misalnya, mengidentifikasi setiap sel atau inti). Jaringan dalam seperti U-Net unggul dalam tugas ini.
- Segmentasi semantik: Label kelas per piksel
- Segmentasi instansi: Memisahkan objek individual
- Akurasi tinggi pada gambar padat atau redup
- Model dasar visi (misalnya, μSAM) kini diadaptasi untuk mikroskopi
Klasifikasi Objek
Setelah segmentasi, AI mengklasifikasikan setiap objek dengan presisi tinggi.
- Identifikasi tipe sel
- Penentuan tahap mitosis
- Deteksi indikator patologi
- Membedakan fenotipe halus yang sulit dihitung secara manual
Pelacakan
Dalam mikroskopi time-lapse, AI melacak sel atau partikel antar frame dengan akurasi luar biasa.
- Pembelajaran mendalam secara dramatis meningkatkan akurasi pelacakan
- Memungkinkan analisis andal terhadap sel yang bergerak
- Menangkap proses biologis dinamis
Penghilangan Noise & Super-Resolusi
Model AI meningkatkan kualitas gambar dengan menghilangkan noise dan blur.
- Model mendalam berbasis fisika mempelajari optik mikroskop
- Merekonstruksi gambar lebih tajam tanpa artefak
- Resolusi lebih tinggi dengan artefak lebih sedikit dibanding metode tradisional
Akuisisi Otomatis
AI mengarahkan mikroskop secara real-time.
- Menganalisis gambar langsung untuk membuat keputusan cerdas
- Secara otomatis menyesuaikan fokus dan memindai area yang diminati
- Mengurangi fototoksisitas dan menghemat waktu
- Memungkinkan eksperimen pencitraan adaptif dan throughput tinggi
Alat AI Populer dalam Pemrosesan Gambar Mikroskop
<ITEM_DESCRIPTION>>>Sebuah ekosistem alat yang kaya mendukung kecerdasan buatan dalam mikroskopi. Para peneliti telah mengembangkan perangkat lunak umum maupun khusus, banyak di antaranya bersifat sumber terbuka:
Cellpose
| Pengembang | Carsen Stringer dan Marius Pachitariu (grup riset MouseLand) |
| Platform yang Didukung |
Memerlukan Python (instalasi pip/conda). GUI tersedia hanya di desktop. |
| Dukungan Bahasa | Dokumentasi berbahasa Inggris; digunakan secara global di laboratorium riset di seluruh dunia |
| Model Harga | Gratis dan open-source di bawah lisensi BSD-3-Clause |
Gambaran Umum
Cellpose adalah alat segmentasi canggih berbasis pembelajaran mendalam yang dirancang untuk gambar mikroskopi. Sebagai algoritma generalis, ia secara akurat memisahkan berbagai jenis sel (inti, sitoplasma, dll.) di berbagai modalitas pencitraan tanpa perlu pelatihan ulang model. Dengan kemampuan keterlibatan manusia, peneliti dapat menyempurnakan hasil, menyesuaikan model dengan data mereka, dan menerapkan sistem ini pada alur kerja pencitraan 2D maupun 3D.
Fitur Utama
Berfungsi langsung untuk berbagai jenis sel, pewarnaan, dan modalitas pencitraan tanpa pelatihan khusus.
Mendukung tumpukan 3D penuh menggunakan pendekatan "2.5D" yang memanfaatkan model 2D untuk data volumetrik.
Koreksi manual hasil segmentasi dan pelatihan ulang model pada data khusus Anda untuk akurasi yang lebih baik.
Akses melalui API Python, antarmuka baris perintah, atau antarmuka pengguna grafis untuk alur kerja yang fleksibel.
Kemampuan denoising, deblurring, dan upsampling untuk meningkatkan kualitas gambar sebelum segmentasi.
Unduh atau Akses
Latar Belakang Teknis
Cellpose diperkenalkan dalam studi penting oleh Stringer, Wang, Michaelos, dan Pachitariu, dilatih pada dataset besar dan sangat beragam yang berisi lebih dari 70.000 objek yang telah disegmentasi. Keanekaragaman ini memungkinkan model untuk menggeneralisasi berbagai bentuk, ukuran sel, dan pengaturan mikroskopi, secara signifikan mengurangi kebutuhan pelatihan khusus dalam sebagian besar kasus penggunaan. Untuk data 3D, Cellpose dengan cerdik menggunakan kembali model 2D-nya dalam cara "2.5D", menghindari kebutuhan data pelatihan berlabel 3D penuh namun tetap memberikan segmentasi volumetrik. Cellpose 2.0 memperkenalkan pelatihan ulang dengan keterlibatan manusia, memungkinkan pengguna mengoreksi prediksi secara manual dan melatih ulang pada gambar mereka sendiri untuk peningkatan performa pada dataset spesifik.
Instalasi & Pengaturan
Siapkan lingkungan Python menggunakan conda:
conda create -n cellpose python=3.10Aktifkan lingkungan dan instal Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeMemulai
Mode GUI
- Luncurkan GUI dengan menjalankan:
python -m cellpose - Seret dan lepas file gambar (
.tif,.png, dll.) ke dalam antarmuka - Pilih tipe model (misalnya, "cyto" untuk sitoplasma atau "nuclei" untuk inti sel)
- Atur perkiraan diameter sel atau biarkan Cellpose mengkalibrasi otomatis
- Klik untuk memulai segmentasi dan lihat hasilnya
Mode API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Sempurnakan & Latih Ulang
- Setelah menghasilkan masker, koreksi segmentasi di GUI dengan menggabungkan atau menghapus masker secara manual
- Gunakan fungsi pelatihan bawaan untuk melatih ulang pada contoh yang telah dikoreksi
- Performa model meningkat pada dataset spesifik Anda
Proses Data 3D
- Muat TIFF multi-Z atau tumpukan volumetrik
- Gunakan flag
--Zstackdi GUI atau API untuk memproses sebagai 3D - Opsional: sempurnakan aliran 3D melalui pelicinan atau parameter khusus untuk segmentasi lebih baik
Keterbatasan & Pertimbangan
- Perdagangan Generalitas Model: Meskipun model generalis bekerja luas, bentuk sel atau kondisi pencitraan yang sangat tidak biasa mungkin memerlukan pelatihan ulang.
- Upaya Anotasi: Pelatihan dengan keterlibatan manusia membutuhkan koreksi manual, yang bisa memakan waktu untuk dataset besar.
- Kompleksitas Instalasi: Instalasi GUI mungkin memerlukan penggunaan baris perintah, lingkungan conda, dan pengelolaan dependensi Python — tidak selalu mudah bagi non-programmer.
- Hanya Desktop: Cellpose dirancang untuk penggunaan desktop; tidak ada aplikasi asli Android atau iOS.
Pertanyaan yang Sering Diajukan
Tidak — Cellpose menyediakan model generalis pra-latih yang sering bekerja baik tanpa pelatihan ulang. Namun, untuk hasil optimal pada data khusus atau tidak biasa, Anda dapat menganotasi dan melatih ulang menggunakan fitur keterlibatan manusia.
Ya — Cellpose mendukung 3D dengan menggunakan kembali model 2D-nya (disebut "2.5D"), dan Anda dapat menjalankan tumpukan volumetrik melalui GUI atau API.
GPU sangat dianjurkan untuk inferensi dan pelatihan lebih cepat, terutama pada dataset besar atau 3D, tetapi Cellpose dapat berjalan pada mesin hanya CPU dengan performa lebih lambat.
Di GUI, atur diameter sel perkiraan secara manual atau biarkan Cellpose mengkalibrasi otomatis. Anda dapat menyempurnakan hasil dan melatih ulang jika segmentasi tidak optimal.
Ya — versi terbaru (Cellpose 3) menyertakan model restorasi gambar untuk denoising, deblurring, dan upsampling guna meningkatkan kualitas segmentasi sebelum pemrosesan.
StarDist
| Pengembang | Uwe Schmidt, Martin Weigert, Coleman Broaddus, dan Gene Myers |
| Platform yang Didukung |
|
| Dukungan Bahasa | Proyek sumber terbuka dengan dokumentasi dan komunitas terutama dalam bahasa Inggris |
| Model Harga | Gratis dan sumber terbuka. Berlisensi di bawah BSD-3-Clause |
Ikhtisar
StarDist adalah alat pembelajaran mendalam untuk segmentasi instansi dalam gambar mikroskopi. Ia merepresentasikan setiap objek (seperti inti sel) sebagai poligon star-konveks di 2D atau polihedron di 3D, memungkinkan deteksi dan pemisahan objek yang padat atau tumpang tindih secara akurat. Dengan arsitektur yang kuat, StarDist banyak digunakan untuk segmentasi otomatis sel dan inti dalam mikroskopi fluoresensi, histopatologi, dan aplikasi analisis bioimage lainnya.
Fitur Utama
Segmentasi instansi sangat akurat menggunakan poligon star-konveks (2D) dan polihedron (3D) untuk deteksi objek yang andal.
Model khusus untuk gambar 2D dan data volumetrik 3D untuk analisis mikroskopi yang komprehensif.
Model siap pakai untuk inti fluoresensi, histologi bertanda H&E, dan skenario pencitraan umum lainnya.
Mengklasifikasikan objek yang terdeteksi ke dalam kelas berbeda (misalnya tipe sel berbeda) dalam satu proses segmentasi.
Integrasi mulus dengan ImageJ/Fiji, QuPath, dan napari untuk alur kerja berbasis GUI yang mudah diakses.
Evaluasi segmentasi instansi yang komprehensif termasuk presisi, recall, skor F1, dan kualitas panoptik.
Latar Belakang Teknis
Awalnya diperkenalkan dalam makalah MICCAI 2018, inovasi inti StarDist adalah prediksi jarak radial sepanjang sinar tetap yang dikombinasikan dengan probabilitas objek untuk setiap piksel, memungkinkan rekonstruksi bentuk star-konveks yang akurat. Pendekatan ini dapat dengan andal memisahkan objek yang sangat berdekatan yang sulit dipisahkan menggunakan metode berbasis piksel atau bounding-box tradisional.
Perkembangan terbaru telah memperluas StarDist ke gambar histopatologi, memungkinkan tidak hanya segmentasi inti tetapi juga klasifikasi multi-kelas objek yang terdeteksi. Metode ini mencapai performa terbaik dalam tantangan seperti CoNIC (Colon Nuclei Identification and Counting).
Unduh atau Akses
Instalasi & Pengaturan
Pasang TensorFlow (versi 1.x atau 2.x) sebagai prasyarat untuk StarDist.
Gunakan pip untuk memasang paket Python StarDist:
pip install stardistUntuk napari:
pip install stardist-napariUntuk QuPath: Pasang ekstensi StarDist dengan menyeret file .jar ke dalam QuPath.
Untuk ImageJ/Fiji: Gunakan manajer plugin bawaan atau instalasi manual melalui menu plugins.
Menjalankan Segmentasi
Muat model pra-latih, normalisasi gambar Anda, dan jalankan prediksi:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Buka gambar Anda di napari, pilih plugin StarDist, pilih model pra-latih atau kustom, dan jalankan prediksi langsung dari GUI.
Gunakan plugin StarDist dari menu Plugins untuk menerapkan model pada tumpukan gambar Anda dengan antarmuka yang intuitif.
Setelah memasang ekstensi, jalankan deteksi StarDist melalui konsol skrip atau antarmuka grafis QuPath untuk analisis histopatologi.
Pelatihan & Penyempurnaan
Buat gambar label ground-truth di mana setiap objek diberi label unik. Gunakan alat anotasi seperti LabKit, QuPath, atau Fiji untuk menyiapkan dataset Anda.
Gunakan API Python StarDist untuk melatih model baru atau menyempurnakan model yang ada dengan data anotasi kustom Anda.
Opsi Pasca-Pemrosesan
- Terapkan non-maximum suppression (NMS) untuk menghilangkan bentuk kandidat yang berlebihan
- Gunakan StarDist OPP (Object Post-Processing) untuk menggabungkan masker untuk bentuk non-star-konveks
Batasan & Pertimbangan
- Asumsi star-konveks mungkin tidak memodelkan bentuk yang sangat non-konveks atau sangat tidak beraturan dengan sempurna
- Kompleksitas instalasi: instalasi khusus memerlukan compiler C++ yang kompatibel untuk membangun ekstensi
- Percepatan GPU bergantung pada versi TensorFlow, CUDA, dan cuDNN yang kompatibel
- Beberapa pengguna melaporkan masalah menjalankan plugin ImageJ karena konfigurasi Java
Pertanyaan yang Sering Diajukan
StarDist bekerja dengan berbagai jenis gambar termasuk fluoresensi, brightfield, dan histopatologi (misalnya H&E), berkat model pra-latih yang fleksibel dan kemampuan adaptasi ke berbagai modalitas pencitraan.
Bisa — StarDist mendukung segmentasi instansi 3D menggunakan polihedron star-konveks untuk data volumetrik, memperluas kemampuan 2D ke analisis 3D penuh.
Tidak selalu. Model pra-latih tersedia dan sering bekerja baik langsung. Namun, untuk data khusus atau baru, memberi anotasi dan melatih model kustom secara signifikan meningkatkan akurasi.
StarDist terintegrasi dengan napari, ImageJ/Fiji, dan QuPath, memungkinkan Anda menjalankan segmentasi dari GUI tanpa coding. Ia juga mendukung penggunaan API Python langsung untuk alur kerja lanjutan.
StarDist menyediakan fungsi bawaan untuk menghitung metrik segmentasi instansi umum termasuk presisi, recall, skor F1, dan kualitas panoptik untuk menilai performa segmentasi.
SAM
Informasi Aplikasi
| Pengembang | Meta AI Research (FAIR) |
| Perangkat yang Didukung |
|
| Bahasa & Ketersediaan | Model dasar open-source tersedia secara global; dokumentasi dalam bahasa Inggris |
| Harga | Gratis — open-source di bawah lisensi Meta melalui GitHub dan integrasi MIB |
Gambaran Umum
SAM (Segment Anything Model) adalah model dasar AI yang kuat yang dibuat oleh Meta yang memungkinkan segmentasi interaktif dan otomatis dari hampir semua objek dalam gambar. Dengan menggunakan prompt seperti titik, kotak pembatas, atau masker kasar, SAM menghasilkan masker segmentasi tanpa memerlukan pelatihan ulang khusus untuk tugas tertentu. Dalam penelitian mikroskopi, fleksibilitas SAM telah diadaptasi untuk segmentasi sel, deteksi organel, dan analisis histopatologi, menawarkan solusi yang dapat diskalakan bagi peneliti yang membutuhkan alat segmentasi serbaguna yang dapat dipicu dengan prompt.
Pengenalan Detail
Awalnya dilatih oleh Meta pada lebih dari 1 miliar masker di lebih dari 11 juta gambar, SAM dirancang sebagai model dasar yang dapat dipicu dengan prompt untuk segmentasi dengan performa "zero-shot" pada domain baru. Dalam penelitian citra medis, SAM telah dievaluasi untuk segmentasi patologi whole-slide, deteksi tumor, dan identifikasi inti sel. Namun, performanya pada instance yang sangat padat—seperti inti sel—bervariasi: bahkan dengan prompt yang banyak (misalnya, 20 klik atau kotak), segmentasi zero-shot dapat mengalami kesulitan pada gambar mikroskopi yang kompleks.
Untuk mengatasi keterbatasan ini, adaptasi domain-spesifik telah muncul:
- SAMCell — Fine-tuning pada dataset mikroskopi besar untuk segmentasi zero-shot yang kuat di berbagai tipe sel tanpa pelatihan ulang per eksperimen
- μSAM — Dilatih ulang pada lebih dari 17.000 gambar mikroskopi yang dianotasi secara manual untuk meningkatkan akurasi pada struktur seluler kecil
Fitur Utama
Interaksi fleksibel menggunakan titik, kotak, dan masker untuk kontrol yang presisi.
Melakukan segmentasi tanpa fine-tuning pada domain gambar baru.
Dapat diadaptasi untuk mikroskopi dan histopatologi melalui pelatihan ulang few-shot atau berbasis prompt.
Tersedia di Microscopy Image Browser (MIB) dengan dukungan segmentasi 3D dan interpolasi.
IDCC-SAM memungkinkan penghitungan sel otomatis dalam imunositokimia tanpa anotasi manual.
Unduh atau Akses
Panduan Pengguna
- Buka Microscopy Image Browser dan navigasikan ke panel segmentasi SAM
- Konfigurasikan interpreter Python dan pilih antara model SAM-1 atau SAM-2
- Untuk akselerasi GPU, pilih "cuda" pada lingkungan eksekusi (disarankan untuk performa optimal)
- Prompt titik: Klik pada objek untuk menentukan seed positif; gunakan Shift + klik untuk memperluas dan Ctrl + klik untuk seed negatif
- Tumpukan 3D: Gunakan mode Interaktif 3D—klik pada satu irisan, gulir shift, dan interpolasi seed di seluruh irisan
- Mode penyesuaian: Ganti, tambah, kurangi masker, atau buat lapisan baru sesuai kebutuhan
- Gunakan opsi "Automatic everything" di panel SAM-2 MIB untuk segmentasi semua objek yang terlihat di suatu area
- Tinjau dan perbaiki masker setelah segmentasi sesuai kebutuhan
- Gunakan pipeline fine-tuning berbasis prompt (misalnya, "All-in-SAM") untuk menghasilkan anotasi tingkat piksel dari prompt pengguna yang jarang
- Untuk penghitungan sel, terapkan IDCC-SAM, yang menggunakan SAM dalam pipeline zero-shot dengan pemrosesan pasca
- Untuk segmentasi sel dengan akurasi tinggi, gunakan SAMCell, yang telah di-fine-tune pada gambar sel mikroskopi
Keterbatasan & Pertimbangan
- Performa zero-shot tidak konsisten pada struktur padat atau tumpang tindih tanpa penyetelan domain
- Kualitas segmentasi sangat bergantung pada desain dan strategi prompt
- GPU sangat disarankan; inference menggunakan CPU sangat lambat
- Kesulitan pada gambar whole-slide resolusi sangat tinggi dan struktur jaringan multi-skala
- Fine-tuning atau adaptasi SAM untuk mikroskopi mungkin memerlukan keahlian pembelajaran mesin
Pertanyaan yang Sering Diajukan
Ya—melalui adaptasi seperti SAMCell, yang melakukan fine-tuning SAM pada dataset mikroskopi khusus untuk tugas segmentasi sel.
Tidak selalu. Dengan IDCC-SAM, Anda dapat melakukan penghitungan sel zero-shot tanpa anotasi manual.
Gunakan fine-tuning berbasis prompt (misalnya, "All-in-SAM") atau versi mikroskopi yang sudah dilatih sebelumnya seperti μSAM, yang dilatih pada lebih dari 17.000 gambar mikroskopi yang dianotasi.
Meski memungkinkan menggunakan CPU, GPU sangat disarankan untuk kecepatan inference yang praktis dan segmentasi interaktif waktu nyata.
Ya—integrasi SAM-2 di MIB mendukung segmentasi 3D dengan interpolasi seed di seluruh irisan untuk analisis volumetrik.
AxonDeepSeg
| Pengembang | NeuroPoly Lab di Polytechnique Montréal dan Université de Montréal |
| Platform yang Didukung |
|
| Bahasa | Dokumentasi berbahasa Inggris; alat sumber terbuka yang digunakan secara global |
| Harga | Gratis dan sumber terbuka |
Gambaran Umum
AxonDeepSeg adalah alat berbasis AI untuk segmentasi otomatis axon dan mielin dalam gambar mikroskopi. Dengan menggunakan jaringan saraf konvolusional, alat ini memberikan segmentasi tiga kelas yang akurat (axon, mielin, latar belakang) pada berbagai modalitas pencitraan termasuk TEM, SEM, dan mikroskopi bright-field. Dengan mengotomatisasi pengukuran morfometrik seperti diameter axon, rasio g, dan ketebalan mielin, AxonDeepSeg mempermudah analisis kuantitatif dalam penelitian neuroscience, secara signifikan mengurangi waktu anotasi manual dan meningkatkan reproduktifitas.
Fitur Utama
Model siap pakai yang dioptimalkan untuk modalitas mikroskopi TEM, SEM, dan bright-field.
Klasifikasi tepat untuk area axon, mielin, dan latar belakang dalam gambar mikroskopi.
Perhitungan otomatis diameter axon, rasio g, ketebalan mielin, dan metrik kepadatan.
Integrasi GUI Napari memungkinkan penyempurnaan manual masker segmentasi untuk akurasi lebih baik.
Terintegrasi mulus ke dalam pipeline kustom untuk analisis jaringan saraf berskala besar.
Skrip pengujian komprehensif memastikan reproduktifitas dan hasil segmentasi yang andal.
Detail Teknis
Dikembangkan oleh NeuroPoly Lab, AxonDeepSeg memanfaatkan pembelajaran mendalam untuk memberikan segmentasi presisi tinggi bagi aplikasi neuroscience. Model pra-latih tersedia untuk berbagai modalitas mikroskopi, menjamin fleksibilitas pada teknik pencitraan. Alat ini terintegrasi dengan Napari, memungkinkan koreksi interaktif masker segmentasi yang meningkatkan akurasi pada dataset yang menantang. AxonDeepSeg menghitung metrik morfometrik utama, mendukung studi throughput tinggi tentang struktur dan patologi jaringan saraf. Kerangka kerja berbasis Python memungkinkan integrasi ke pipeline kustom untuk analisis berskala besar morfologi axon dan mielin.
Unduh atau Akses
Instalasi & Pengaturan
Pastikan Python 3.8 atau versi lebih baru sudah terpasang, lalu instal AxonDeepSeg dan Napari menggunakan pip:
pip install axondeepseg napariJalankan skrip pengujian yang disediakan untuk memastikan semua komponen terpasang dan berfungsi dengan baik.
Impor gambar mikroskopi (TEM, SEM, atau bright-field) ke dalam Napari atau lingkungan Python Anda.
Pilih model pra-latih yang sesuai dengan modalitas pencitraan Anda dan jalankan segmentasi untuk menghasilkan masker axon dan mielin.
Hitung secara otomatis pengukuran morfometrik termasuk diameter axon, rasio g, kepadatan, dan ketebalan mielin, lalu ekspor hasil dalam format CSV.
Gunakan GUI Napari untuk menyesuaikan masker segmentasi secara manual jika diperlukan, menggabungkan atau menghapus masker demi akurasi yang lebih baik.
Pertimbangan Penting
- Performa dapat menurun pada modalitas pencitraan baru atau yang belum dilatih
- Koreksi manual mungkin diperlukan untuk area yang kompleks atau menantang
- GPU direkomendasikan untuk pemrosesan dataset besar yang lebih cepat; pemrosesan CPU juga didukung
Pertanyaan yang Sering Diajukan
AxonDeepSeg mendukung TEM (Transmission Electron Microscopy), SEM (Scanning Electron Microscopy), dan mikroskopi bright-field dengan model pra-latih yang dioptimalkan untuk setiap modalitas.
Ya, AxonDeepSeg sepenuhnya gratis dan sumber terbuka, tersedia untuk penggunaan akademik dan komersial.
Ya, AxonDeepSeg secara otomatis menghitung diameter axon, rasio g, ketebalan mielin, dan metrik kepadatan dari gambar yang sudah disegmentasi.
GPU direkomendasikan untuk segmentasi dataset besar agar lebih cepat, namun pemrosesan CPU juga didukung untuk analisis yang lebih kecil.
Ya, integrasi GUI Napari memungkinkan koreksi interaktif dan penyempurnaan masker segmentasi untuk akurasi lebih tinggi pada area yang menantang.
Ilastik
| Pengembang | Tim Ilastik di European Molecular Biology Laboratory (EMBL) dan mitra akademik terkait |
| Platform yang Didukung |
|
| Bahasa | Inggris |
| Harga | Gratis dan sumber terbuka |
Ikhtisar
Ilastik adalah alat bertenaga AI untuk segmentasi gambar interaktif, klasifikasi, dan analisis data mikroskopi. Dengan teknik pembelajaran mesin seperti pengklasifikasi Random Forest, alat ini memungkinkan peneliti untuk menyegmentasi piksel, mengklasifikasikan objek, melacak sel dari waktu ke waktu, dan melakukan penghitungan kepadatan pada dataset 2D dan 3D. Dengan antarmuka intuitif dan umpan balik waktu nyata, Ilastik dapat diakses oleh ilmuwan tanpa keahlian pemrograman dan banyak digunakan dalam biologi sel, ilmu saraf, dan pencitraan biomedis.
Fitur Utama
Umpan balik waktu nyata saat Anda memberi anotasi wilayah representatif untuk hasil segmentasi instan.
Mengategorikan struktur yang telah disegmentasi berdasarkan fitur morfologi dan intensitas.
Melacak pergerakan dan pembelahan sel dalam eksperimen mikroskopi time-lapse 2D dan 3D.
Mengukur wilayah padat tanpa segmentasi eksplisit objek individual.
Segmentasi semi-otomatis untuk volume 3D kompleks dengan interaksi yang intuitif.
Memproses banyak gambar secara otomatis menggunakan mode baris perintah tanpa antarmuka grafis.
Unduhan
Panduan Memulai
Unduh Ilastik untuk sistem operasi Anda dari situs resmi. Paket ini sudah termasuk semua dependensi Python yang diperlukan, jadi ikuti petunjuk instalasi sesuai platform Anda.
Buka Ilastik dan pilih alur kerja analisis Anda: Klasifikasi Piksel, Klasifikasi Objek, Pelacakan, atau Penghitungan Kepadatan. Muat dataset gambar Anda, yang dapat mencakup multi-saluran, 3D, atau gambar time-lapse.
Beri label beberapa piksel atau objek representatif dalam gambar Anda. Pengklasifikasi Random Forest Ilastik belajar dari anotasi ini dan secara otomatis memprediksi label di seluruh dataset Anda.
Terapkan model yang telah dilatih untuk menyegmentasi atau mengklasifikasikan seluruh dataset Anda. Ekspor hasil sebagai gambar berlabel, peta probabilitas, atau tabel kuantitatif untuk analisis dan visualisasi lanjutan.
Gunakan mode headless Ilastik untuk memproses banyak gambar secara otomatis tanpa intervensi manual, ideal untuk pipeline analisis skala besar.
Batasan & Pertimbangan
- Pelabelan interaktif dapat memakan waktu untuk dataset yang sangat besar
- Akurasi bergantung pada kualitas dan representativitas anotasi pengguna
- Kebutuhan memori — dataset beresolusi sangat tinggi atau multi-gigabyte mungkin memerlukan RAM yang besar
- Data kompleks — pengklasifikasi Random Forest mungkin kurang optimal dibandingkan jaringan saraf dalam pada data pencitraan yang sangat bervariasi atau kompleks
Pertanyaan yang Sering Diajukan
Ya, Ilastik sepenuhnya mendukung volume 3D dan eksperimen time-lapse untuk segmentasi, pelacakan, dan analisis kuantitatif di berbagai titik waktu.
Ya, Ilastik sepenuhnya gratis dan sumber terbuka, tersedia untuk semua pengguna tanpa batasan lisensi.
Tidak, Ilastik menyediakan antarmuka grafis yang intuitif dengan umpan balik waktu nyata, sehingga dapat diakses oleh peneliti tanpa keahlian pemrograman. Pengguna lanjutan juga dapat menggunakan pemrosesan batch melalui baris perintah.
Ya, alur kerja pelacakan khusus memungkinkan analisis pergerakan dan pembelahan sel pada dataset time-lapse 2D dan 3D dengan pelacakan garis keturunan otomatis.
Output segmentasi dapat diekspor sebagai gambar berlabel, peta probabilitas, atau tabel kuantitatif, memungkinkan integrasi mulus dengan alat analisis lanjutan dan perangkat lunak visualisasi.
Alat-alat ini mencakup tingkat pemula hingga ahli. Banyak yang gratis dan sumber terbuka, memudahkan alur kerja AI yang dapat direproduksi dan dibagikan di komunitas riset.
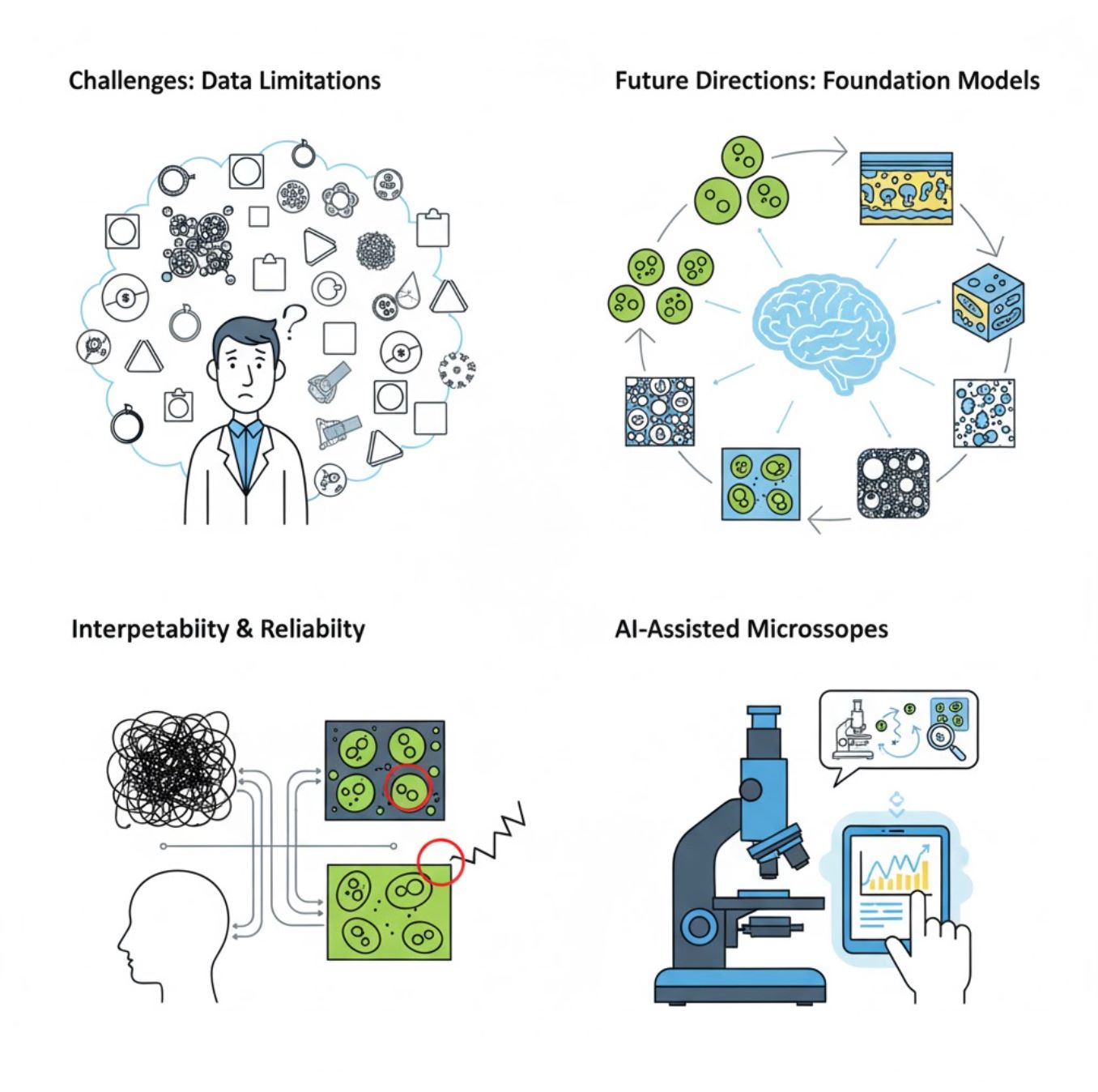
Tantangan dan Arah Masa Depan
Tantangan Saat Ini
Tren Baru
Model Dasar Visi
Sistem AI generasi berikutnya menjanjikan pengurangan kebutuhan pelatihan khusus tugas.
- Model seperti SAM dan sistem berbasis CLIP
- Satu AI menangani banyak tugas mikroskopi
- Penerapan dan adaptasi lebih cepat
Mikroskop Berbantuan AI
Sistem mikroskopi otonom dan cerdas sepenuhnya mulai menjadi kenyataan.
- Kontrol bahasa alami melalui LLM
- Loop umpan balik otomatis penuh
- Mendemokratisasi akses mikroskopi canggih
Poin Penting
- AI dengan cepat mengubah pemrosesan gambar mikroskop dengan akurasi dan otomatisasi yang lebih baik
- Pembelajaran mendalam mengungguli pembelajaran mesin tradisional pada gambar mikroskopi yang kompleks dan bervariasi
- CNN secara otomatis mempelajari fitur hierarkis dari piksel mentah untuk analisis yang tangguh
- Aplikasi utama meliputi segmentasi, klasifikasi, pelacakan, penghilangan noise, dan akuisisi otomatis
- Keberhasilan bergantung pada data berkualitas dan validasi cermat oleh ahli
- Model dasar visi dan mikroskop berbantuan AI mewakili masa depan bidang ini
Dengan kemajuan berkelanjutan dan upaya komunitas (alat sumber terbuka, dataset bersama), AI akan semakin menjadi bagian inti dari "mata" mikroskop, membantu ilmuwan melihat yang tak terlihat.







No comments yet. Be the first to comment!