Umělá inteligence v zpracování mikroskopických snímků
Umělá inteligence revolučně mění zpracování mikroskopických snímků díky výkonným schopnostem, jako je přesná segmentace, redukce šumu, superrozlišení a automatizované získávání snímků. Tento článek zdůrazňuje klíčové nástroje AI a nové trendy ve vědeckém výzkumu.
Techniky umělé inteligence revolučně mění mikroskopii tím, že optimalizují získávání snímků a automatizují analýzu. V moderních chytrých mikroskopech mohou AI moduly upravovat parametry snímání za běhu (např. zaostření, osvětlení), aby minimalizovaly fotoblení a zlepšily signál. Mezitím algoritmy hlubokého učení dokážou procházet složitá obrazová data, aby odhalily skryté biologické poznatky a dokonce propojily snímky s dalšími daty (např. genomikou).
Metody AI: strojové učení vs hluboké učení
Metody AI sahají od klasického strojového učení (ML) po moderní hluboké učení (DL). Každý přístup má své silné a slabé stránky:
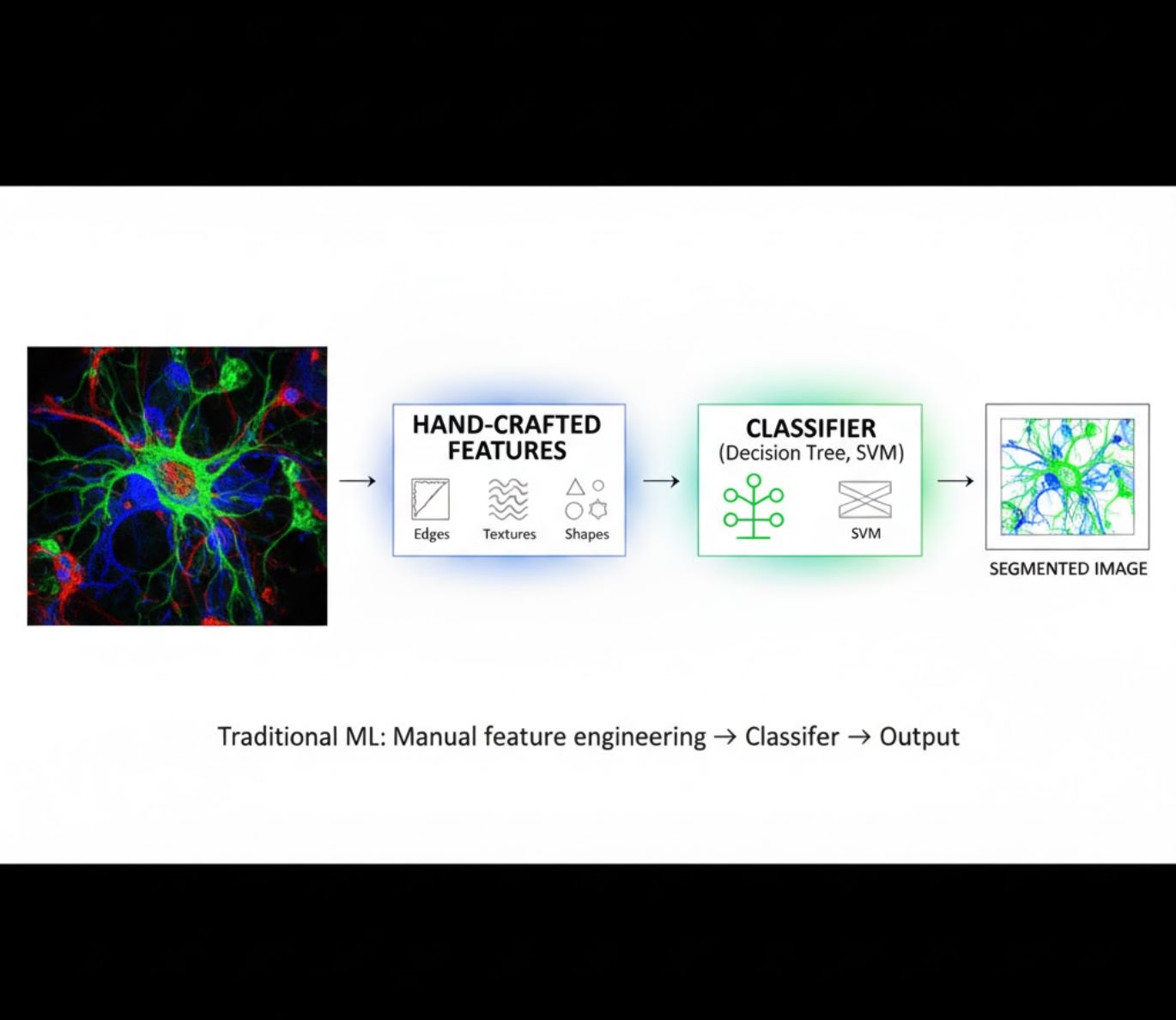
Ručně vytvořené rysy
- Výzkumníci ručně vytvářejí obrazové rysy (hrany, textury, tvary)
- Rysy jsou předávány klasifikátorům (rozhodovací stromy, SVM)
- Rychlé učení
- Obtížné u složitých nebo šumových snímků
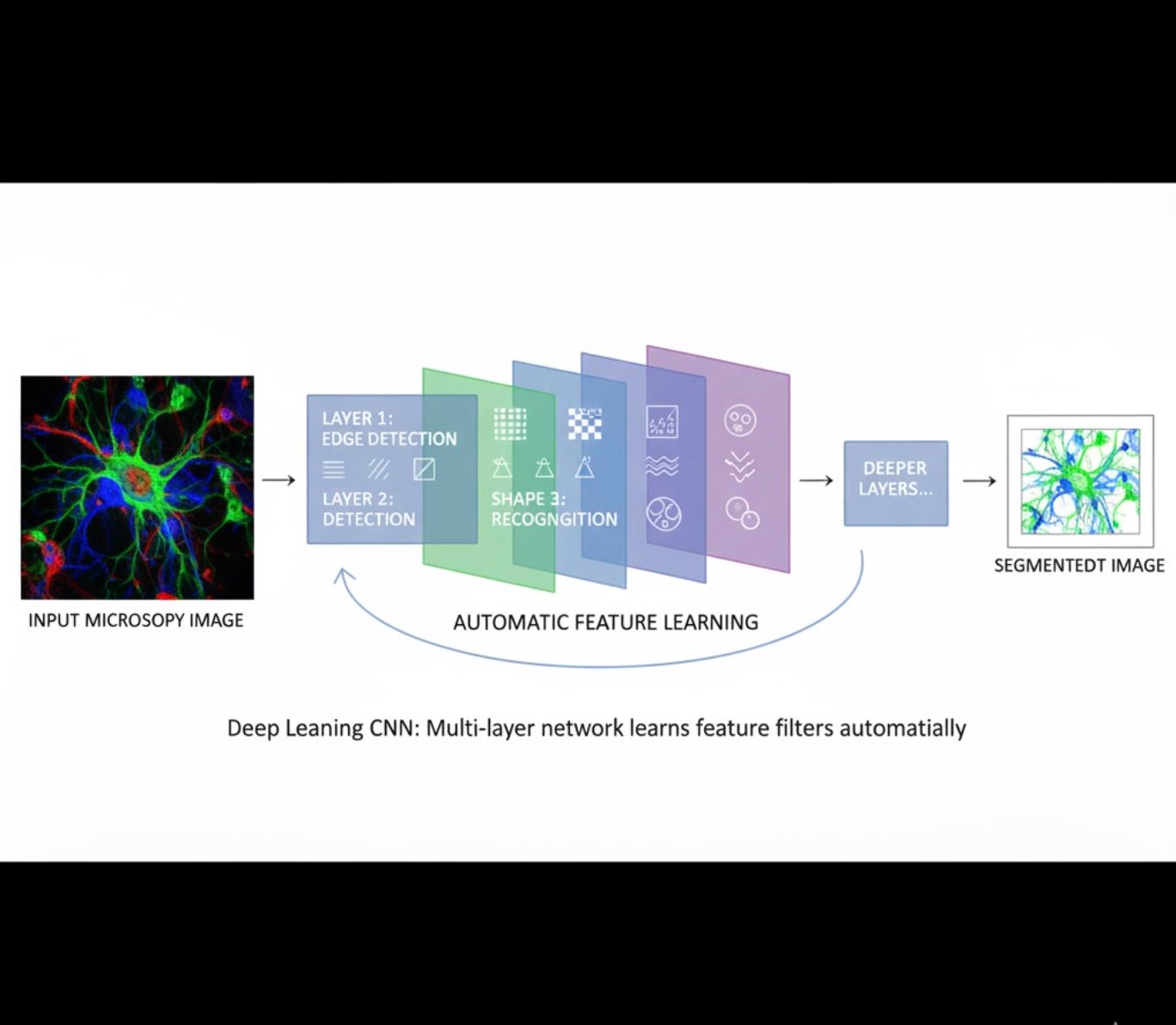
Automatické učení rysů
- Vícevrstvé neuronové sítě (CNN) se učí rysy automaticky
- Učení od surových pixelů end-to-end
- Výrazně odolnější vůči variacím
- Spolehlivě zachycuje složité textury a struktury
Jak CNN fungují: Konvoluční neuronové sítě aplikují postupné filtry na mikroskopické snímky, učí se detekovat jednoduché vzory (hrany) v raných vrstvách a složité struktury (tvary buněk, textury) v hlubších vrstvách. Toto hierarchické učení činí DL mimořádně odolným i při výrazných změnách intenzity.
Vizualní srovnání: ML vs DL pipeline
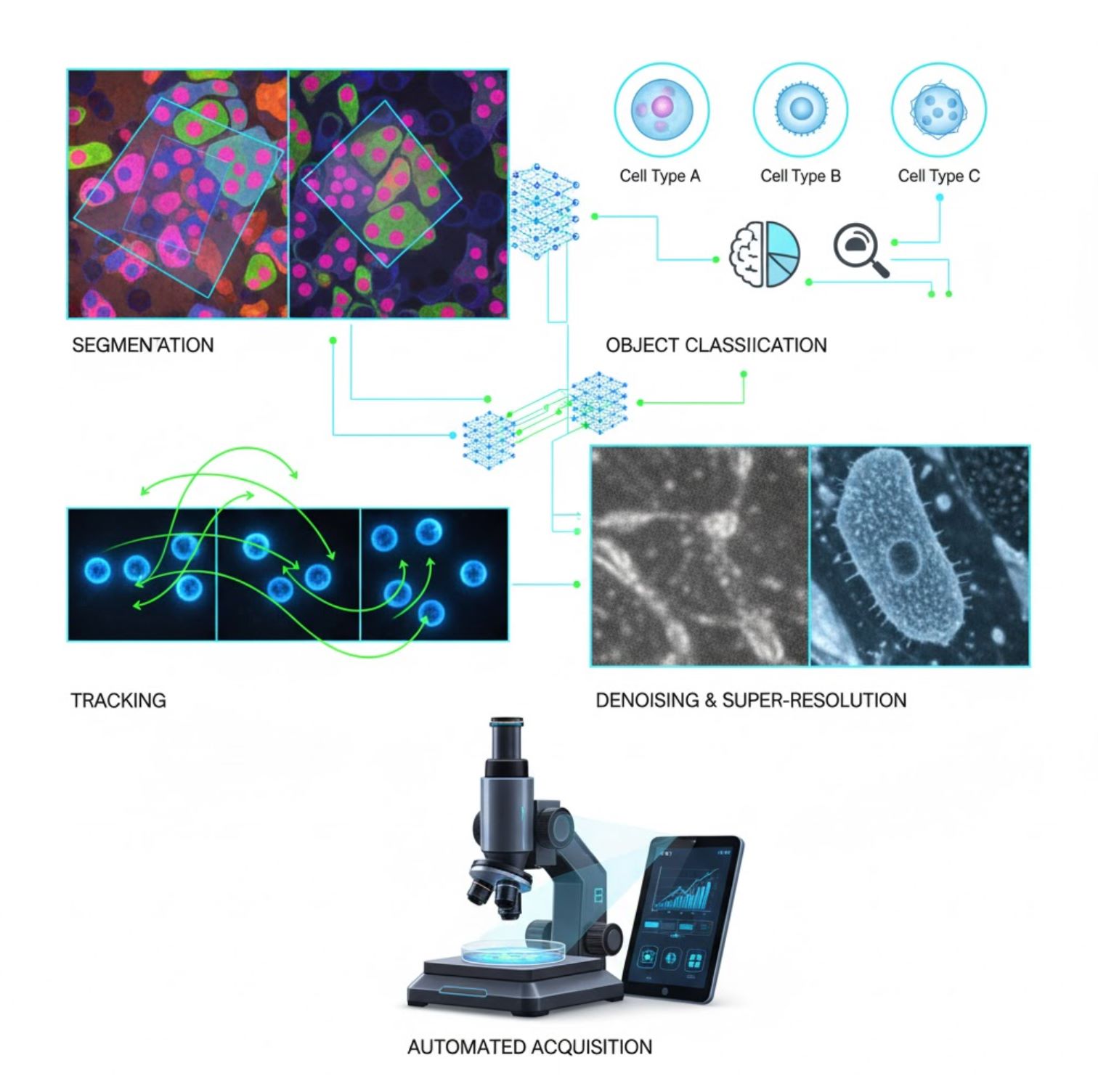
Klíčové aplikace AI v mikroskopii
AI je nyní integrována do mnoha úloh zpracování obrazu v rámci mikroskopického workflow:
Segmentace
Rozdělení snímků na oblasti (např. identifikace každé buňky nebo jádra). Hluboké sítě jako U-Net v tomto excelují.
- Semantická segmentace: označení tříd po pixelech
- Instance segmentace: oddělení jednotlivých objektů
- Vysoká přesnost u hustých nebo slabých snímků
- Vision foundation modely (např. μSAM) nyní přizpůsobené mikroskopii
Klasifikace objektů
Po segmentaci AI klasifikuje každý objekt s vysokou přesností.
- Identifikace typu buňky
- Určení mitotické fáze
- Detekce patologických indikátorů
- Rozlišuje jemné fenotypy obtížně kvantifikovatelné ručně
Sledování
V časosběrné mikroskopii AI sleduje buňky nebo částice mezi snímky s bezprecedentní přesností.
- Hluboké učení výrazně zlepšuje přesnost sledování
- Umožňuje spolehlivou analýzu pohybujících se buněk
- Zachycuje dynamické biologické procesy
Odšumění a superrozlišení
AI modely zlepšují kvalitu snímků odstraněním šumu a rozmazání.
- Fyzikálně informované hluboké modely se učí optiku mikroskopu
- Rekonstruují ostřejší, bezartefaktové snímky
- Vyšší rozlišení s menším množstvím artefaktů oproti tradičním metodám
Automatizované získávání snímků
AI řídí mikroskop v reálném čase.
- Analyzuje živé snímky a činí inteligentní rozhodnutí
- Automaticky upravuje zaostření a skenuje oblasti zájmu
- Snižuje fototoxicitu a šetří čas
- Umožňuje vysokopropustné a adaptivní zobrazovací experimenty
Oblíbené nástroje AI v zpracování mikroskopických snímků
<ITEM_DESCRIPTION>Ekosystém nástrojů bohatě podporuje umělou inteligenci v mikroskopii. Výzkumníci vyvinuli jak obecné, tak specializované softwary, z nichž mnoho je open-source:</ITEM_DESCRIPTION>
Cellpose
| Vývojáři | Carsen Stringer a Marius Pachitariu (výzkumná skupina MouseLand) |
| Podporované platformy |
Vyžaduje Python (instalace přes pip/conda). GUI dostupné pouze na desktopu. |
| Jazyková podpora | Dokumentace v angličtině; celosvětově používaný ve výzkumných laboratořích |
| Cenový model | Zdarma a open-source pod licencí BSD-3-Clause |
Přehled
Cellpose je pokročilý nástroj pro segmentaci založený na hlubokém učení, určený pro mikroskopické snímky. Jako univerzální algoritmus přesně segmentuje různé typy buněk (jádra, cytoplazma atd.) napříč různými zobrazovacími modalitami bez nutnosti přeškolení modelu. Díky funkci zpětné vazby uživatele mohou vědci výsledky vylepšovat, přizpůsobovat model svým datům a používat systém jak pro 2D, tak 3D zobrazovací postupy.
Klíčové vlastnosti
Fungují ihned pro širokou škálu typů buněk, barvení a zobrazovacích modalit bez potřeby vlastního tréninku.
Podpora kompletních 3D dat pomocí přístupu „2,5D“, který znovu využívá 2D modely pro objemová data.
Manuální oprava výsledků segmentace a přeškolení modelu na vlastních datech pro lepší přesnost.
Přístup přes Python API, příkazový řádek nebo grafické uživatelské rozhraní pro flexibilní pracovní postupy.
Odšumování, odstranění rozmazání a zvětšení rozlišení pro zlepšení kvality obrazu před segmentací.
Stažení nebo přístup
Technické pozadí
Cellpose byl představen v průlomové studii autorů Stringer, Wang, Michaelos a Pachitariu, trénovaný na rozsáhlém a velmi rozmanitém datasetu obsahujícím přes 70 000 segmentovaných objektů. Tato rozmanitost umožňuje modelu zobecnit různé tvary, velikosti buněk a mikroskopická nastavení, což výrazně snižuje potřebu vlastního tréninku ve většině případů. Pro 3D data Cellpose chytře znovu využívá svůj 2D model v režimu „2,5D“, čímž se vyhne nutnosti plně 3D anotovaných tréninkových dat a přesto poskytuje objemovou segmentaci. Verze Cellpose 2.0 zavedla přeškolení s uživatelskou zpětnou vazbou, které umožňuje uživatelům manuálně opravovat předpovědi a přeškolit model na vlastních snímcích pro lepší výkon na specifických datech.
Instalace a nastavení
Nastavte Python prostředí pomocí conda:
conda create -n cellpose python=3.10Aktivujte prostředí a nainstalujte Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeZačínáme
Režim GUI
- Spusťte GUI příkazem:
python -m cellpose - Přetáhněte soubory s obrázky (
.tif,.pngatd.) do rozhraní - Vyberte typ modelu (např. „cyto“ pro cytoplazmu nebo „nuclei“ pro jádra)
- Nastavte odhadovaný průměr buněk nebo nechte Cellpose automaticky kalibrovat
- Klikněte pro spuštění segmentace a zobrazení výsledků
Režim Python API
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Vylepšení a přeškolení
- Po vytvoření masek ručně opravte segmentaci v GUI sloučením nebo odstraněním masek
- Použijte vestavěné tréninkové funkce k přeškolení na opravených příkladech
- Zlepšení výkonu modelu na vašem konkrétním datasetu
Zpracování 3D dat
- Nahrajte multi-Z TIFF nebo objemový stack
- V GUI nebo API použijte přepínač
--Zstackpro zpracování jako 3D - Volitelně vylepšete 3D toky pomocí vyhlazování nebo specializovaných parametrů pro lepší segmentaci
Omezení a úvahy
- Obecnost modelu: I když univerzální model funguje široce, velmi neobvyklé tvary buněk nebo zobrazovací podmínky mohou vyžadovat přeškolení.
- Časová náročnost anotací: Trénink s uživatelskou zpětnou vazbou vyžaduje manuální opravy, což může být časově náročné u velkých datasetů.
- Složitost instalace: Instalace GUI může vyžadovat práci s příkazovým řádkem, conda prostředími a správu Python závislostí – není vždy jednoduchá pro neprogramátory.
- Pouze desktop: Cellpose je navržen pro desktopové použití; nejsou k dispozici nativní aplikace pro Android nebo iOS.
Často kladené otázky
Ne – Cellpose poskytuje předtrénované univerzální modely, které často fungují dobře bez přeškolení. Pro optimální výsledky na speciálních nebo neobvyklých datech však můžete anotovat a přeškolit model pomocí funkcí s uživatelskou zpětnou vazbou.
Ano – podporuje 3D zpracování znovuvyužitím svého 2D modelu (tzv. „2,5D“) a můžete zpracovávat objemové stacky přes GUI nebo API.
GPU je velmi doporučena pro rychlejší inferenci a trénink, zejména u velkých nebo 3D datasetů, ale Cellpose lze provozovat i na strojích pouze s CPU s pomalejším výkonem.
V GUI nastavte odhadovaný průměr buněk ručně nebo nechte Cellpose automaticky kalibrovat. Výsledky můžete vylepšit a model přeškolit, pokud segmentace není optimální.
Ano – novější verze (Cellpose 3) obsahují modely pro obnovu obrazu, které umožňují odšumování, odstranění rozmazání a zvětšení rozlišení pro zlepšení kvality segmentace před zpracováním.
StarDist
| Vývojáři | Uwe Schmidt, Martin Weigert, Coleman Broaddus a Gene Myers |
| Podporované platformy |
|
| Jazyková podpora | Open-source projekt s dokumentací a komunitou převážně v angličtině |
| Cenový model | Zdarma a otevřený zdroj. Licencováno pod BSD-3-Clause |
Přehled
StarDist je nástroj hlubokého učení pro segmentaci instancí v mikroskopických snímcích. Každý objekt (například buněčná jádra) reprezentuje jako hvězdicově konvexní polygon v 2D nebo polyedr v 3D, což umožňuje přesnou detekci a oddělení hustě uspořádaných nebo překrývajících se objektů. Díky robustní architektuře je StarDist široce používán pro automatizovanou segmentaci buněk a jader ve fluorescenční mikroskopii, histopatologii a dalších aplikacích bioobrazové analýzy.
Klíčové vlastnosti
Vysoce přesná segmentace instancí pomocí hvězdicově konvexních polygonů (2D) a polyedrů (3D) pro spolehlivou detekci objektů.
Specializované modely pro 2D snímky i 3D objemová data pro komplexní mikroskopickou analýzu.
Modely připravené k použití pro fluorescenční jádra, histologii barvenou H&E a další běžné zobrazovací scénáře.
Klasifikace detekovaných objektů do různých tříd (např. různé typy buněk) v jednom běhu segmentace.
Bezproblémová integrace s ImageJ/Fiji, QuPath a napari pro přístupné workflow založené na GUI.
Komplexní vyhodnocení segmentace instancí včetně přesnosti, úplnosti, F1 skóre a panoptické kvality.
Technické pozadí
Původně představený v článku MICCAI 2018, hlavní inovací StarDist je předpověď radiálních vzdáleností podél pevně daných paprsků v kombinaci s pravděpodobností objektu pro každý pixel, což umožňuje přesnou rekonstrukci hvězdicově konvexních tvarů. Tento přístup spolehlivě segmentuje těsně sousedící objekty, které je obtížné oddělit tradičními metodami založenými na pixelech nebo ohraničujících rámečcích.
Novější vývoj rozšířil StarDist na histopatologické snímky, umožňující nejen segmentaci jader, ale i klasifikaci více tříd detekovaných objektů. Metoda dosáhla špičkových výsledků v soutěžích jako CoNIC (Colon Nuclei Identification and Counting).
Stažení nebo přístup
Instalace a nastavení
Nainstalujte TensorFlow (verze 1.x nebo 2.x) jako předpoklad pro StarDist.
Použijte pip k instalaci Python balíčku StarDist:
pip install stardistPro napari:
pip install stardist-napariPro QuPath: Nainstalujte rozšíření StarDist přetažením souboru .jar do QuPath.
Pro ImageJ/Fiji: Použijte vestavěný správce pluginů nebo manuální instalaci přes menu pluginů.
Spuštění segmentace
Nahrajte předtrénovaný model, normalizujte svůj snímek a spusťte predikci:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Otevřete svůj snímek v napari, vyberte plugin StarDist, zvolte předtrénovaný nebo vlastní model a spusťte predikci přímo z GUI.
Použijte plugin StarDist z menu Pluginy pro aplikaci modelu na váš obrazový zásobník s intuitivním rozhraním.
Po instalaci rozšíření spusťte detekci StarDist přes skriptovací konzoli nebo grafické rozhraní QuPath pro histopatologickou analýzu.
Trénink a doladění
Vytvořte obrázky s pravdivými anotacemi, kde je každý objekt jednoznačně označen. Použijte nástroje pro anotace jako LabKit, QuPath nebo Fiji k přípravě datové sady.
Použijte Python API StarDist k natrénování nového modelu nebo doladění existujícího s vašimi vlastními anotovanými daty.
Možnosti post-processingu
- Aplikujte potlačení maxim (NMS) k odstranění redundantních kandidátních tvarů
- Použijte StarDist OPP (post-processing objektů) pro sloučení masek u ne-hvězdicově konvexních tvarů
Omezení a úvahy
- Předpoklad hvězdicové konvexnosti nemusí dokonale modelovat vysoce ne-konvexní nebo velmi nepravidelné tvary
- Složitost instalace: vlastní instalace vyžadují kompatibilní C++ kompilátor pro sestavení rozšíření
- Akcelerace na GPU závisí na kompatibilních verzích TensorFlow, CUDA a cuDNN
- Někteří uživatelé hlásí problémy se spuštěním pluginu ImageJ kvůli konfiguraci Javy
Často kladené otázky
StarDist pracuje s různými typy snímků včetně fluorescenčních, světlopólových a histopatologických (např. H&E), díky flexibilním předtrénovaným modelům a přizpůsobitelnosti různým zobrazovacím modalitám.
Ano — StarDist podporuje 3D segmentaci instancí pomocí hvězdicově konvexních polyedrů pro objemová data, čímž rozšiřuje 2D možnosti na plnou 3D analýzu.
Není to nutné. Předtrénované modely jsou k dispozici a často fungují dobře ihned po instalaci. Nicméně pro specializovaná nebo nová data výrazně zvyšuje přesnost anotace a trénink vlastních modelů.
StarDist se integruje s napari, ImageJ/Fiji a QuPath, což umožňuje spouštět segmentaci z GUI bez programování. Podporuje také přímé použití Python API pro pokročilé workflow.
StarDist poskytuje vestavěné funkce pro výpočet běžných metrik segmentace instancí včetně přesnosti, úplnosti, F1 skóre a panoptické kvality pro hodnocení výkonu segmentace.
SAM
Informace o aplikaci
| Vývojář | Meta AI Research (FAIR) |
| Podporovaná zařízení |
|
| Jazyk a dostupnost | Otevřený základní model dostupný globálně; dokumentace v angličtině |
| Cena | Zdarma — open-source pod licencí Meta přes GitHub a integraci v MIB |
Obecný přehled
SAM (Segment Anything Model) je výkonný základní model umělé inteligence vytvořený společností Meta, který umožňuje interaktivní a automatickou segmentaci prakticky jakéhokoli objektu na obrázcích. Pomocí promptů jako jsou body, ohraničující rámečky nebo hrubé masky SAM generuje segmentační masky bez nutnosti specifického přeškolení pro daný úkol. Ve výzkumu mikroskopie byla flexibilita SAM přizpůsobena pro segmentaci buněk, detekci organel a analýzu histopatologie, což nabízí škálovatelné řešení pro výzkumníky, kteří potřebují promptovatelný, univerzální segmentační nástroj.
Podrobný úvod
Původně trénovaný společností Meta na více než 1 miliardě masek napříč 11 miliony obrázků, byl SAM navržen jako promptovatelný základní model pro segmentaci s výkonem „zero-shot“ na nových doménách. Ve výzkumu lékařského zobrazování byl SAM testován na segmentaci celých skenů patologie, detekci nádorů a identifikaci buněčných jader. Jeho výkon u hustě uspořádaných instancí – jako jsou buněčná jádra – je však smíšený: i při rozsáhlých promptech (např. 20 kliknutí nebo rámečků) může segmentace bez doladění v komplexních mikroskopických obrazech selhávat.
Pro řešení tohoto omezení vznikly doménově specifické adaptace:
- SAMCell — Doladěný na rozsáhlých mikroskopických datech pro silnou segmentaci bez nutnosti přeškolení pro různé typy buněk
- μSAM — Přeškolený na více než 17 000 ručně anotovaných mikroskopických obrázcích pro zvýšení přesnosti u malých buněčných struktur
Klíčové vlastnosti
Flexibilní interakce pomocí bodů, rámečků a masek pro přesnou kontrolu.
Provádí segmentaci bez doladění na nových obrazových doménách.
Přizpůsobitelný pro mikroskopii a histopatologii pomocí few-shot nebo promptového přeškolení.
Dostupný v Microscopy Image Browser (MIB) s podporou 3D a interpolované segmentace.
IDCC-SAM umožňuje automatické počítání buněk v imunocytochemii bez manuální anotace.
Stažení nebo přístup
Uživatelská příručka
- Otevřete Microscopy Image Browser a přejděte do panelu segmentace SAM
- Nakonfigurujte Python interpreter a vyberte mezi modely SAM-1 nebo SAM-2
- Pro akceleraci na GPU vyberte „cuda“ v prostředí spuštění (doporučeno pro optimální výkon)
- Prompty bodů: Klikněte na objekt pro definování pozitivního semínka; použijte Shift + klik pro rozšíření a Ctrl + klik pro negativní semínka
- 3D zásobníky: Použijte interaktivní 3D režim – klikněte na jeden řez, posuňte kolečkem s Shift a interpolujte semínka mezi řezy
- Režim úprav: Nahrazujte, přidávejte, odečítejte masky nebo vytvářejte novou vrstvu podle potřeby
- Použijte možnost „Automaticky vše“ v panelu SAM-2 v MIB pro segmentaci všech viditelných objektů v oblasti
- Po segmentaci masky zkontrolujte a případně upravte
- Použijte promptové doladění (např. „All-in-SAM“) pro generování pixelových anotací z řídkých uživatelských promptů
- Pro počítání buněk aplikujte IDCC-SAM, který využívá SAM v zero-shot pipeline s následným zpracováním
- Pro vysoce přesnou segmentaci buněk použijte SAMCell, doladěný na mikroskopických snímcích buněk
Omezení a úvahy
- Zero-shot výkon nekonzistentní u hustých nebo překrývajících se struktur bez doménového doladění
- Kvalita segmentace silně závisí na navržení a strategii promptu
- Důrazně doporučeno GPU; inference na CPU je velmi pomalá
- Má potíže s velmi vysokým rozlišením celoplošných snímků a víceúrovňovými tkáňovými strukturami
- Doladění nebo adaptace SAM pro mikroskopii může vyžadovat znalosti strojového učení
Často kladené otázky
Ano – prostřednictvím adaptací jako SAMCell, který doladí SAM na mikroskopických datech speciálně pro úkoly segmentace buněk.
Ne vždy. S IDCC-SAM můžete provádět počítání buněk bez předchozích manuálních anotací.
Použijte promptové doladění (např. „All-in-SAM“) nebo předtrénované mikroskopické verze jako μSAM, který je trénován na více než 17 000 anotovaných mikroskopických obrázcích.
Ačkoliv je možné běžet na CPU, GPU je velmi doporučeno pro praktickou rychlost inference a interaktivní segmentaci v reálném čase.
Ano – integrace SAM-2 v MIB podporuje 3D segmentaci s interpolací semínek mezi řezy pro objemovou analýzu.
AxonDeepSeg
| Vývojář | NeuroPoly Lab na Polytechnique Montréal a Université de Montréal |
| Podporované platformy |
|
| Jazyk | Dokumentace v angličtině; open-source nástroj používaný globálně |
| Cena | Zdarma a open-source |
Přehled
AxonDeepSeg je nástroj poháněný umělou inteligencí pro automatickou segmentaci axonů a myelinu v mikroskopických snímcích. Pomocí konvolučních neuronových sítí poskytuje přesnou třídu segmentace (axon, myelin, pozadí) napříč různými zobrazovacími modalitami včetně TEM, SEM a světlého pole. Automatizací morfometrických měření, jako je průměr axonu, g-poměr a tloušťka myelinu, AxonDeepSeg zefektivňuje kvantitativní analýzu v neurovědeckém výzkumu, výrazně snižuje čas potřebný pro manuální anotace a zlepšuje reprodukovatelnost.
Klíčové vlastnosti
Modely připravené k použití, optimalizované pro modalitu TEM, SEM a světlé pole.
Přesná klasifikace oblastí axonu, myelinu a pozadí v mikroskopických snímcích.
Automatický výpočet průměru axonu, g-poměru, tloušťky myelinu a hustoty.
Integrace s Napari GUI umožňuje ruční úpravy segmentačních masek pro zvýšení přesnosti.
Bezproblémová integrace do vlastních pracovních postupů pro rozsáhlou analýzu nervové tkáně.
Komplexní testovací skripty zajišťují reprodukovatelnost a spolehlivé výsledky segmentace.
Technické detaily
Vyvinutý NeuroPoly Labem, AxonDeepSeg využívá hluboké učení k dosažení vysoce přesné segmentace pro neurovědecké aplikace. Předtrénované modely jsou dostupné pro různé mikroskopické modality, což zajišťuje univerzálnost napříč zobrazovacími technikami. Nástroj se integruje s Napari, což umožňuje interaktivní korekce segmentačních masek a zvyšuje přesnost u náročných datových sad. AxonDeepSeg počítá klíčové morfometrické metriky a podporuje tak rozsáhlé studie struktury a patologie nervové tkáně. Jeho pythonové prostředí umožňuje integraci do vlastních pracovních postupů pro rozsáhlou analýzu morfologie axonů a myelinu.
Stažení nebo přístup
Instalace a nastavení
Ujistěte se, že máte nainstalovaný Python 3.8 nebo novější, poté nainstalujte AxonDeepSeg a Napari pomocí pip:
pip install axondeepseg napariSpusťte dodané testovací skripty, abyste potvrdili, že všechny komponenty jsou správně nainstalovány a fungují.
Importujte mikroskopické snímky (TEM, SEM nebo světlé pole) do Napari nebo vašeho Python prostředí.
Vyberte vhodný předtrénovaný model pro vaši zobrazovací modalitu a spusťte segmentaci pro vytvoření masek axonů a myelinu.
Automaticky vypočítejte morfometrická měření včetně průměru axonu, g-poměru, hustoty a tloušťky myelinu a exportujte výsledky ve formátu CSV.
Použijte grafické rozhraní Napari k ruční úpravě segmentačních masek tam, kde je to potřeba, slučujte nebo mažte masky pro lepší přesnost.
Důležité poznámky
- Výkon může být nižší u nových nebo nevytrénovaných zobrazovacích modalit
- Pro náročné nebo složité oblasti může být nutná ruční korekce
- Pro rychlejší zpracování velkých datových sad se doporučuje GPU; zpracování na CPU je také podporováno
Často kladené otázky
AxonDeepSeg podporuje TEM (transmisní elektronová mikroskopie), SEM (skenovací elektronová mikroskopie) a světlou pole s předtrénovanými modely optimalizovanými pro každou modalitu.
Ano, AxonDeepSeg je zcela zdarma a open-source, dostupný pro akademické i komerční použití.
Ano, AxonDeepSeg automaticky vypočítává průměr axonu, g-poměr, tloušťku myelinu a hustotu z segmentovaných snímků.
GPU se doporučuje pro rychlejší segmentaci velkých datových sad, ale zpracování na CPU je podporováno pro menší analýzy.
Ano, integrace s Napari GUI umožňuje interaktivní korekce a úpravy segmentačních masek pro vyšší přesnost u náročných oblastí.
Ilastik
| Vývojář | Tým Ilastik při Evropské molekulární biologické laboratoři (EMBL) a přidružení akademičtí partneři |
| Podporované platformy |
|
| Jazyk | Angličtina |
| Cena | Zdarma a open-source |
Přehled
Ilastik je výkonný nástroj řízený umělou inteligencí pro interaktivní segmentaci, klasifikaci a analýzu mikroskopických dat. Pomocí technik strojového učení, jako jsou klasifikátory Random Forest, umožňuje vědcům segmentovat pixely, klasifikovat objekty, sledovat buňky v čase a provádět počítání hustoty v 2D i 3D datech. Díky intuitivnímu rozhraní a zpětné vazbě v reálném čase je Ilastik přístupný vědcům bez programátorských znalostí a je široce využíván v buněčné biologii, neurovědách a biomedicínském zobrazování.
Klíčové vlastnosti
Zpětná vazba v reálném čase při anotaci reprezentativních oblastí pro okamžité výsledky segmentace.
Kategorizace segmentovaných struktur na základě morfologických a intenzitních vlastností.
Sledování pohybu a dělení buněk v 2D a 3D časových mikroskopických experimentech.
Kvantifikace hustě zaplněných oblastí bez explicitní segmentace jednotlivých objektů.
Poloautomatická segmentace složitých 3D objemů s intuitivní interakcí.
Automatické zpracování více snímků pomocí režimu bez grafického rozhraní (headless).
Stažení
Průvodce začátkem práce
Stáhněte Ilastik pro svůj operační systém z oficiálních stránek. Balíček obsahuje všechny potřebné Python závislosti, proto postupujte podle instalačních pokynů pro vaši platformu.
Otevřete Ilastik a vyberte svůj analytický workflow: klasifikace pixelů, klasifikace objektů, sledování nebo počítání hustoty. Načtěte svůj obrazový dataset, který může obsahovat vícekanálové, 3D nebo časové snímky.
Označte několik reprezentativních pixelů nebo objektů ve svých snímcích. Klasifikátor Random Forest v Ilastiku se z těchto anotací naučí a automaticky předpoví štítky pro celý dataset.
Aplikujte natrénovaný model pro segmentaci nebo klasifikaci celého datasetu. Exportujte výsledky jako označené obrázky, pravděpodobnostní mapy nebo kvantitativní tabulky pro další analýzu a vizualizaci.
Využijte režim bez grafického rozhraní Ilastiku pro automatické zpracování více snímků bez manuálního zásahu, ideální pro rozsáhlé analytické pipeline.
Omezení a úvahy
- Interaktivní označování může být časově náročné u velmi rozsáhlých datasetů
- Přesnost závisí na kvalitě a reprezentativnosti uživatelských anotací
- Požadavky na paměť — velmi vysoké rozlišení nebo vícergigabajtové datasetů mohou vyžadovat značnou kapacitu RAM
- Složitá data — klasifikátory Random Forest mohou být méně výkonné než hluboké neuronové sítě u vysoce variabilních nebo složitých obrazových dat
Často kladené otázky
Ano, Ilastik plně podporuje 3D objemy a časové experimenty pro segmentaci, sledování a kvantitativní analýzu napříč více časovými body.
Ano, Ilastik je zcela zdarma a open-source, dostupný všem uživatelům bez licenčních omezení.
Ne, Ilastik nabízí intuitivní grafické rozhraní se zpětnou vazbou v reálném čase, což jej činí přístupným vědcům bez programátorských znalostí. Pokročilí uživatelé mohou také využít dávkové zpracování přes příkazový řádek.
Ano, specializovaný workflow pro sledování umožňuje analýzu pohybu a dělení buněk v 2D i 3D časových datech s automatickým sledováním rodokmenu.
Výstupy segmentace lze exportovat jako označené obrázky, pravděpodobnostní mapy nebo kvantitativní tabulky, což umožňuje bezproblémovou integraci s nástroji pro další analýzu a vizualizaci.
Tyto nástroje pokrývají úrovně od začátečníků po experty. Mnohé jsou zdarma a open-source, což usnadňuje reprodukovatelné a sdílené AI pracovní postupy v rámci výzkumné komunity.
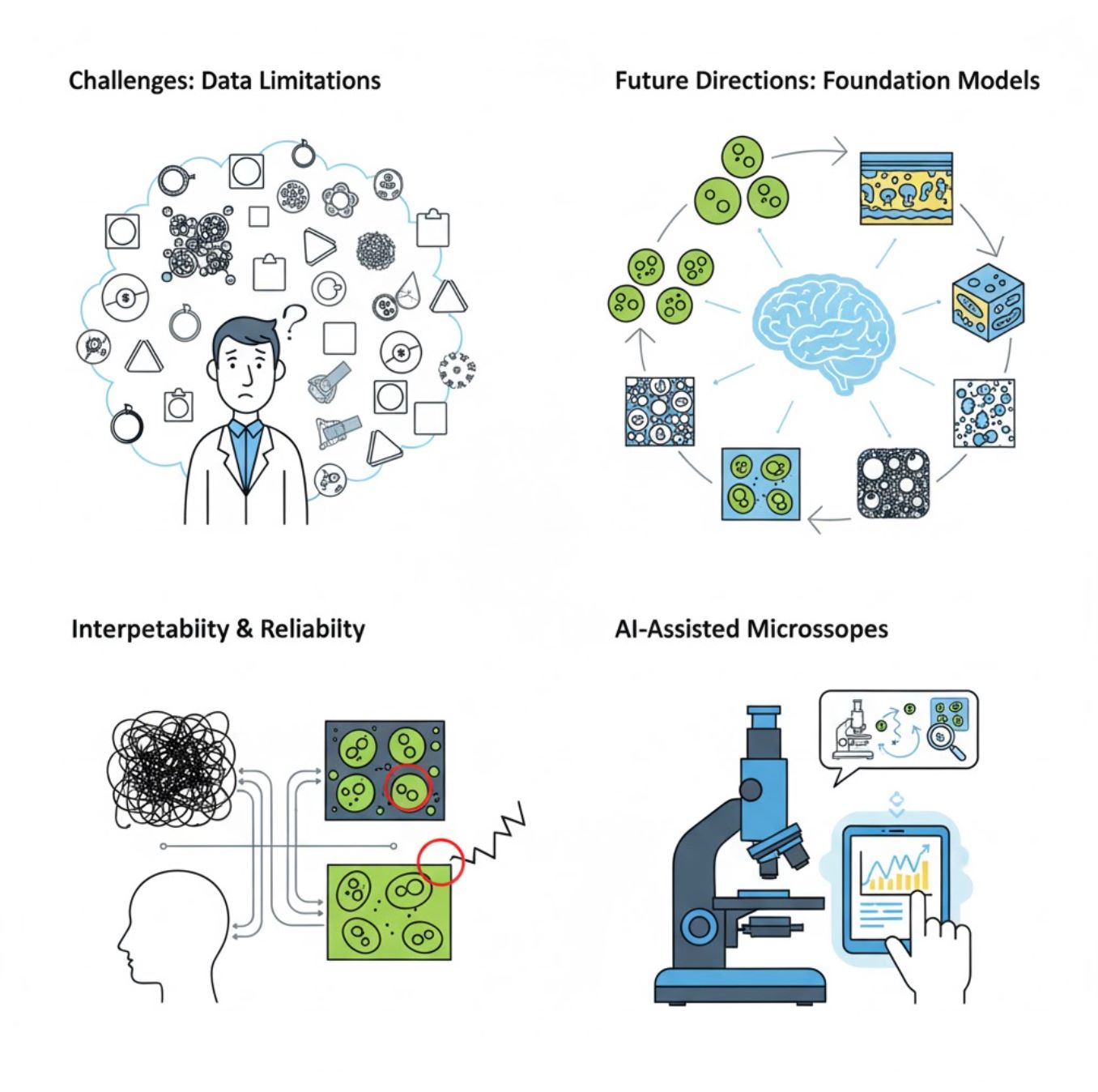
Výzvy a budoucí směřování
Současné výzvy
Nové trendy
Vision foundation modely
Systémy nové generace slibují snížit potřebu úzce specializovaného tréninku.
- Modely jako SAM a systémy založené na CLIP
- Jeden AI zvládne mnoho mikroskopických úloh
- Rychlejší nasazení a adaptace
Mikroskopy s asistencí AI
Plně autonomní a inteligentní mikroskopické systémy se stávají realitou.
- Ovládání přirozeným jazykem pomocí LLM
- Plně automatizované zpětnovazební smyčky
- Dostupnost pokročilé mikroskopie pro širší veřejnost
Klíčové závěry
- AI rychle mění zpracování mikroskopických snímků s lepší přesností a automatizací
- Hluboké učení překonává tradiční strojové učení u složitých, variabilních mikroskopických snímků
- CNN automaticky učí hierarchické rysy ze surových pixelů pro robustní analýzu
- Klíčové aplikace zahrnují segmentaci, klasifikaci, sledování, odšumění a automatizované získávání snímků
- Úspěch závisí na kvalitních datech a pečlivé validaci odborníky
- Vision foundation modely a mikroskopy s asistencí AI představují budoucnost oboru
S pokračujícím pokrokem a komunitními snahami (open-source nástroje, sdílené datové sady) se AI stále více stane jádrem „oka“ mikroskopu, pomáhajíc vědcům vidět neviditelné.







No comments yet. Be the first to comment!