Inteligența artificială în procesarea imaginilor microscopice
Inteligența artificială revoluționează procesarea imaginilor microscopice cu capacități puternice precum segmentarea precisă, reducerea zgomotului, super-rezoluția și achiziția automată a imaginilor. Acest articol evidențiază instrumentele esențiale de inteligență artificială și tendințele emergente în cercetarea științifică.
Tehnicile de inteligență artificială revoluționează microscopie prin optimizarea achiziției imaginilor și automatizarea analizei. În microscoapele inteligente moderne, modulele AI pot ajusta parametrii de imagistică în timp real (de exemplu, focalizarea, iluminarea) pentru a minimiza fotodegradarea și a îmbunătăți semnalul. Între timp, algoritmii de învățare profundă pot analiza date complexe de imagini pentru a extrage informații biologice ascunse și chiar pentru a corela imaginile cu alte date (de exemplu, genomica).
Metode AI: Învățare Automată vs Învățare Profundă
Metodele AI variază de la clasicul învățare automată (ML) la modernul învățare profundă (DL). Fiecare abordare are puncte forte și limitări distincte:
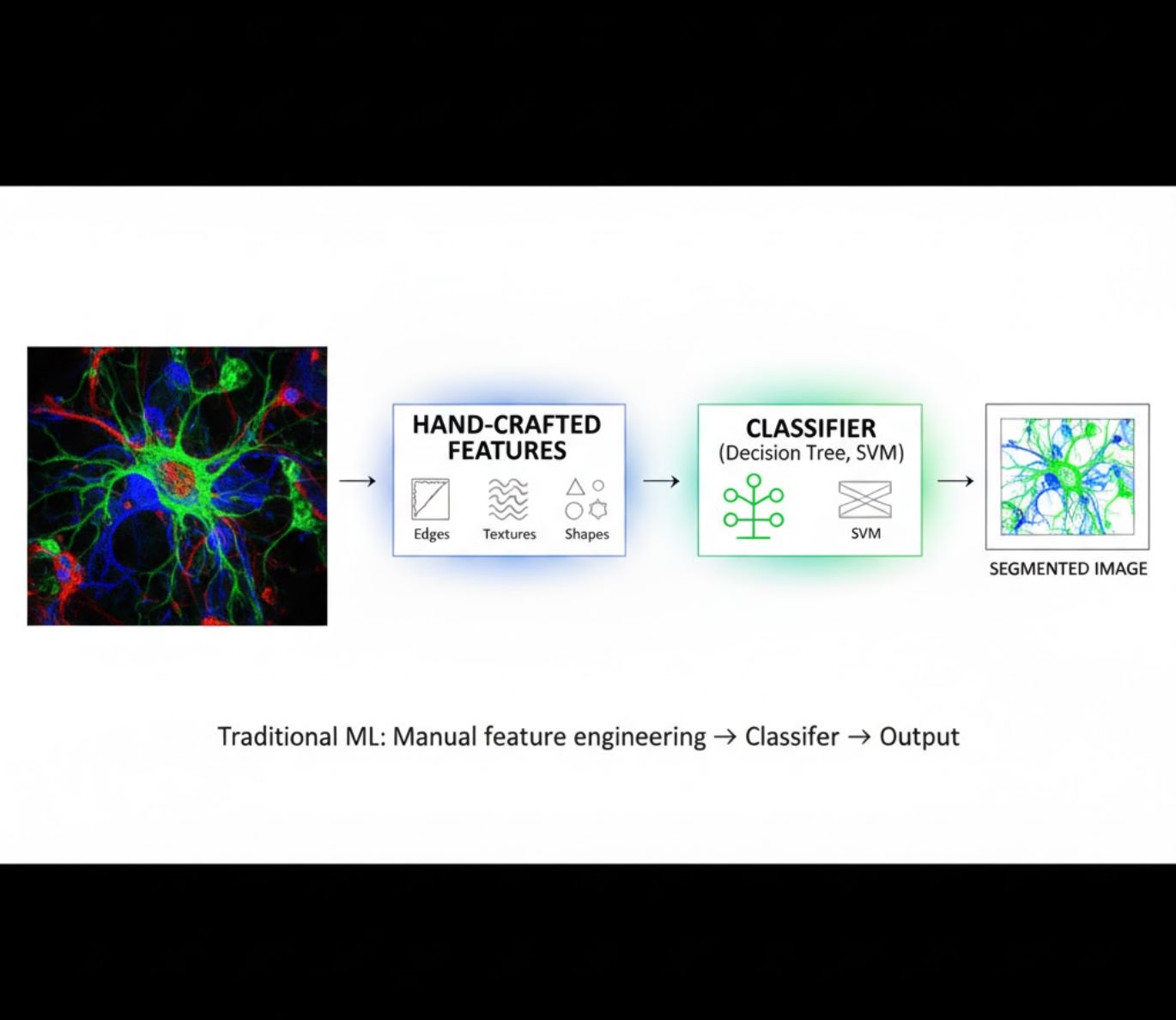
Caracteristici realizate manual
- Cercetătorii creează manual caracteristici ale imaginii (margini, texturi, forme)
- Caracteristicile sunt introduse în clasificatori (arbori decizionali, SVM)
- Rapid de antrenat
- Se descurcă greu cu imagini complexe sau zgomotoase
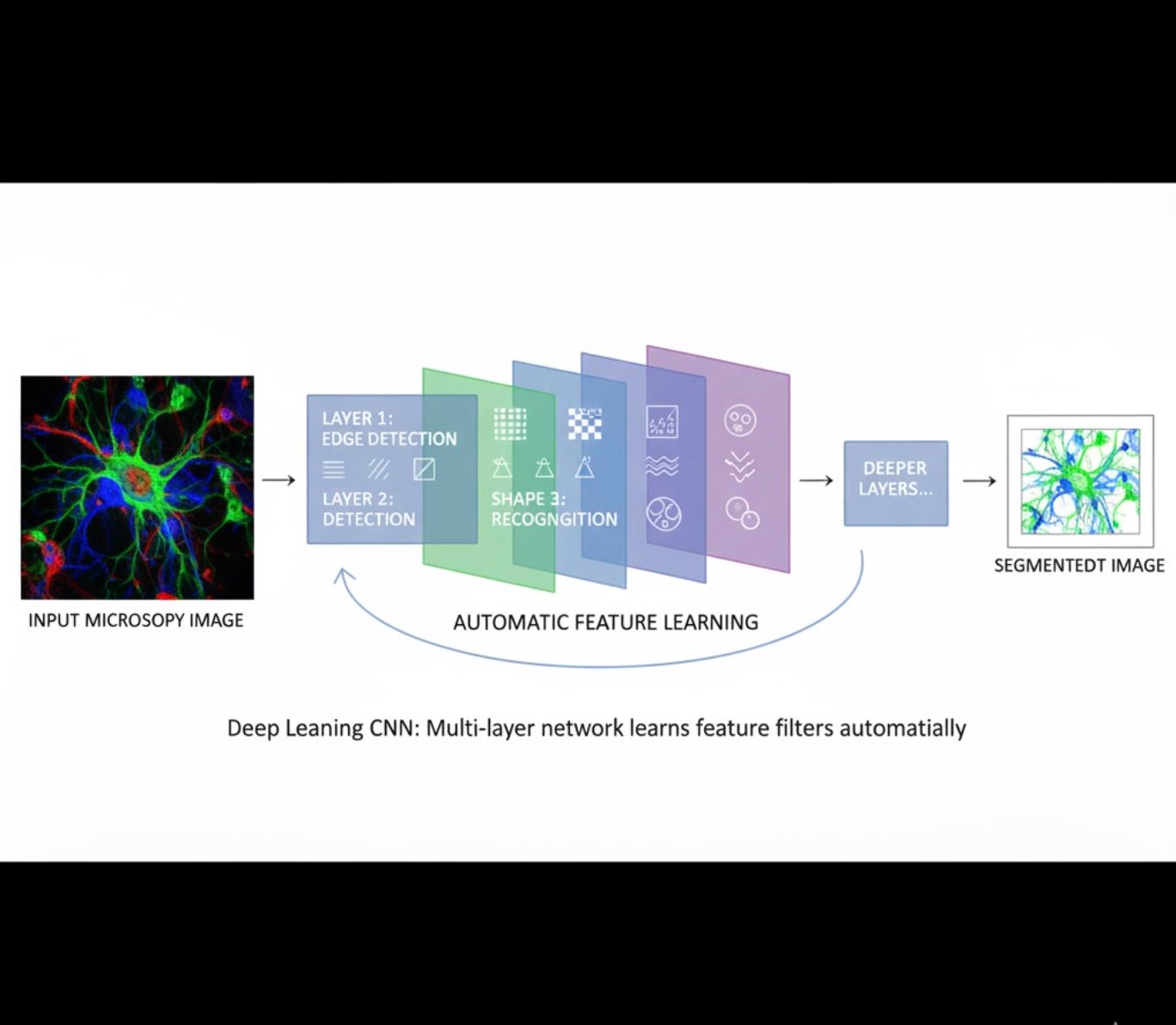
Învățare automată a caracteristicilor
- Rețele neuronale multi-strat (CNN) învață caracteristicile automat
- Învățare end-to-end din pixeli neprelucrați
- Mult mai robustă la variații
- Captură fiabilă a texturilor și structurilor complexe
Cum funcționează CNN-urile: Rețelele neuronale convoluționale aplică filtre succesive imaginilor microscopice, învățând să detecteze tipare simple (margini) în straturile inițiale și structuri complexe (forme celulare, texturi) în straturile profunde. Această învățare ierarhică face ca DL să fie excepțional de robustă chiar și când profilurile de intensitate variază semnificativ.
Comparare vizuală: fluxuri ML vs DL
Aplicații cheie AI în microscopie
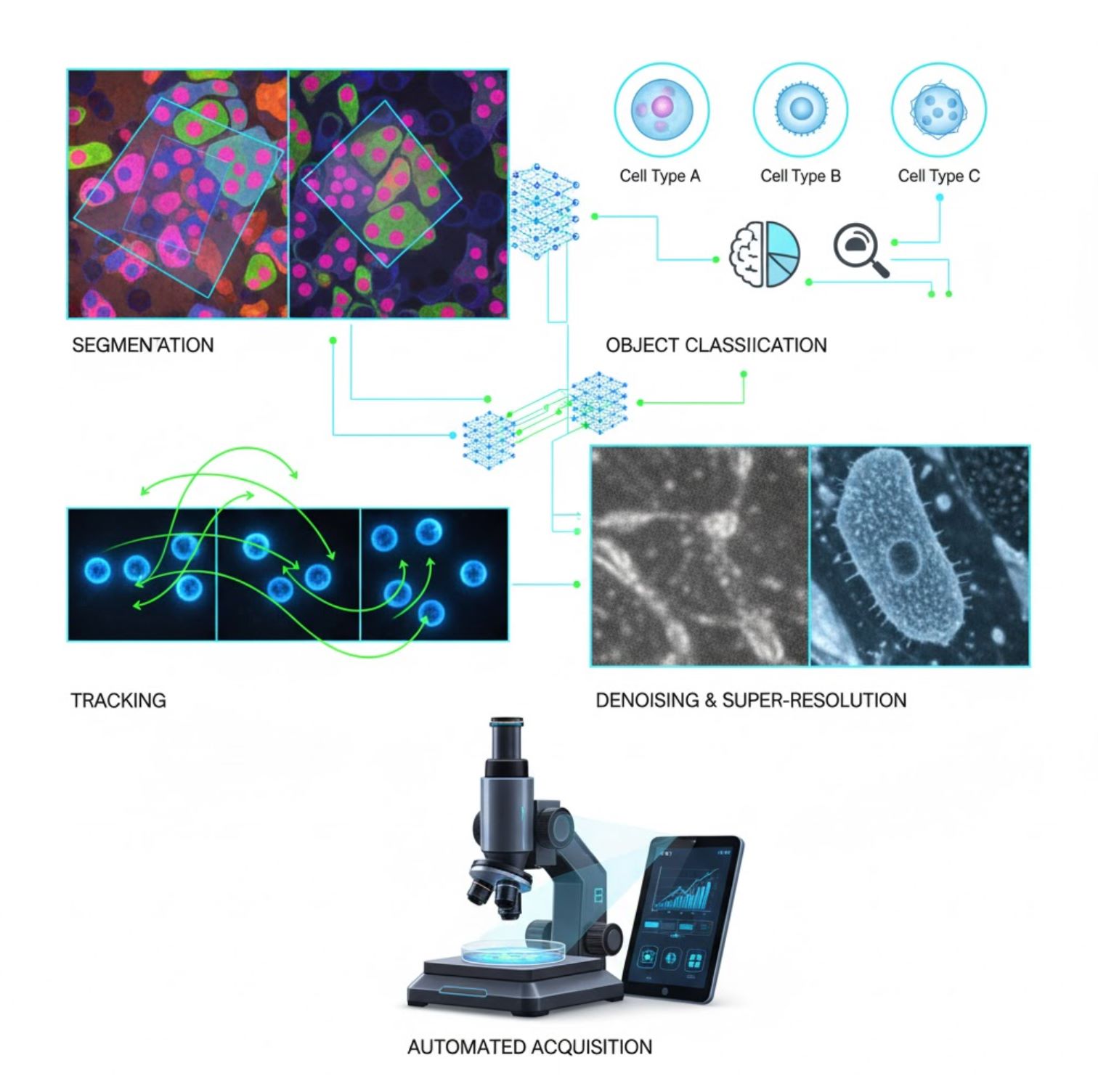
Inteligența artificială este acum integrată în multe sarcini de procesare a imaginilor pe tot fluxul de lucru al microscopiei:
Segmentare
Împărțirea imaginilor în regiuni (de exemplu, identificarea fiecărei celule sau nucleu). Rețelele profunde precum U-Net excelează în această sarcină.
- Segmentare semantică: etichete pe pixel
- Segmentare pe instanțe: separarea obiectelor individuale
- Precizie ridicată pe imagini aglomerate sau întunecate
- Modele fundamentale de viziune (ex. μSAM) adaptate acum pentru microscopie
Clasificarea obiectelor
După segmentare, AI clasifică fiecare obiect cu precizie înaltă.
- Identificarea tipului celular
- Determinarea stadiului mitotic
- Detectarea indicatorilor patologici
- Distingerea fenotipurilor subtile greu de cuantificat manual
Urmărire
În microscopie time-lapse, AI urmărește celulele sau particulele prin cadre cu o precizie fără precedent.
- Învățarea profundă îmbunătățește dramatic acuratețea urmăririi
- Permite analiza fiabilă a celulelor în mișcare
- Captură a proceselor biologice dinamice
Reducerea zgomotului și super-rezoluție
Modelele AI îmbunătățesc calitatea imaginii prin eliminarea zgomotului și a neclarității.
- Modele profunde informate de fizică învață optica microscopului
- Reconstruiesc imagini mai clare, fără artefacte
- Rezoluție mai mare cu artefacte reduse față de metodele tradiționale
Achiziție automată
AI controlează microscopul în timp real.
- Analizează imagini live pentru a lua decizii inteligente
- Ajustează automat focalizarea și scanează zonele de interes
- Reduce fototoxicitatea și economisește timp
- Permite experimente de imagistică adaptivă și cu debit mare
Instrumente populare AI în procesarea imaginilor microscopice
<ITEM_DESCRIPTION>>>Un ecosistem bogat de instrumente sprijină inteligența artificială în microscopie. Cercetătorii au dezvoltat atât software de uz general, cât și specializat, multe dintre acestea fiind open-source: <END_ITEM_DESCRIPTION>>>
Cellpose
| Dezvoltator | Carsen Stringer și Marius Pachitariu (grupul de cercetare MouseLand) |
| Platforme suportate |
Necesită Python (instalare pip/conda). GUI disponibil doar pe desktop. |
| Suport lingvistic | Documentație în limba engleză; adoptat global în laboratoare de cercetare din întreaga lume |
| Model de preț | Gratuit și open-source sub licența BSD-3-Clause |
Prezentare generală
Cellpose este un instrument avansat de segmentare bazat pe învățare profundă, conceput pentru imagini microscopice. Ca algoritm generalist, segmentează cu precizie tipuri diverse de celule (nuclee, citoplasmă etc.) în diferite modalități de imagistică, fără a necesita reantrenarea modelului. Cu capabilități de intervenție umană, cercetătorii pot rafina rezultatele, adapta modelul la propriile date și aplica sistemul atât pe fluxuri de lucru 2D, cât și 3D.
Caracteristici cheie
Funcționează imediat pentru o gamă largă de tipuri de celule, coloranți și modalități de imagistică, fără antrenament personalizat.
Suportă stive 3D complete folosind o abordare „2.5D” care reutilizează modelele 2D pentru date volumetrice.
Corectați manual rezultatele segmentării și reantrenați modelul pe datele dvs. personalizate pentru o acuratețe îmbunătățită.
Acces prin API Python, interfață în linie de comandă sau interfață grafică pentru fluxuri de lucru flexibile.
Capabilități de reducere a zgomotului, eliminare a neclarității și mărire a rezoluției pentru îmbunătățirea calității imaginii înainte de segmentare.
Descărcare sau acces
Fundal tehnic
Cellpose a fost introdus într-un studiu fundamental realizat de Stringer, Wang, Michaelos și Pachitariu, antrenat pe un set de date mare și foarte variat ce conține peste 70.000 de obiecte segmentate. Această diversitate permite modelului să generalizeze peste forme, dimensiuni și setări microscopice diferite, reducând semnificativ necesitatea antrenamentului personalizat în majoritatea cazurilor. Pentru date 3D, Cellpose reutilizează inteligent modelul 2D într-un mod „2.5D”, evitând necesitatea datelor complet etichetate 3D, oferind totuși segmentare volumetrică. Cellpose 2.0 a introdus reantrenarea cu intervenția utilizatorului, permițând corectarea manuală a predicțiilor și reantrenarea pe propriile imagini pentru performanțe îmbunătățite pe seturi specifice de date.
Instalare & configurare
Configurați un mediu Python folosind conda:
conda create -n cellpose python=3.10Activați mediul și instalați Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeÎnceput rapid
Modul GUI
- Lansați GUI rulând:
python -m cellpose - Trageți și plasați fișierele de imagine (
.tif,.pngetc.) în interfață - Selectați tipul modelului (de exemplu, „cyto” pentru citoplasmă sau „nuclei” pentru nuclei)
- Setați diametrul estimat al celulei sau lăsați Cellpose să se calibreze automat
- Faceți clic pentru a începe segmentarea și a vizualiza rezultatele
Modul API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Rafinare & reantrenare
- După generarea măștilor, corectați segmentarea în GUI prin combinarea sau ștergerea măștilor manual
- Folosiți funcțiile încorporate de antrenament pentru a reantrena pe exemplele corectate
- Performanță îmbunătățită a modelului pe setul dvs. specific de date
Procesarea datelor 3D
- Încărcați un TIFF multi-Z sau o stivă volumetrică
- Folosiți flag-ul
--Zstackîn GUI sau API pentru procesare 3D - Opțional, rafinați fluxurile 3D prin netezire sau parametri specializați pentru segmentare mai bună
Limitări & considerații
- Compromis privind generalitatea modelului: Deși modelul generalist funcționează pe scară largă, formele celulare foarte neobișnuite sau condițiile de imagistică pot necesita reantrenare.
- Efort de adnotare: Antrenamentul cu intervenția utilizatorului necesită corecții manuale, care pot fi consumatoare de timp pentru seturi mari de date.
- Complexitate la instalare: Instalarea GUI poate necesita utilizarea liniei de comandă, medii conda și gestionarea dependențelor Python — nu întotdeauna simplu pentru neprogramatori.
- Doar desktop: Cellpose este conceput pentru utilizare pe desktop; nu există aplicații native pentru Android sau iOS.
Întrebări frecvente
Nu — Cellpose oferă modele generaliste pre-antrenate care funcționează bine adesea fără reantrenare. Totuși, pentru rezultate optime pe date speciale sau neobișnuite, puteți adnota și reantrena folosind funcțiile cu intervenția utilizatorului.
Da — suportă 3D prin reutilizarea modelului său 2D (așa-numitul „2.5D”), și puteți procesa stive volumetrice prin GUI sau API.
Un GPU este foarte recomandat pentru inferență și antrenament mai rapid, în special pe seturi mari sau 3D, dar Cellpose poate rula și pe mașini doar cu CPU, cu performanțe mai lente.
În GUI, setați manual diametrul estimat al celulei sau lăsați Cellpose să îl calibreze automat. Puteți rafina rezultatele și reantrena dacă segmentarea nu este optimă.
Da — versiunile mai noi (Cellpose 3) includ modele de restaurare a imaginii pentru reducerea zgomotului, eliminarea neclarității și mărirea rezoluției pentru a îmbunătăți calitatea segmentării înainte de procesare.
StarDist
| Dezvoltatori | Uwe Schmidt, Martin Weigert, Coleman Broaddus și Gene Myers |
| Platforme suportate |
|
| Suport lingvistic | Proiect open-source cu documentație și comunitate în principal în limba engleză |
| Model de preț | Gratuit și open source. Licențiat sub BSD-3-Clause |
Prezentare generală
StarDist este un instrument de învățare profundă pentru segmentarea instanțelor în imagini microscopice. Reprezintă fiecare obiect (cum ar fi nucleele celulare) ca un poligon star-convex în 2D sau poliedru în 3D, permițând detectarea și separarea precisă a obiectelor dens grupate sau suprapuse. Cu arhitectura sa robustă, StarDist este utilizat pe scară largă pentru segmentarea automată a celulelor și nucleelor în microscopie cu fluorescență, histopatologie și alte aplicații de analiză bioimagistică.
Caracteristici cheie
Segmentare de instanțe extrem de precisă folosind poligoane star-convexe (2D) și poliedre (3D) pentru detectarea fiabilă a obiectelor.
Modele dedicate atât pentru imagini 2D, cât și pentru date volumetrice 3D pentru o analiză completă a microscopiei.
Modele gata de utilizare pentru nuclee fluorescente, histologie colorată H&E și alte scenarii comune de imagistică.
Clasifică obiectele detectate în clase distincte (de exemplu, tipuri diferite de celule) într-o singură rulare de segmentare.
Integrare fără întreruperi cu ImageJ/Fiji, QuPath și napari pentru fluxuri de lucru accesibile bazate pe GUI.
Evaluare completă a segmentării instanțelor, inclusiv precizie, recall, scor F1 și calitate panoptică.
Fundal tehnic
Inițial introdus într-un articol MICCAI 2018, inovația centrală a StarDist este prezicerea distanțelor radiale de-a lungul razelor fixe combinate cu probabilitatea obiectului pentru fiecare pixel, permițând reconstrucția precisă a formelor star-convexe. Această abordare segmentează fiabil obiectele apropiate care sunt dificil de separat folosind metode tradiționale bazate pe pixeli sau cutii de delimitare.
Dezvoltările recente au extins StarDist la imagini histopatologice, permițând nu doar segmentarea nucleelor, ci și clasificarea multi-clasă a obiectelor detectate. Metoda a obținut performanțe de top în provocări precum CoNIC (Colon Nuclei Identification and Counting).
Descărcare sau acces
Instalare & configurare
Instalează TensorFlow (versiunea 1.x sau 2.x) ca prerechizit pentru StarDist.
Folosește pip pentru a instala pachetul Python StarDist:
pip install stardistPentru napari:
pip install stardist-napariPentru QuPath: Instalează extensia StarDist prin tragerea fișierului .jar în QuPath.
Pentru ImageJ/Fiji: Folosește managerul de pluginuri încorporat sau instalarea manuală prin meniul de pluginuri.
Rularea segmentării
Încarcă un model pre-antrenat, normalizează imaginea și rulează predicția:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Deschide imaginea în napari, selectează pluginul StarDist, alege un model pre-antrenat sau personalizat și rulează predicția direct din GUI.
Folosește pluginul StarDist din meniul Plugins pentru a aplica un model pe stiva ta de imagini cu o interfață intuitivă.
După instalarea extensiei, rulează detectarea StarDist prin consola de scripting sau interfața grafică QuPath pentru analiza histopatologică.
Antrenare & ajustare fină
Crează imagini cu etichete ground-truth unde fiecare obiect este etichetat unic. Folosește unelte de adnotare precum LabKit, QuPath sau Fiji pentru a pregăti setul tău de date.
Folosește API-ul Python StarDist pentru a antrena un model nou sau pentru a ajusta fin unul existent cu datele tale adnotate personalizate.
Opțiuni de post-procesare
- Aplică suprimarea non-maximă (NMS) pentru a elimina formele candidate redundante
- Folosește StarDist OPP (Object Post-Processing) pentru a uni măștile în cazul formelor non-star-convexe
Limitări & considerații
- Presupunerea star-convexă poate să nu modeleze perfect formele foarte non-convexe sau foarte neregulate
- Complexitatea instalării: instalările personalizate necesită un compilator C++ compatibil pentru construirea extensiilor
- Accelerarea GPU depinde de versiunile compatibile TensorFlow, CUDA și cuDNN
- Unii utilizatori raportează probleme la rularea pluginului ImageJ din cauza configurației Java
Întrebări frecvente
StarDist funcționează cu o varietate de tipuri de imagini, inclusiv fluorescență, brightfield și histopatologie (de exemplu, H&E), datorită modelelor sale pre-antrenate flexibile și adaptabilității la diferite modalități de imagistică.
Da — StarDist suportă segmentarea instanțelor 3D folosind poliedre star-convexe pentru date volumetrice, extinzând capabilitățile 2D la analiza completă 3D.
Nu neapărat. Modelele pre-antrenate sunt disponibile și funcționează bine de obicei imediat. Totuși, pentru date specializate sau noi, adnotarea și antrenarea modelelor personalizate îmbunătățesc semnificativ acuratețea.
StarDist se integrează cu napari, ImageJ/Fiji și QuPath, permițând rularea segmentării dintr-un GUI fără programare. De asemenea, suportă utilizarea directă a API-ului Python pentru fluxuri de lucru avansate.
StarDist oferă funcții încorporate pentru calcularea metricilor comune de segmentare a instanțelor, inclusiv precizie, recall, scor F1 și calitate panoptică pentru a evalua performanța segmentării.
SAM
Informații despre aplicație
| Dezvoltator | Meta AI Research (FAIR) |
| Dispozitive suportate |
|
| Limbă și disponibilitate | Model fundamental open-source disponibil la nivel global; documentație în limba engleză |
| Preț | Gratuit — open-source sub licența Meta prin GitHub și integrarea MIB |
Prezentare generală
SAM (Segment Anything Model) este un model fundamental puternic de inteligență artificială creat de Meta care permite segmentarea interactivă și automată a practic oricărui obiect din imagini. Folosind prompturi precum puncte, casete de delimitare sau măști aproximative, SAM generează măști de segmentare fără a necesita reantrenare specifică sarcinii. În cercetarea în microscopie, flexibilitatea SAM a fost adaptată pentru segmentarea celulelor, detectarea organitelor și analiza histopatologică, oferind o soluție scalabilă pentru cercetătorii care au nevoie de un instrument de segmentare generalist, controlabil prin prompturi.
Introducere detaliată
Inițial antrenat de Meta pe peste 1 miliard de măști pe 11 milioane de imagini, SAM a fost conceput ca un model fundamental controlabil prin prompturi pentru segmentare cu performanță "zero-shot" pe domenii noi. În cercetarea imagisticii medicale, SAM a fost evaluat pentru segmentarea patologiei pe lame întregi, detectarea tumorilor și identificarea nucleelor celulare. Totuși, performanța sa pe instanțe dens grupate—cum ar fi nucleele celulare—este mixtă: chiar și cu prompturi extinse (de exemplu, 20 de clicuri sau casete), segmentarea zero-shot poate întâmpina dificultăți în imagini microscopice complexe.
Pentru a aborda această limitare, au apărut adaptări specifice domeniului:
- SAMCell — Ajustat fin pe seturi mari de date microscopice pentru segmentare zero-shot puternică pe diverse tipuri de celule, fără reantrenare per experiment
- μSAM — Reantrenat pe peste 17.000 de imagini microscopice adnotate manual pentru a îmbunătăți acuratețea pe structuri celulare mici
Caracteristici cheie
Interacțiune flexibilă folosind puncte, casete și măști pentru control precis.
Realizează segmentarea fără ajustare fină pe noi domenii de imagini.
Adaptabil pentru microscopie și histopatologie prin reantrenare few-shot sau bazată pe prompturi.
Disponibil în Microscopy Image Browser (MIB) cu suport pentru segmentare 3D și interpolată.
IDCC-SAM permite numărarea automată a celulelor în imunocitochimie fără adnotare manuală.
Descărcare sau acces
Ghid utilizator
- Deschideți Microscopy Image Browser și navigați la panoul de segmentare SAM
- Configurați interpretatorul Python și selectați între modelele SAM-1 sau SAM-2
- Pentru accelerare GPU, selectați "cuda" în mediul de execuție (recomandat pentru performanță optimă)
- Prompturi punctuale: Faceți clic pe un obiect pentru a defini o sămânță pozitivă; folosiți Shift + clic pentru extindere și Ctrl + clic pentru sămânțe negative
- Stack-uri 3D: Folosiți modul interactiv 3D—faceți clic pe o felie, derulați cu shift și interpolați sămânțele între felii
- Mod ajustare: Înlocuiți, adăugați, scădeți măști sau creați un strat nou după necesitate
- Folosiți opțiunea "Automatic everything" din panoul SAM-2 al MIB pentru a segmenta toate obiectele vizibile într-o regiune
- Revizuiți și rafinați măștile după segmentare, după necesitate
- Folosiți pipeline-uri de ajustare fină bazate pe prompturi (de ex., "All-in-SAM") pentru a genera adnotări la nivel de pixel din prompturi sparse ale utilizatorului
- Pentru numărarea celulelor, aplicați IDCC-SAM, care utilizează SAM într-un pipeline zero-shot cu post-procesare
- Pentru segmentare celulară de înaltă acuratețe, folosiți SAMCell, ajustat fin pe imagini microscopice de celule
Limitări și considerații
- Performanță zero-shot inconsistentă pe structuri dense sau suprapuse fără ajustare pe domeniu
- Calitatea segmentării depinde mult de proiectarea și strategia prompturilor
- GPU este puternic recomandat; inferența pe CPU este foarte lentă
- Întâmpină dificultăți cu imagini întregi de rezoluție foarte mare și structuri tisulare multi-scală
- Ajustarea fină sau adaptarea SAM pentru microscopie poate necesita competențe în învățare automată
Întrebări frecvente
Da—prin adaptări precum SAMCell, care ajustează fin SAM pe seturi de date microscopice special pentru sarcini de segmentare celulară.
Nu întotdeauna. Cu IDCC-SAM, puteți efectua numărarea celulelor în zero-shot fără adnotări manuale.
Folosiți ajustarea fină bazată pe prompturi (de ex., "All-in-SAM") sau versiuni preantrenate pentru microscopie precum μSAM, antrenat pe peste 17.000 de imagini microscopice adnotate.
Deși este posibil pe CPU, GPU este puternic recomandat pentru viteza practică a inferenței și segmentarea interactivă în timp real.
Da—integrarea SAM-2 în MIB suportă segmentarea 3D cu interpolarea sămânțelor între felii pentru analiza volumetrică.
AxonDeepSeg
| Dezvoltator | NeuroPoly Lab la Polytechnique Montréal și Université de Montréal |
| Platforme suportate |
|
| Limbaj | Documentație în limba engleză; instrument open-source utilizat la nivel global |
| Preț | Gratuit și open-source |
Prezentare generală
AxonDeepSeg este un instrument bazat pe inteligență artificială pentru segmentarea automată a axonilor și mielinei în imaginile microscopice. Folosind rețele neuronale convoluționale, oferă o segmentare precisă în trei clase (axon, mielină, fundal) pe multiple modalități de imagistică, inclusiv TEM, SEM și microscopie în câmp luminos. Prin automatizarea măsurătorilor morfometrice precum diametrul axonului, raportul g și grosimea mielinei, AxonDeepSeg eficientizează analiza cantitativă în cercetarea neuroștiințifică, reducând semnificativ timpul de adnotare manuală și îmbunătățind reproductibilitatea.
Caracteristici cheie
Modele gata de utilizare, optimizate pentru modalitățile de microscopie TEM, SEM și câmp luminos.
Clasificare precisă a regiunilor axon, mielină și fundal în imaginile microscopice.
Calcul automat al diametrului axonului, raportului g, grosimii mielinei și metricilor de densitate.
Integrarea cu interfața Napari permite rafinarea manuală a măștilor de segmentare pentru o acuratețe sporită.
Se integrează perfect în fluxuri de lucru personalizate pentru analiza la scară largă a țesutului neural.
Scripturi de testare cuprinzătoare asigură reproductibilitatea și rezultate fiabile ale segmentării.
Detalii Tehnice
Dezvoltat de NeuroPoly Lab, AxonDeepSeg utilizează învățarea profundă pentru a oferi segmentare de înaltă precizie în aplicații neuroștiințifice. Modelele pre-antrenate sunt disponibile pentru diferite modalități de microscopie, asigurând versatilitate în tehnicile de imagistică. Instrumentul se integrează cu Napari, permițând corecții interactive ale măștilor de segmentare, ceea ce îmbunătățește acuratețea pe seturi de date dificile. AxonDeepSeg calculează metrici morfometrici esențiali, susținând studii de mare volum privind structura și patologia țesutului neural. Cadru său bazat pe Python permite integrarea în fluxuri personalizate pentru analiza la scară largă a morfologiei axonilor și mielinei.
Descărcare sau Acces
Instalare și Configurare
Asigurați-vă că Python 3.8 sau o versiune ulterioară este instalată, apoi instalați AxonDeepSeg și Napari folosind pip:
pip install axondeepseg napariRulați scripturile de test furnizate pentru a confirma că toate componentele sunt instalate corect și funcționează.
Importați imaginile microscopice (TEM, SEM sau câmp luminos) în Napari sau în mediul dvs. Python.
Alegeți modelul pre-antrenat corespunzător modalității dvs. de imagistică și rulați segmentarea pentru a genera măști pentru axon și mielină.
Calculați automat măsurătorile morfometrice, inclusiv diametrul axonului, raportul g, densitatea și grosimea mielinei, apoi exportați rezultatele în format CSV.
Folosiți interfața Napari pentru a ajusta manual măștile de segmentare acolo unde este necesar, combinând sau ștergând măști pentru o acuratețe îmbunătățită.
Considerații Importante
- Performanța poate scădea pe modalități noi sau neantrenate de imagistică
- Corecții manuale pot fi necesare pentru regiuni dificile sau complexe
- GPU este recomandat pentru procesarea rapidă a seturilor mari de date; procesarea pe CPU este de asemenea suportată
Întrebări frecvente
AxonDeepSeg suportă TEM (Microscopie Electronică de Transmisie), SEM (Microscopie Electronică de Scanare) și microscopie în câmp luminos, cu modele pre-antrenate optimizate pentru fiecare modalitate.
Da, AxonDeepSeg este complet gratuit și open-source, disponibil pentru utilizare academică și comercială.
Da, AxonDeepSeg calculează automat diametrul axonului, raportul g, grosimea mielinei și metrici de densitate din imaginile segmentate.
GPU este recomandat pentru segmentarea rapidă a seturilor mari de date, dar procesarea pe CPU este de asemenea suportată pentru analize mai mici.
Da, integrarea cu interfața Napari permite corecții interactive și rafinarea măștilor de segmentare pentru o acuratețe mai mare în regiunile dificile.
Ilastik
| Dezvoltator | Echipa Ilastik de la Laboratorul European de Biologie Moleculară (EMBL) și parteneri academici asociați |
| Platforme suportate |
|
| Limbă | Engleză |
| Preț | Gratuit și open-source |
Prezentare generală
Ilastik este un instrument puternic, bazat pe inteligență artificială, pentru segmentarea interactivă a imaginilor, clasificare și analiză a datelor de microscopie. Folosind tehnici de învățare automată precum clasificatorii Random Forest, permite cercetătorilor să segmenteze pixeli, să clasifice obiecte, să urmărească celulele în timp și să efectueze numărarea densității în seturi de date 2D și 3D. Cu o interfață intuitivă și feedback în timp real, Ilastik este accesibil oamenilor de știință fără expertiză în programare și este larg adoptat în biologia celulară, neuroștiințe și imagistica biomedicală.
Caracteristici cheie
Feedback în timp real pe măsură ce adnotați regiuni reprezentative pentru rezultate instantanee de segmentare.
Categorizați structurile segmentate pe baza caracteristicilor morfologice și de intensitate.
Urmăriți mișcarea și diviziunea celulelor în experimente de microscopie time-lapse 2D și 3D.
Quantificați regiunile aglomerate fără segmentarea explicită a obiectelor individuale.
Segmentare semi-automată pentru volume 3D complexe cu interacțiune intuitivă.
Procesați automat mai multe imagini folosind modul headless din linia de comandă.
Descărcare
Ghid de început
Descărcați Ilastik pentru sistemul dumneavoastră de operare de pe site-ul oficial. Pachetul include toate dependențele Python necesare, așadar urmați instrucțiunile de instalare pentru platforma dvs.
Deschideți Ilastik și alegeți fluxul de lucru pentru analiză: Clasificare pixel, Clasificare obiect, Urmărire sau Numărare densitate. Încărcați setul de imagini, care poate include imagini multi-canal, 3D sau time-lapse.
Marcați câțiva pixeli sau obiecte reprezentative în imaginile dvs. Clasificatorul Random Forest al Ilastik învață din aceste adnotări și prezice automat etichetele pentru întregul set de date.
Aplicați modelul antrenat pentru a segmenta sau clasifica întregul set de date. Exportați rezultatele ca imagini etichetate, hărți de probabilitate sau tabele cantitative pentru analiza și vizualizarea ulterioară.
Folosiți modul headless al Ilastik pentru a procesa automat mai multe imagini fără intervenție manuală, ideal pentru fluxuri de analiză la scară largă.
Limitări și considerații
- Etichetarea interactivă poate fi consumatoare de timp pentru seturi de date foarte mari
- Acuratețea depinde de calitatea și reprezentativitatea adnotărilor utilizatorului
- Cerințe de memorie — seturile de date cu rezoluție foarte înaltă sau multi-gigabyte pot necesita memorie RAM semnificativă
- Date complexe — clasificatorii Random Forest pot avea performanțe mai slabe comparativ cu rețelele neuronale profunde pentru date de imagistică foarte variabile sau complexe
Întrebări frecvente
Da, Ilastik suportă complet volume 3D și experimente time-lapse pentru segmentare, urmărire și analiză cantitativă pe mai multe momente în timp.
Da, Ilastik este complet gratuit și open-source, disponibil pentru toți utilizatorii fără restricții de licențiere.
Nu, Ilastik oferă o interfață grafică intuitivă cu feedback în timp real, fiind accesibil cercetătorilor fără expertiză în programare. Utilizatorii avansați pot folosi și procesarea în loturi din linia de comandă.
Da, fluxul de lucru dedicat urmării permite analiza mișcării și diviziunii celulare în seturi de date time-lapse 2D și 3D cu urmărire automată a liniei celulare.
Rezultatele segmentării pot fi exportate ca imagini etichetate, hărți de probabilitate sau tabele cantitative, permițând integrarea facilă cu instrumente de analiză și software de vizualizare ulterioară.
Aceste instrumente acoperă niveluri de la începători la experți. Multe sunt gratuite și open-source, facilitând fluxuri de lucru AI reproducibile și partajabile în comunitatea de cercetare.
Provocări și direcții viitoare
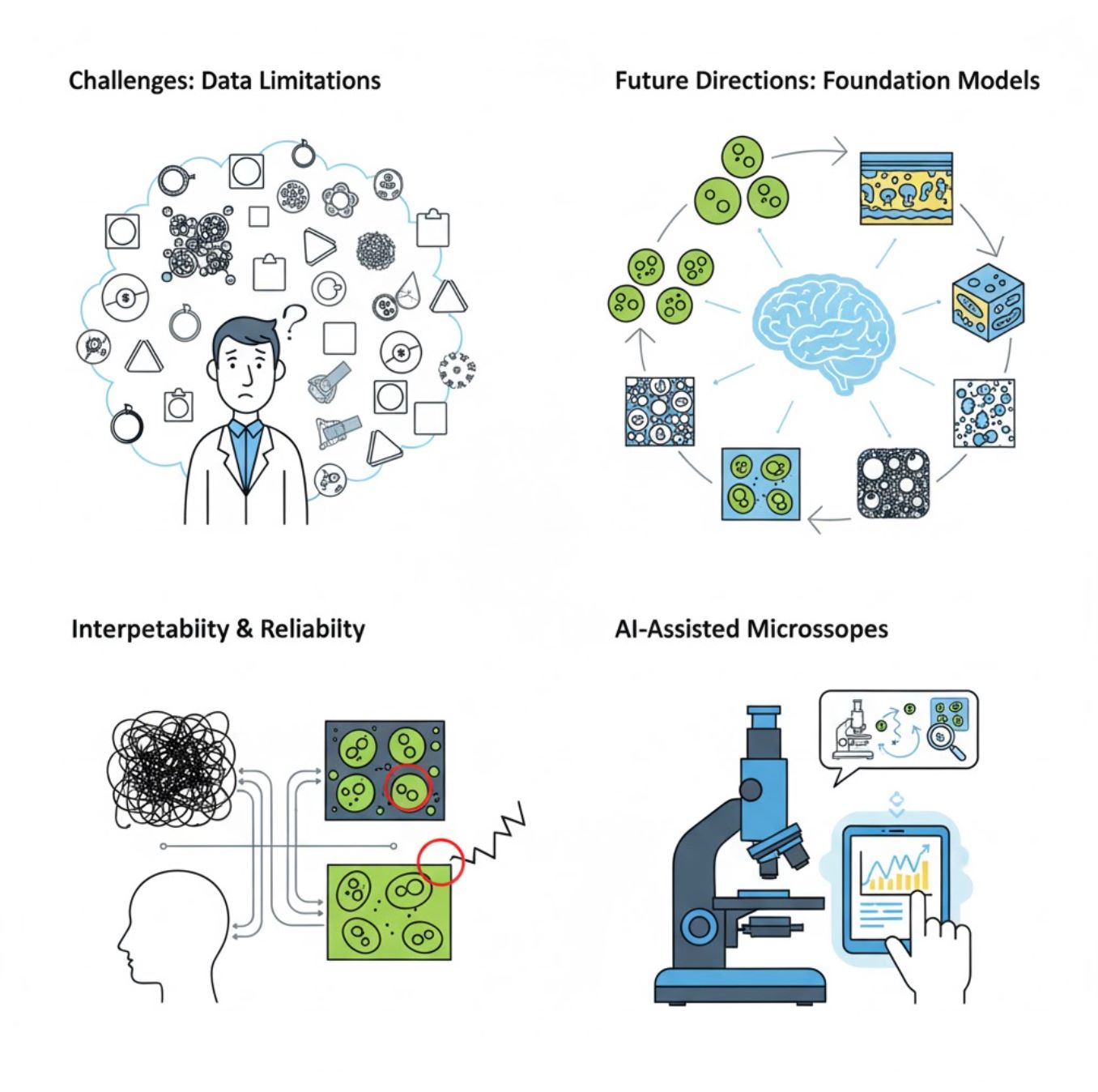
Provocări actuale
Tendințe emergente
Modele fundamentale de viziune
Sistemele AI de nouă generație promit să reducă nevoia de antrenament specific pe sarcini.
- Modele precum SAM și sisteme bazate pe CLIP
- Un singur AI gestionează multe sarcini de microscopie
- Implementare și adaptare mai rapidă
Microscoape asistate de AI
Sisteme de microscopie complet autonome și inteligente devin realitate.
- Control prin limbaj natural via LLM-uri
- Bucle de feedback complet automate
- Democratizează accesul la microscopie avansată
Concluzii cheie
- Inteligența artificială transformă rapid procesarea imaginilor microscopice cu precizie și automatizare îmbunătățite
- Învățarea profundă depășește învățarea automată tradițională pe imagini microscopice complexe și variabile
- CNN-urile învață automat caracteristici ierarhice din pixeli neprelucrați pentru o analiză robustă
- Aplicațiile cheie includ segmentarea, clasificarea, urmărirea, reducerea zgomotului și achiziția automată
- Succesul depinde de date de calitate și validare atentă de către experți
- Modelele fundamentale de viziune și microscoapele asistate de AI reprezintă viitorul domeniului
Cu progrese continue și eforturi comunitare (instrumente open-source, seturi de date partajate), AI va deveni tot mai mult o parte esențială a „ochiului” microscopului, ajutând oamenii de știință să vadă ceea ce este invizibil.







No comments yet. Be the first to comment!