IA no Processamento de Imagens de Microscópio
A IA está revolucionando o processamento de imagens de microscópio com capacidades poderosas, como segmentação precisa, redução de ruído, super-resolução e aquisição automática de imagens. Este artigo destaca ferramentas essenciais de IA e tendências emergentes na pesquisa científica.
As técnicas de IA estão revolucionando a microscopia ao otimizar a aquisição de imagens e automatizar a análise. Em microscópios inteligentes modernos, módulos de IA podem ajustar parâmetros de imagem em tempo real (por exemplo, foco, iluminação) para minimizar o fotobranqueamento e melhorar o sinal. Enquanto isso, algoritmos de aprendizado profundo podem filtrar dados complexos de imagens para extrair insights biológicos ocultos e até mesmo vincular imagens a outros dados (por exemplo, genômica).
Métodos de IA: Aprendizado de Máquina vs Aprendizado Profundo
Os métodos de IA variam desde o clássico aprendizado de máquina (ML) até o moderno aprendizado profundo (DL). Cada abordagem tem forças e limitações distintas:
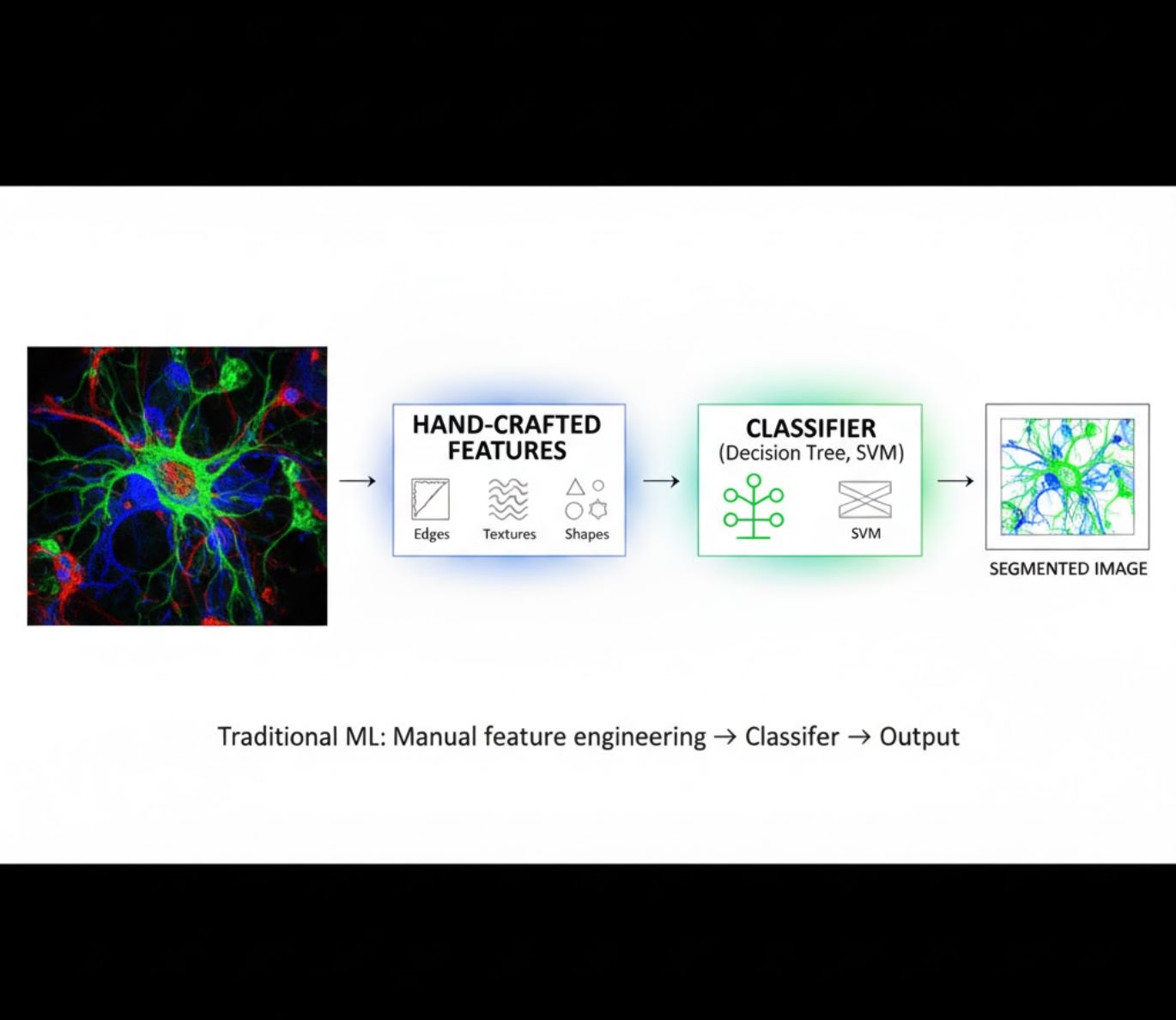
Características Criadas Manualmente
- Pesquisadores criam manualmente características de imagem (bordas, texturas, formas)
- Características são fornecidas a classificadores (árvores de decisão, SVM)
- Rápido para treinar
- Dificuldade com imagens complexas ou ruidosas
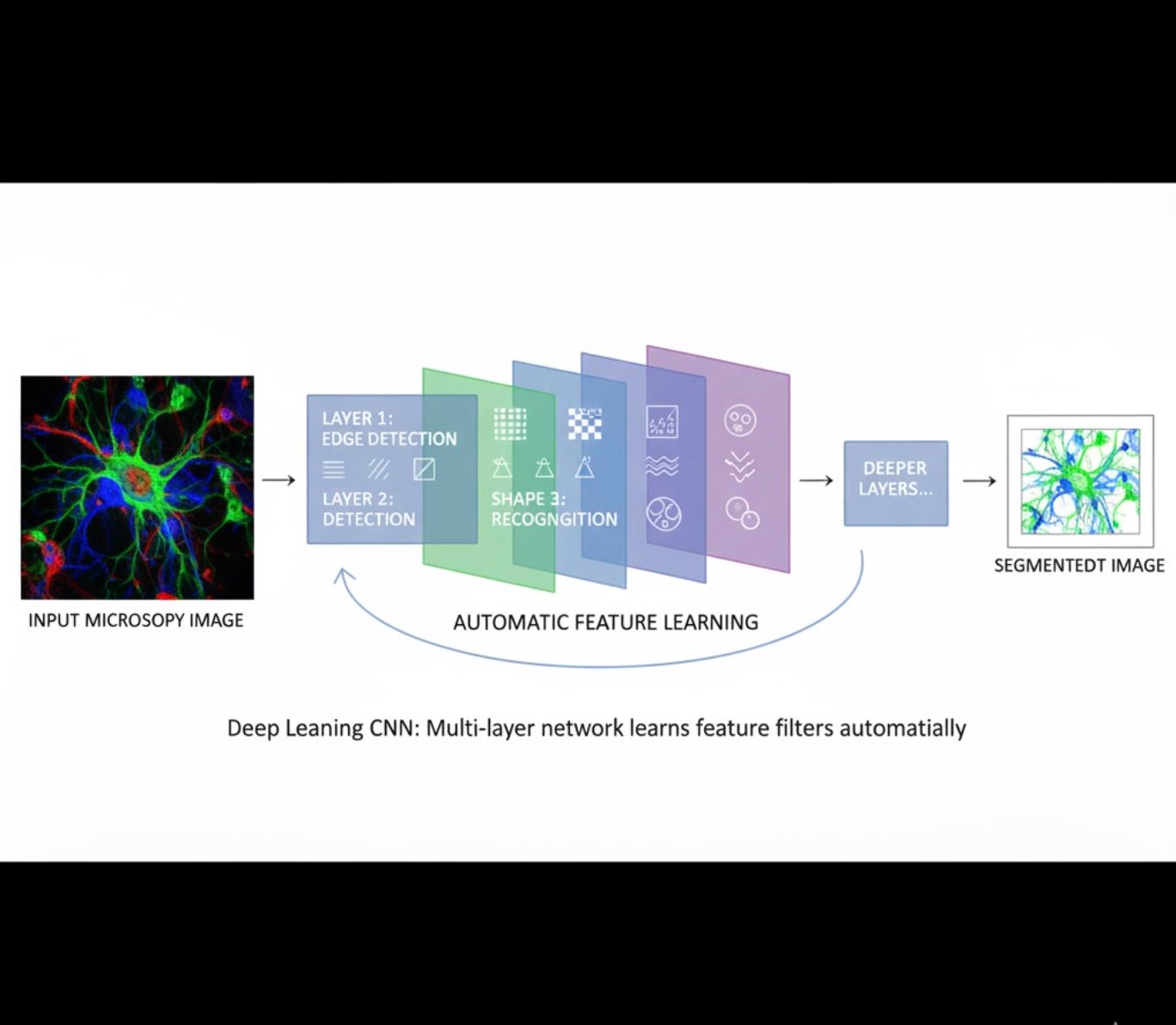
Aprendizado Automático de Características
- Redes neurais multicamadas (CNNs) aprendem características automaticamente
- Aprendizado de ponta a ponta a partir de pixels brutos
- Muito mais robusto a variações
- Captura texturas e estruturas intrincadas de forma confiável
Como funcionam as CNNs: Redes neurais convolucionais aplicam filtros sucessivos às imagens de microscopia, aprendendo a detectar padrões simples (bordas) nas camadas iniciais e estruturas complexas (formas celulares, texturas) nas camadas mais profundas. Esse aprendizado hierárquico torna o DL excepcionalmente robusto mesmo quando os perfis de intensidade variam significativamente.
Comparação Visual: Fluxos ML vs DL
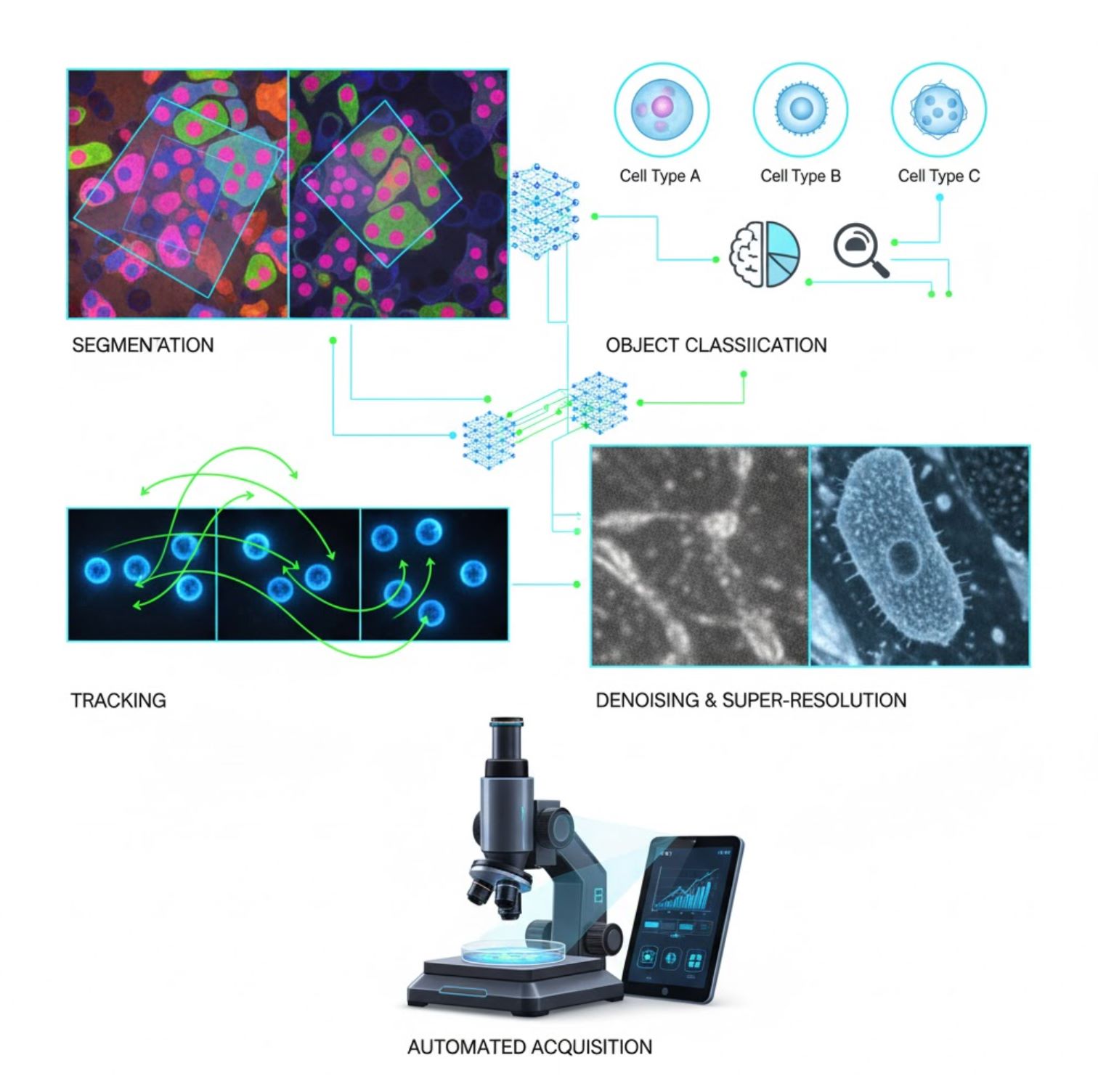
Principais Aplicações de IA na Microscopia
A IA está agora incorporada em muitas tarefas de processamento de imagem ao longo do fluxo de trabalho da microscopia:
Segmentação
Divisão das imagens em regiões (por exemplo, identificando cada célula ou núcleo). Redes profundas como U-Net se destacam nessa tarefa.
- Segmentação semântica: Rótulos de classe por pixel
- Segmentação por instância: Separação de objetos individuais
- Alta precisão em imagens lotadas ou escuras
- Modelos fundacionais de visão (ex.: μSAM) agora adaptados para microscopia
Classificação de Objetos
Após a segmentação, a IA classifica cada objeto com alta precisão.
- Identificação do tipo celular
- Determinação do estágio mitótico
- Detecção de indicadores patológicos
- Distingue fenótipos sutis difíceis de quantificar manualmente
Rastreamento
Na microscopia em lapso de tempo, a IA rastreia células ou partículas entre quadros com precisão sem precedentes.
- Aprendizado profundo melhora dramaticamente a precisão do rastreamento
- Permite análise confiável de células em movimento
- Captura processos biológicos dinâmicos
Redução de Ruído & Super-Resolução
Modelos de IA melhoram a qualidade da imagem removendo ruído e desfoque.
- Modelos profundos informados pela física aprendem a óptica do microscópio
- Reconstrução de imagens mais nítidas e sem artefatos
- Maior resolução com menos artefatos comparado a métodos tradicionais
Aquisição Automática
A IA orienta o próprio microscópio em tempo real.
- Análise de imagens ao vivo para decisões inteligentes
- Ajuste automático de foco e varredura de áreas de interesse
- Reduz fototoxicidade e economiza tempo
- Permite experimentos de imagem adaptativos e de alto rendimento
Ferramentas Populares de IA no Processamento de Imagens de Microscópio
<ITEM_DESCRIPTION>>>Um ecossistema robusto de ferramentas que apoia a inteligência artificial em microscopia. Pesquisadores desenvolveram tanto softwares de uso geral quanto especializados, muitos deles de código aberto:
Cellpose
| Desenvolvedores | Carsen Stringer e Marius Pachitariu (grupo de pesquisa MouseLand) |
| Plataformas Suportadas |
Requer Python (instalação via pip/conda). GUI disponível apenas no desktop. |
| Suporte de Idioma | Documentação em inglês; amplamente adotado em laboratórios de pesquisa ao redor do mundo |
| Modelo de Preço | Gratuito e open-source sob licença BSD-3-Clause |
Visão Geral
Cellpose é uma ferramenta avançada de segmentação baseada em aprendizado profundo, projetada para imagens de microscopia. Como um algoritmo generalista, segmenta com precisão diversos tipos celulares (núcleos, citoplasma, etc.) em diferentes modalidades de imagem sem necessidade de re-treinamento do modelo. Com capacidades de intervenção humana, pesquisadores podem refinar resultados, adaptar o modelo aos seus dados e aplicar o sistema em fluxos de trabalho 2D e 3D.
Principais Características
Funciona imediatamente para uma ampla variedade de tipos celulares, corantes e modalidades de imagem sem treinamento personalizado.
Suporta pilhas 3D completas usando uma abordagem "2.5D" que reutiliza modelos 2D para dados volumétricos.
Corrija manualmente os resultados da segmentação e re-treine o modelo com seus dados personalizados para maior precisão.
Acesso via API Python, interface de linha de comando ou interface gráfica para fluxos de trabalho flexíveis.
Capacidades de remoção de ruído, desfoque e aumento de resolução para melhorar a qualidade da imagem antes da segmentação.
Download ou Acesso
Contexto Técnico
Cellpose foi apresentado em um estudo seminal por Stringer, Wang, Michaelos e Pachitariu, treinado em um conjunto de dados grande e altamente variado contendo mais de 70.000 objetos segmentados. Essa diversidade permite que o modelo generalize entre formas, tamanhos celulares e configurações de microscopia, reduzindo significativamente a necessidade de treinamento personalizado na maioria dos casos. Para dados 3D, Cellpose reutiliza inteligentemente seu modelo 2D em uma abordagem "2.5D", evitando a necessidade de dados de treinamento totalmente anotados em 3D, enquanto ainda entrega segmentação volumétrica. O Cellpose 2.0 introduziu o re-treinamento com intervenção humana, permitindo que usuários corrijam manualmente previsões e re-treinarem com suas próprias imagens para melhor desempenho em conjuntos de dados específicos.
Instalação & Configuração
Configure um ambiente Python usando conda:
conda create -n cellpose python=3.10Ative o ambiente e instale o Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposePrimeiros Passos
Modo GUI
- Inicie a GUI executando:
python -m cellpose - Arraste e solte arquivos de imagem (
.tif,.png, etc.) na interface - Selecione o tipo de modelo (ex.: "cyto" para citoplasma ou "nuclei" para núcleos)
- Defina o diâmetro estimado da célula ou deixe o Cellpose calibrar automaticamente
- Clique para iniciar a segmentação e visualizar os resultados
Modo API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Refinar & Re-treinar
- Após gerar máscaras, corrija a segmentação na GUI mesclando ou excluindo máscaras manualmente
- Use funções de treinamento integradas para re-treinar com exemplos corrigidos
- Melhore o desempenho do modelo no seu conjunto de dados específico
Processar Dados 3D
- Carregue um TIFF multi-Z ou pilha volumétrica
- Use a flag
--Zstackna GUI ou API para processar como 3D - Opcionalmente, refine os fluxos 3D via suavização ou parâmetros especializados para melhor segmentação
Limitações & Considerações
- Compromisso na Generalidade do Modelo: Embora o modelo generalista funcione amplamente, formas celulares ou condições de imagem muito incomuns podem exigir re-treinamento.
- Esforço de Anotação: O treinamento com intervenção humana requer correções manuais, que podem ser demoradas para grandes conjuntos de dados.
- Complexidade de Instalação: A instalação da GUI pode exigir uso de linha de comando, ambientes conda e gerenciamento de dependências Python — nem sempre simples para não programadores.
- Apenas Desktop: Cellpose é projetado para uso em desktop; não há aplicativos nativos para Android ou iOS.
Perguntas Frequentes
Não — Cellpose fornece modelos generalistas pré-treinados que geralmente funcionam bem sem re-treinamento. Contudo, para resultados ótimos em dados especiais ou incomuns, você pode anotar e re-treinar usando os recursos de intervenção humana.
Sim — ele suporta 3D reutilizando seu modelo 2D (chamado "2.5D"), e você pode processar pilhas volumétricas via GUI ou API.
GPU é altamente recomendada para inferência e treinamento mais rápidos, especialmente em conjuntos de dados grandes ou 3D, mas o Cellpose pode rodar em máquinas apenas com CPU com desempenho mais lento.
Na GUI, defina manualmente o diâmetro estimado da célula ou deixe o Cellpose calibrar automaticamente. Você pode refinar os resultados e re-treinar se a segmentação não estiver ideal.
Sim — versões mais recentes (Cellpose 3) incluem modelos de restauração de imagem para remoção de ruído, desfoque e aumento de resolução para melhorar a qualidade da segmentação antes do processamento.
StarDist
| Desenvolvedores | Uwe Schmidt, Martin Weigert, Coleman Broaddus e Gene Myers |
| Plataformas Suportadas |
|
| Suporte de Idioma | Projeto open-source com documentação e comunidade principalmente em inglês |
| Modelo de Preço | Gratuito e open source. Licenciado sob BSD-3-Clause |
Visão Geral
StarDist é uma ferramenta de aprendizado profundo para segmentação de instâncias em imagens de microscopia. Representa cada objeto (como núcleos celulares) como um polígono estelar convexo em 2D ou poliedro em 3D, permitindo detecção e separação precisas de objetos densamente agrupados ou sobrepostos. Com sua arquitetura robusta, StarDist é amplamente usado para segmentação automatizada de células e núcleos em microscopia de fluorescência, histopatologia e outras aplicações de análise bioimagem.
Principais Funcionalidades
Segmentação de instâncias altamente precisa usando polígonos estelares convexos (2D) e poliedros (3D) para detecção confiável de objetos.
Modelos dedicados para imagens 2D e dados volumétricos 3D para análise microscópica abrangente.
Modelos prontos para uso para núcleos fluorescentes, histologia corada com H&E e outros cenários comuns de imagem.
Classifique objetos detectados em classes distintas (ex.: diferentes tipos celulares) em uma única execução de segmentação.
Integração fluida com ImageJ/Fiji, QuPath e napari para fluxos de trabalho acessíveis via GUI.
Avaliação abrangente da segmentação de instâncias incluindo precisão, recall, F1 score e qualidade panóptica.
Contexto Técnico
Originalmente apresentado em artigo MICCAI 2018, a inovação central do StarDist é a predição de distâncias radiais ao longo de raios fixos combinada com a probabilidade do objeto para cada pixel, permitindo reconstrução precisa de formas estelares convexas. Essa abordagem segmenta confiavelmente objetos muito próximos que são difíceis de separar com métodos tradicionais baseados em pixels ou caixas delimitadoras.
Desenvolvimentos recentes expandiram o StarDist para imagens de histopatologia, permitindo não só segmentação de núcleos, mas também classificação multiclasse dos objetos detectados. O método alcançou desempenho de destaque em desafios como o CoNIC (Colon Nuclei Identification and Counting).
Download ou Acesso
Instalação & Configuração
Instale o TensorFlow (versão 1.x ou 2.x) como pré-requisito para o StarDist.
Use pip para instalar o pacote Python StarDist:
pip install stardistPara napari:
pip install stardist-napariPara QuPath: Instale a extensão StarDist arrastando o arquivo .jar para dentro do QuPath.
Para ImageJ/Fiji: Use o gerenciador de plugins embutido ou instalação manual via menu de plugins.
Executando a Segmentação
Carregue um modelo pré-treinado, normalize sua imagem e execute a predição:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Abra sua imagem no napari, selecione o plugin StarDist, escolha um modelo pré-treinado ou personalizado e execute a predição diretamente pela GUI.
Use o plugin StarDist no menu Plugins para aplicar um modelo na sua pilha de imagens com uma interface intuitiva.
Após instalar a extensão, execute a detecção StarDist via console de scripts ou interface gráfica do QuPath para análise histopatológica.
Treinamento & Ajuste Fino
Crie imagens de rótulo de referência onde cada objeto é rotulado de forma única. Use ferramentas de anotação como LabKit, QuPath ou Fiji para preparar seu conjunto de dados.
Use a API Python do StarDist para treinar um modelo novo ou ajustar um existente com seus dados anotados personalizados.
Opções de Pós-Processamento
- Aplique supressão de não-máximos (NMS) para eliminar formas candidatas redundantes
- Use StarDist OPP (Pós-Processamento de Objetos) para mesclar máscaras de formas não estelares convexas
Limitações & Considerações
- A suposição estelar convexa pode não modelar perfeitamente formas altamente não convexas ou muito irregulares
- Complexidade na instalação: instalações personalizadas requerem compilador C++ compatível para construir extensões
- Aceleração por GPU depende de versões compatíveis do TensorFlow, CUDA e cuDNN
- Alguns usuários relatam problemas ao executar o plugin ImageJ devido à configuração do Java
Perguntas Frequentes
StarDist funciona com vários tipos de imagem, incluindo fluorescência, campo claro e histopatologia (ex.: H&E), graças aos seus modelos pré-treinados flexíveis e adaptabilidade a diferentes modalidades de imagem.
Sim — o StarDist suporta segmentação de instâncias 3D usando poliedros estelares convexos para dados volumétricos, estendendo as capacidades 2D para análise completa em 3D.
Não necessariamente. Modelos pré-treinados estão disponíveis e frequentemente funcionam bem imediatamente. Contudo, para dados especializados ou novos, anotar e treinar modelos personalizados melhora significativamente a precisão.
StarDist integra-se com napari, ImageJ/Fiji e QuPath, permitindo executar segmentação via GUI sem programação. Também suporta uso direto da API Python para fluxos avançados.
StarDist oferece funções integradas para calcular métricas comuns de segmentação de instâncias, incluindo precisão, recall, F1 score e qualidade panóptica para avaliar o desempenho da segmentação.
SAM
Informações da Aplicação
| Desenvolvedor | Meta AI Research (FAIR) |
| Dispositivos Suportados |
|
| Idioma & Disponibilidade | Modelo fundamental open-source disponível globalmente; documentação em inglês |
| Preço | Gratuito — open-source sob licença da Meta via GitHub e integração no MIB |
Visão Geral
SAM (Segment Anything Model) é um poderoso modelo fundamental de IA criado pela Meta que permite segmentação interativa e automática de praticamente qualquer objeto em imagens. Usando prompts como pontos, caixas delimitadoras ou máscaras aproximadas, o SAM gera máscaras de segmentação sem necessidade de re-treinamento específico para a tarefa. Na pesquisa em microscopia, a flexibilidade do SAM foi adaptada para segmentação de células, detecção de organelas e análise histopatológica, oferecendo uma solução escalável para pesquisadores que precisam de uma ferramenta de segmentação geral e acionada por prompts.
Introdução Detalhada
Originalmente treinado pela Meta em mais de 1 bilhão de máscaras em 11 milhões de imagens, o SAM foi projetado como um modelo fundamental acionável para segmentação com desempenho "zero-shot" em domínios novos. Na pesquisa em imagens médicas, o SAM foi avaliado para segmentação de lâminas inteiras em patologia, detecção de tumores e identificação de núcleos celulares. Contudo, seu desempenho em instâncias densamente agrupadas—como núcleos celulares—é variável: mesmo com prompts extensos (ex.: 20 cliques ou caixas), a segmentação zero-shot pode apresentar dificuldades em imagens complexas de microscopia.
Para superar essa limitação, surgiram adaptações específicas para domínios:
- SAMCell — Ajustado em grandes conjuntos de dados de microscopia para segmentação zero-shot robusta em diversos tipos celulares sem necessidade de re-treinamento por experimento
- μSAM — Re-treinado em mais de 17.000 imagens de microscopia anotadas manualmente para melhorar a precisão em estruturas celulares pequenas
Principais Funcionalidades
Interação flexível usando pontos, caixas e máscaras para controle preciso.
Realiza segmentação sem ajuste fino em novos domínios de imagem.
Adaptável para microscopia e histopatologia via re-treinamento few-shot ou baseado em prompts.
Disponível no Microscopy Image Browser (MIB) com suporte para segmentação 3D e interpolada.
IDCC-SAM permite contagem automática de células em imunocitoquímica sem necessidade de anotação manual.
Download ou Acesso
Guia do Usuário
- Abra o Microscopy Image Browser e navegue até o painel de segmentação SAM
- Configure o interpretador Python e selecione entre os modelos SAM-1 ou SAM-2
- Para aceleração por GPU, selecione "cuda" no ambiente de execução (recomendado para desempenho ideal)
- Prompts por ponto: Clique em um objeto para definir uma semente positiva; use Shift + clique para expandir e Ctrl + clique para sementes negativas
- Pilhas 3D: Use o modo interativo 3D—clique em um corte, role com shift e interpole sementes entre cortes
- Modo de ajuste: Substitua, adicione, subtraia máscaras ou crie uma nova camada conforme necessário
- Use a opção "Automatic everything" do MIB no painel SAM-2 para segmentar todos os objetos visíveis em uma região
- Revise e refine as máscaras após a segmentação conforme necessário
- Use pipelines de ajuste fino baseados em prompts (ex.: "All-in-SAM") para gerar anotações em nível de pixel a partir de prompts esparsos do usuário
- Para contagem celular, aplique o IDCC-SAM, que usa o SAM em pipeline zero-shot com pós-processamento
- Para segmentação celular de alta precisão, use o SAMCell, ajustado em imagens de células de microscopia
Limitações & Considerações
- Desempenho zero-shot inconsistente em estruturas densas ou sobrepostas sem ajuste de domínio
- Qualidade da segmentação depende fortemente do design e estratégia do prompt
- GPU fortemente recomendada; inferência por CPU é muito lenta
- Dificuldades com imagens de lâminas inteiras em altíssima resolução e estruturas teciduais multi-escala
- Ajuste fino ou adaptação do SAM para microscopia pode requerer proficiência em aprendizado de máquina
Perguntas Frequentes
Sim—através de adaptações como o SAMCell, que ajusta o SAM em conjuntos de dados de microscopia especificamente para tarefas de segmentação celular.
Nem sempre. Com o IDCC-SAM, você pode realizar contagem celular zero-shot sem anotações manuais.
Use ajuste fino baseado em prompts (ex.: "All-in-SAM") ou versões pré-treinadas para microscopia como o μSAM, treinado em mais de 17.000 imagens anotadas de microscopia.
Embora seja possível usar CPU, GPU é altamente recomendada para velocidade prática de inferência e segmentação interativa em tempo real.
Sim—a integração do SAM-2 no MIB suporta segmentação 3D com interpolação de sementes entre cortes para análise volumétrica.
AxonDeepSeg
| Desenvolvedor | NeuroPoly Lab na Polytechnique Montréal e Université de Montréal |
| Plataformas Suportadas |
|
| Idioma | Documentação em inglês; ferramenta open-source usada globalmente |
| Preço | Gratuito e open-source |
Visão Geral
AxonDeepSeg é uma ferramenta baseada em IA para segmentação automática de axônios e mielina em imagens de microscopia. Utilizando redes neurais convolucionais, oferece segmentação precisa de três classes (axônio, mielina, fundo) em múltiplas modalidades de imagem, incluindo TEM, SEM e microscopia de campo claro. Ao automatizar medições morfométricas como diâmetro do axônio, razão g e espessura da mielina, AxonDeepSeg agiliza a análise quantitativa em pesquisas em neurociência, reduzindo significativamente o tempo de anotação manual e melhorando a reprodutibilidade.
Principais Funcionalidades
Modelos prontos para uso otimizados para as modalidades de microscopia TEM, SEM e campo claro.
Classificação precisa das regiões de axônio, mielina e fundo em imagens de microscopia.
Cálculo automático do diâmetro do axônio, razão g, espessura da mielina e métricas de densidade.
Integração com GUI Napari permite refinamento manual das máscaras de segmentação para maior precisão.
Integração fluida em pipelines personalizados para análise em larga escala de tecido neural.
Scripts de teste abrangentes garantem reprodutibilidade e resultados confiáveis de segmentação.
Detalhes Técnicos
Desenvolvido pelo NeuroPoly Lab, AxonDeepSeg utiliza aprendizado profundo para oferecer segmentação de alta precisão para aplicações neurocientíficas. Modelos pré-treinados estão disponíveis para diferentes modalidades de microscopia, garantindo versatilidade entre técnicas de imagem. A ferramenta integra-se ao Napari, permitindo correções interativas das máscaras de segmentação, o que aumenta a precisão em conjuntos de dados desafiadores. AxonDeepSeg calcula métricas morfométricas chave, apoiando estudos de alto rendimento sobre a estrutura e patologia do tecido neural. Seu framework baseado em Python possibilita integração em pipelines personalizados para análise em larga escala da morfologia de axônios e mielina.
Download ou Acesso
Instalação & Configuração
Certifique-se de que o Python 3.8 ou superior está instalado, depois instale AxonDeepSeg e Napari usando pip:
pip install axondeepseg napariExecute os scripts de teste fornecidos para confirmar que todos os componentes estão instalados e funcionando corretamente.
Importe imagens de microscopia (TEM, SEM ou campo claro) no Napari ou no seu ambiente Python.
Escolha o modelo pré-treinado adequado para sua modalidade de imagem e execute a segmentação para gerar máscaras de axônio e mielina.
Calcule automaticamente medições morfométricas incluindo diâmetro do axônio, razão g, densidade e espessura da mielina, depois exporte os resultados em formato CSV.
Use a GUI Napari para ajustar manualmente as máscaras de segmentação onde necessário, mesclando ou excluindo máscaras para maior precisão.
Considerações Importantes
- O desempenho pode diminuir em modalidades de imagem novas ou não treinadas
- Correções manuais podem ser necessárias para regiões desafiadoras ou complexas
- GPU é recomendada para processamento mais rápido de grandes conjuntos de dados; processamento em CPU também é suportado
Perguntas Frequentes
AxonDeepSeg suporta TEM (Microscopia Eletrônica de Transmissão), SEM (Microscopia Eletrônica de Varredura) e microscopia de campo claro com modelos pré-treinados otimizados para cada modalidade.
Sim, AxonDeepSeg é completamente gratuito e open-source, disponível para uso acadêmico e comercial.
Sim, AxonDeepSeg calcula automaticamente diâmetro do axônio, razão g, espessura da mielina e métricas de densidade a partir das imagens segmentadas.
GPU é recomendada para segmentação mais rápida de grandes conjuntos de dados, mas o processamento em CPU também é suportado para análises menores.
Sim, a integração com a GUI Napari permite correções interativas e refinamento das máscaras de segmentação para maior precisão em regiões desafiadoras.
Ilastik
| Desenvolvedor | Equipe Ilastik do Laboratório Europeu de Biologia Molecular (EMBL) e parceiros acadêmicos associados |
| Plataformas Suportadas |
|
| Idioma | Inglês |
| Preço | Gratuito e de código aberto |
Visão Geral
O Ilastik é uma ferramenta poderosa, orientada por IA, para segmentação interativa, classificação e análise de imagens de microscopia. Utilizando técnicas de aprendizado de máquina como classificadores Random Forest, permite que pesquisadores segmentem pixels, classifiquem objetos, rastreiem células ao longo do tempo e realizem contagem de densidade em conjuntos de dados 2D e 3D. Com sua interface intuitiva e feedback em tempo real, o Ilastik é acessível a cientistas sem experiência em programação e é amplamente adotado em biologia celular, neurociência e imagem biomédica.
Principais Funcionalidades
Feedback em tempo real enquanto você anota regiões representativas para resultados instantâneos de segmentação.
Categorize estruturas segmentadas com base em características morfológicas e de intensidade.
Rastreie o movimento e a divisão celular em experimentos de microscopia em lapso de tempo 2D e 3D.
Quantifique regiões densamente povoadas sem segmentação explícita de objetos individuais.
Segmentação semi-automática para volumes 3D complexos com interação intuitiva.
Processe múltiplas imagens automaticamente usando o modo headless via linha de comando.
Download
Guia de Início Rápido
Baixe o Ilastik para seu sistema operacional no site oficial. O pacote inclui todas as dependências Python necessárias, então siga as instruções de instalação para sua plataforma.
Abra o Ilastik e escolha seu fluxo de análise: Classificação de Pixels, Classificação de Objetos, Rastreamento ou Contagem de Densidade. Carregue seu conjunto de imagens, que pode incluir múltiplos canais, 3D ou imagens em lapso de tempo.
Rotule alguns pixels ou objetos representativos em suas imagens. O classificador Random Forest do Ilastik aprende com essas anotações e prevê automaticamente os rótulos para todo o conjunto de dados.
Aplique o modelo treinado para segmentar ou classificar seu conjunto completo. Exporte os resultados como imagens rotuladas, mapas de probabilidade ou tabelas quantitativas para análise e visualização posteriores.
Use o modo headless do Ilastik para processar automaticamente múltiplas imagens sem intervenção manual, ideal para pipelines de análise em larga escala.
Limitações e Considerações
- Rotulagem interativa pode ser demorada para conjuntos de dados muito grandes
- Acurácia depende da qualidade e representatividade das anotações do usuário
- Requisitos de memória — conjuntos de dados de altíssima resolução ou multi-gigabytes podem exigir muita RAM
- Dados complexos — classificadores Random Forest podem ter desempenho inferior comparado a redes neurais profundas em dados de imagem altamente variáveis ou complexos
Perguntas Frequentes
Sim, o Ilastik suporta totalmente volumes 3D e experimentos em lapso de tempo para segmentação, rastreamento e análise quantitativa em múltiplos pontos temporais.
Sim, o Ilastik é completamente gratuito e de código aberto, disponível para todos os usuários sem restrições de licença.
Não, o Ilastik oferece uma interface gráfica intuitiva com feedback em tempo real, tornando-o acessível a pesquisadores sem experiência em programação. Usuários avançados também podem usar o processamento em lote via linha de comando.
Sim, o fluxo de trabalho dedicado ao rastreamento permite analisar o movimento e a divisão celular em conjuntos de dados 2D e 3D em lapso de tempo com rastreamento automático de linhagem.
Os resultados da segmentação podem ser exportados como imagens rotuladas, mapas de probabilidade ou tabelas quantitativas, permitindo integração fácil com ferramentas de análise e softwares de visualização posteriores.
Essas ferramentas abrangem níveis de novato a especialista. Muitas são gratuitas e de código aberto, facilitando fluxos de trabalho de IA reproduzíveis e compartilháveis na comunidade de pesquisa.
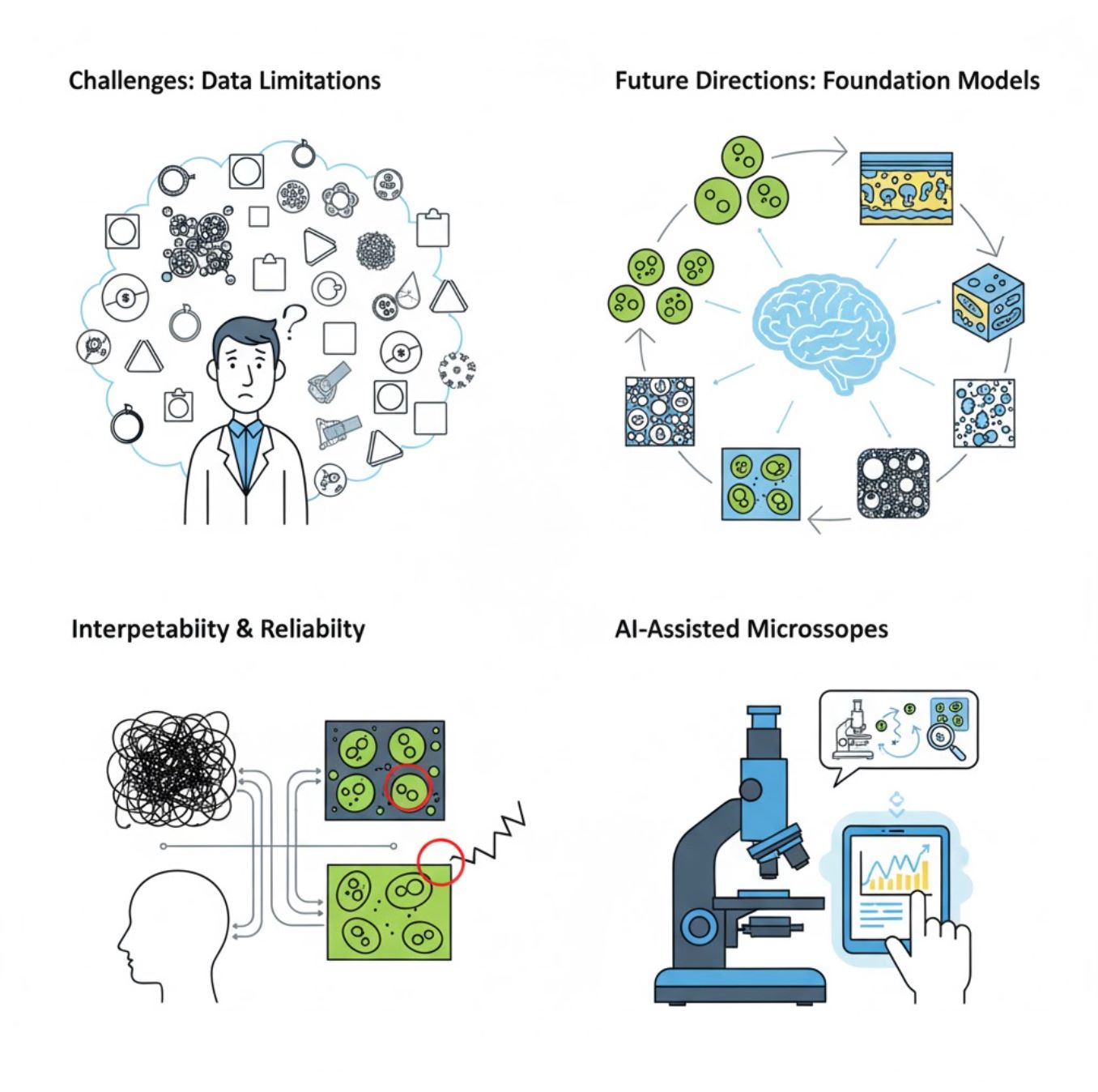
Desafios e Direções Futuras
Desafios Atuais
Tendências Emergentes
Modelos Fundacionais de Visão
Sistemas de IA de próxima geração prometem reduzir a necessidade de treinamento específico para cada tarefa.
- Modelos como SAM e sistemas baseados em CLIP
- Uma IA para várias tarefas de microscopia
- Implantação e adaptação mais rápidas
Microscópios Assistidos por IA
Sistemas de microscopia totalmente autônomos e inteligentes estão se tornando realidade.
- Controle por linguagem natural via LLMs
- Loops de feedback totalmente automatizados
- Democratiza o acesso à microscopia avançada
Principais Conclusões
- A IA está transformando rapidamente o processamento de imagens de microscópio com maior precisão e automação
- O aprendizado profundo supera o aprendizado de máquina tradicional em imagens complexas e variáveis de microscopia
- CNNs aprendem automaticamente características hierárquicas a partir de pixels brutos para análise robusta
- Aplicações-chave incluem segmentação, classificação, rastreamento, redução de ruído e aquisição automática
- O sucesso depende de dados de qualidade e validação cuidadosa por especialistas
- Modelos fundacionais de visão e microscópios assistidos por IA representam o futuro da área
Com avanços contínuos e esforços comunitários (ferramentas open-source, conjuntos de dados compartilhados), a IA se tornará cada vez mais parte central do "olho" do microscópio, ajudando cientistas a ver o invisível.







No comments yet. Be the first to comment!