AI dalam Pemprosesan Imej Mikroskop
AI sedang merevolusikan pemprosesan imej mikroskop dengan keupayaan hebat seperti segmentasi tepat, pengurangan hingar, super-resolusi, dan pemerolehan imej automatik. Artikel ini menyorot alat AI penting dan trend terkini dalam penyelidikan saintifik.
Teknik AI sedang merevolusikan mikroskopi dengan mengoptimumkan pemerolehan imej dan mengautomasikan analisis. Dalam mikroskop pintar moden, modul AI boleh melaraskan parameter pengimejan secara langsung (contohnya, fokus, pencahayaan) untuk mengurangkan photobleaching dan meningkatkan isyarat. Sementara itu, algoritma pembelajaran mendalam boleh menapis data imej yang kompleks untuk mengekstrak wawasan biologi tersembunyi dan malah menghubungkan imej dengan data lain (contohnya, genomik).
Kaedah AI: Pembelajaran Mesin vs Pembelajaran Mendalam
Kaedah AI merangkumi dari pembelajaran mesin (ML) klasik hingga pembelajaran mendalam (DL) moden. Setiap pendekatan mempunyai kekuatan dan keterbatasan tersendiri:
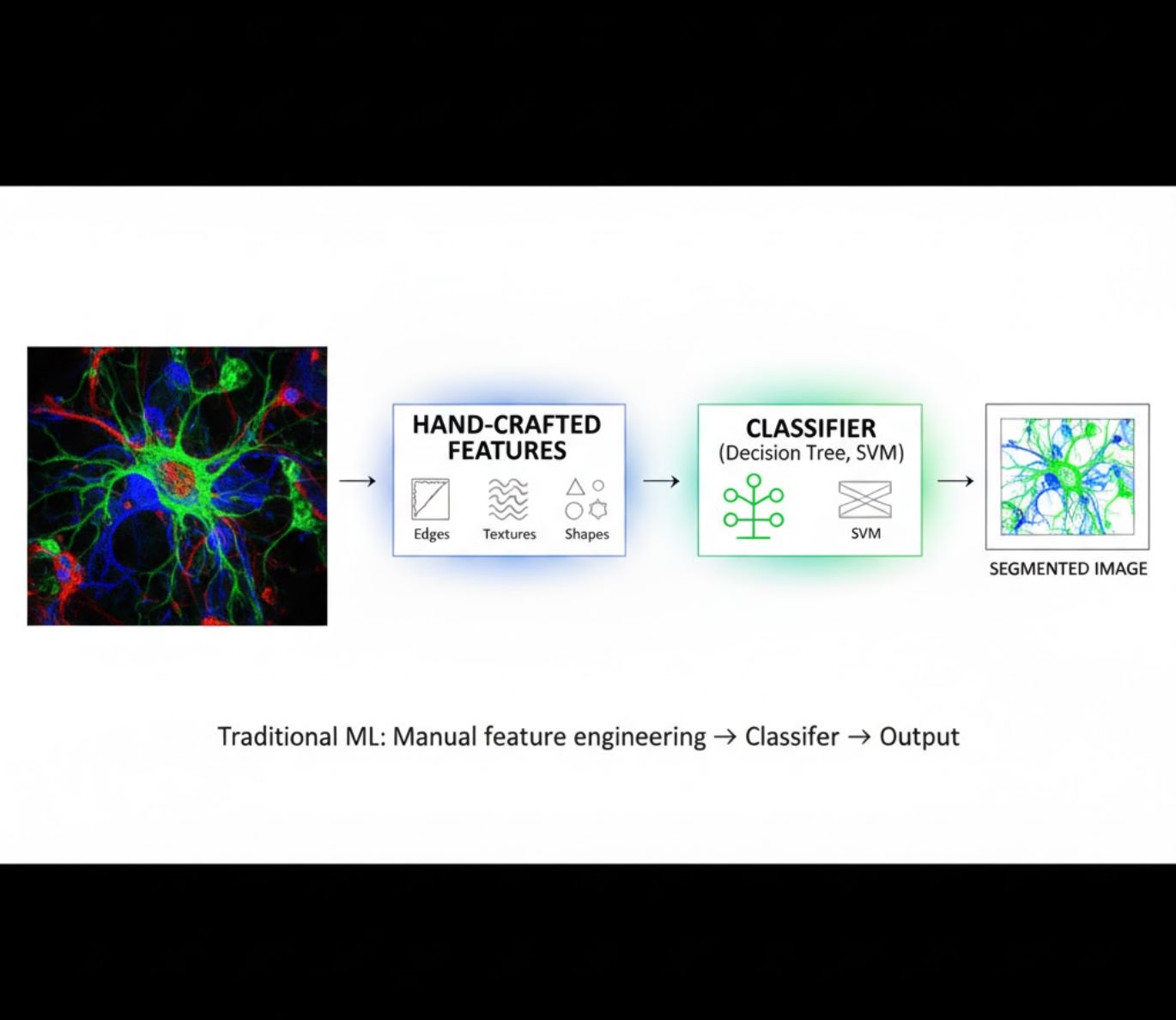
Ciri-ciri Buatan Tangan
- Penyelidik secara manual mencipta ciri imej (tepi, tekstur, bentuk)
- Ciri-ciri diberi kepada pengelasan (pokok keputusan, SVM)
- Latihan pantas
- Kurang berkesan dengan imej kompleks atau bising
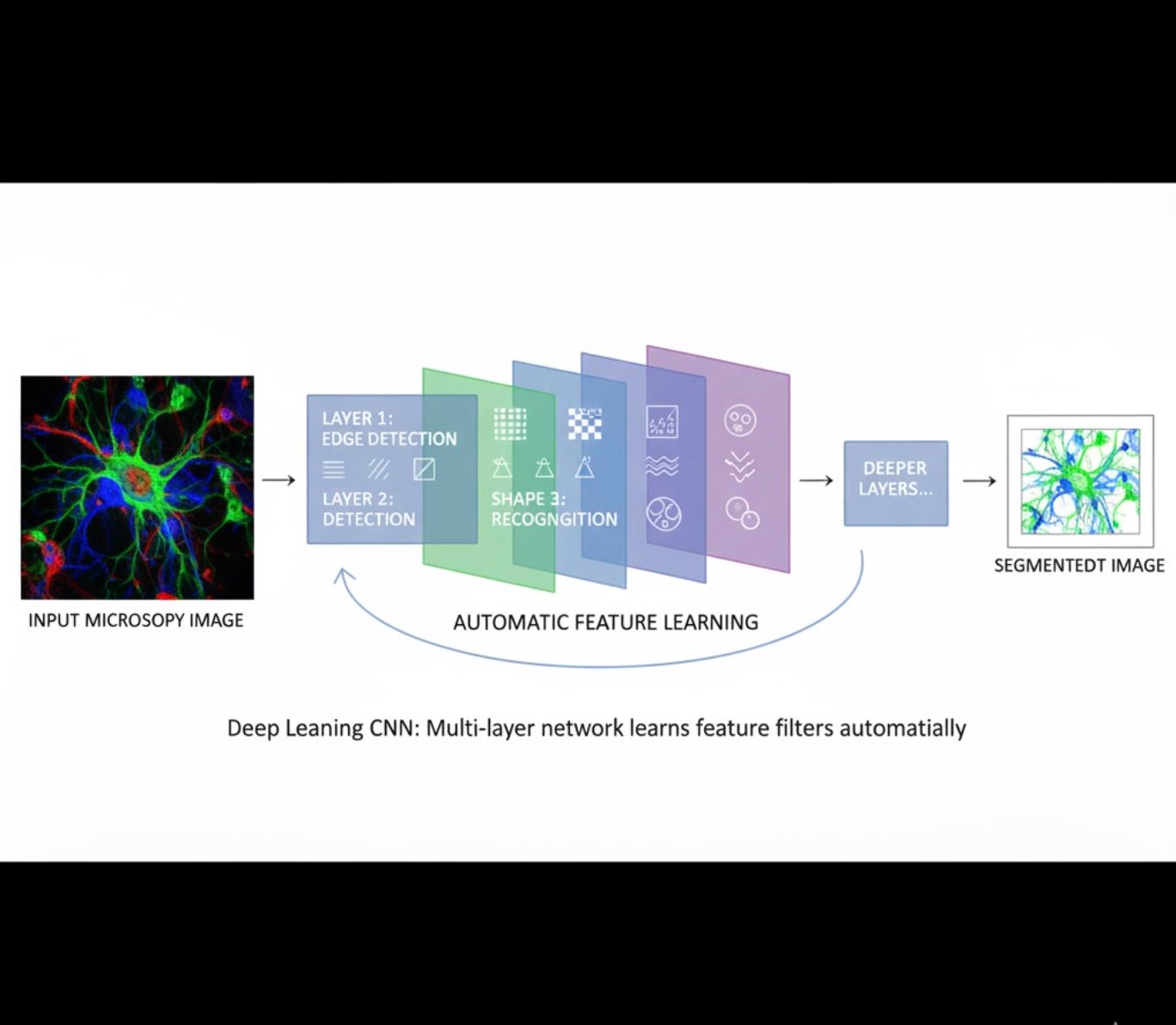
Pembelajaran Ciri Automatik
- Rangkaian neural berlapis-lapis (CNN) belajar ciri secara automatik
- Pembelajaran hujung-ke-hujung dari piksel mentah
- Lebih tahan terhadap variasi
- Menangkap tekstur dan struktur rumit dengan boleh dipercayai
Cara CNN berfungsi: Rangkaian neural konvolusi menggunakan penapis berturut-turut pada imej mikroskopi, belajar mengesan corak mudah (tepi) pada lapisan awal dan struktur kompleks (bentuk sel, tekstur) pada lapisan lebih dalam. Pembelajaran berhierarki ini menjadikan DL sangat tahan walaupun profil intensiti berubah dengan ketara.
Perbandingan Visual: Saluran ML vs DL
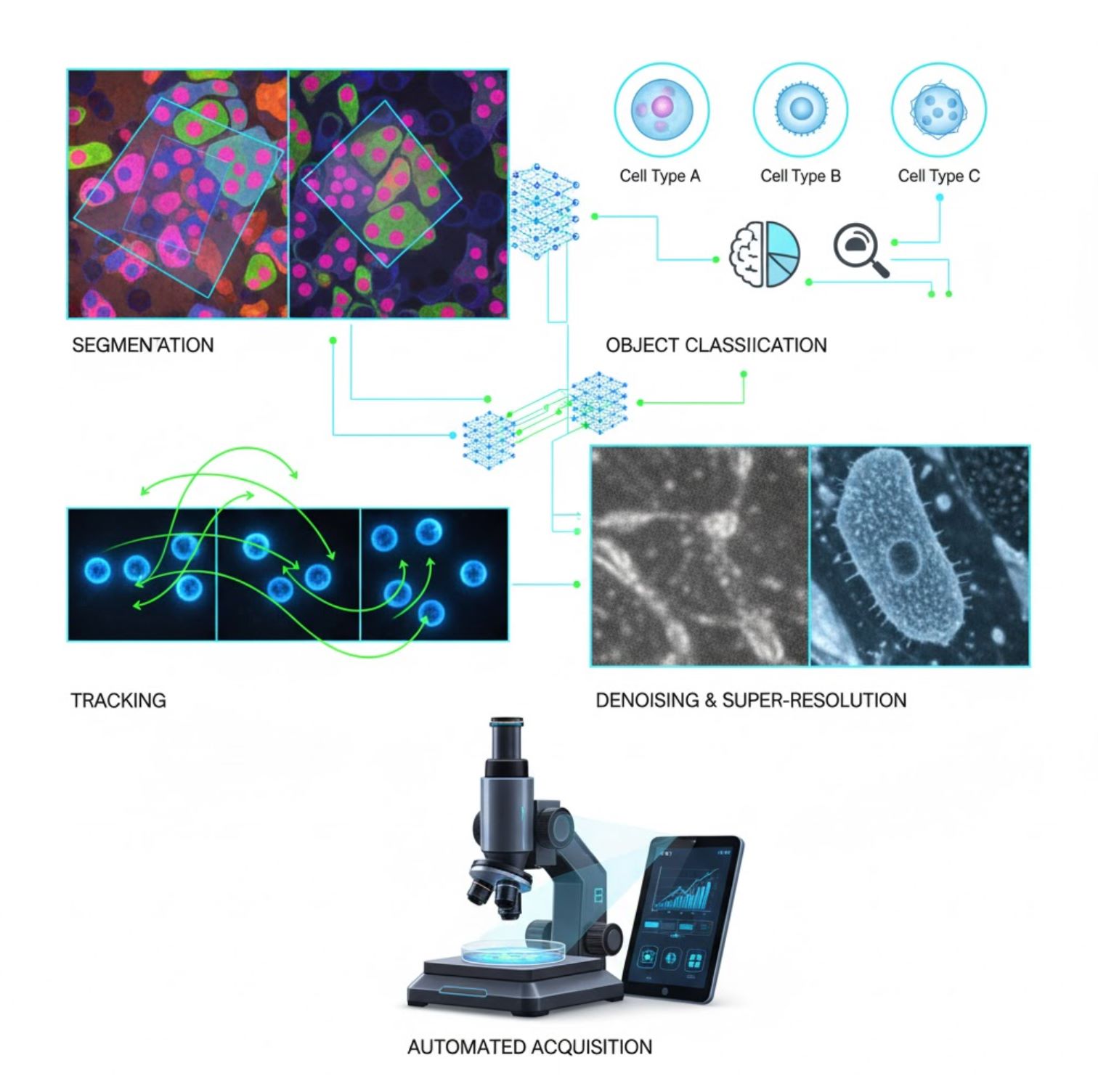
Aplikasi Utama AI dalam Mikroskopi
AI kini tertanam dalam banyak tugas pemprosesan imej sepanjang aliran kerja mikroskopi:
Segmentasi
Membahagikan imej kepada kawasan (contohnya, mengenal pasti setiap sel atau nukleus). Rangkaian mendalam seperti U-Net sangat cemerlang dalam tugas ini.
- Segmentasi semantik: Label kelas per piksel
- Segmentasi instans: Memisahkan objek individu
- Ketepatan tinggi pada imej sesak atau malap
- Model asas visi (contohnya, μSAM) kini disesuaikan untuk mikroskopi
Pengelasan Objek
Selepas segmentasi, AI mengklasifikasikan setiap objek dengan ketepatan tinggi.
- Pengenalpastian jenis sel
- Penentuan tahap mitosis
- Pengesanan penunjuk patologi
- Membedakan fenotip halus yang sukar dikira secara manual
Penjejakan
Dalam mikroskopi masa nyata, AI menjejaki sel atau zarah merentasi bingkai dengan ketepatan tiada tandingan.
- Pembelajaran mendalam meningkatkan ketepatan penjejakan secara dramatik
- Membolehkan analisis boleh dipercayai sel yang bergerak
- Menangkap proses biologi dinamik
Pengurangan Hingar & Super-Resolusi
Model AI meningkatkan kualiti imej dengan membuang hingar dan kabur.
- Model mendalam berasaskan fizik belajar optik mikroskop
- Menghasilkan imej lebih tajam tanpa artifak
- Resolusi lebih tinggi dengan artifak dikurangkan berbanding kaedah tradisional
Pemerolehan Automatik
AI mengawal mikroskop itu sendiri secara masa nyata.
- Menganalisis imej langsung untuk membuat keputusan pintar
- Melaraskan fokus dan mengimbas kawasan berminat secara automatik
- Mengurangkan fototoksisiti dan menjimatkan masa
- Membolehkan eksperimen pengimejan berkapasiti tinggi dan adaptif
Alat AI Popular dalam Pemprosesan Imej Mikroskop
<ITEM_DESCRIPTION>Sejenis ekosistem alat yang kaya menyokong kecerdasan buatan dalam mikroskopi. Para penyelidik telah membina perisian umum dan khusus, kebanyakannya sumber terbuka:</ITEM_DESCRIPTION>
Cellpose
| Pembangun | Carsen Stringer dan Marius Pachitariu (kumpulan penyelidikan MouseLand) |
| Platform Disokong |
Memerlukan Python (pemasangan pip/conda). GUI tersedia hanya pada desktop. |
| Sokongan Bahasa | Dokumentasi dalam Bahasa Inggeris; digunakan secara meluas di makmal penyelidikan di seluruh dunia |
| Model Harga | Percuma dan sumber terbuka di bawah lesen BSD-3-Clause |
Gambaran Keseluruhan
Cellpose adalah alat segmentasi canggih berasaskan pembelajaran mendalam yang direka untuk imej mikroskopi. Sebagai algoritma generalis, ia memisahkan dengan tepat pelbagai jenis sel (nukleus, sitoplasma, dan lain-lain) merentasi pelbagai mod pengimejan tanpa memerlukan latihan semula model. Dengan keupayaan campur tangan manusia, penyelidik boleh memperbaiki keputusan, menyesuaikan model dengan data mereka, dan menggunakan sistem ini untuk aliran kerja pengimejan 2D dan 3D.
Ciri-ciri Utama
Berfungsi terus guna untuk pelbagai jenis sel, pewarnaan, dan mod pengimejan tanpa latihan tersuai.
Menyokong tumpukan 3D penuh menggunakan pendekatan "2.5D" yang menggunakan semula model 2D untuk data volumetrik.
Betulkan hasil segmentasi secara manual dan latih semula model pada data tersuai anda untuk ketepatan yang lebih baik.
Akses melalui API Python, antara muka baris perintah, atau antara muka grafik untuk aliran kerja yang fleksibel.
Keupayaan penyingkiran hingar, pengaburan, dan peningkatan resolusi untuk meningkatkan kualiti imej sebelum segmentasi.
Muat Turun atau Akses
Latar Belakang Teknikal
Cellpose diperkenalkan dalam kajian penting oleh Stringer, Wang, Michaelos, dan Pachitariu, dilatih menggunakan set data besar dan sangat pelbagai yang mengandungi lebih daripada 70,000 objek yang telah disegmentasi. Kepelbagaian ini membolehkan model menggeneralisasi merentasi bentuk, saiz sel, dan tetapan mikroskopi, mengurangkan keperluan latihan tersuai dalam kebanyakan kes penggunaan. Untuk data 3D, Cellpose menggunakan semula model 2D secara bijak dalam cara "2.5D", mengelakkan keperluan data latihan berlabel 3D sepenuhnya sambil tetap memberikan segmentasi volumetrik. Cellpose 2.0 memperkenalkan latihan semula dengan campur tangan manusia, membolehkan pengguna membetulkan ramalan secara manual dan melatih semula pada imej mereka sendiri untuk prestasi yang lebih baik pada set data tertentu.
Pemasangan & Persediaan
Sediakan persekitaran Python menggunakan conda:
conda create -n cellpose python=3.10Aktifkan persekitaran dan pasang Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeMula Menggunakan
Mod GUI
- Jalankan GUI dengan arahan:
python -m cellpose - Seret dan lepaskan fail imej (
.tif,.png, dan lain-lain) ke dalam antara muka - Pilih jenis model (contoh: "cyto" untuk sitoplasma atau "nuclei" untuk nukleus)
- Tetapkan anggaran diameter sel atau biarkan Cellpose kalibrasi secara automatik
- Klik untuk mula segmentasi dan lihat keputusan
Mod API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Perbaiki & Latih Semula
- Selepas menghasilkan topeng, betulkan segmentasi dalam GUI dengan menggabung atau memadam topeng secara manual
- Gunakan fungsi latihan terbina dalam untuk melatih semula pada contoh yang telah diperbaiki
- Prestasi model bertambah baik pada set data khusus anda
Proses Data 3D
- Muatkan fail TIFF multi-Z atau tumpukan volumetrik
- Gunakan bendera
--Zstackdalam GUI atau API untuk memproses sebagai 3D - Opsyenal: perhalusi aliran 3D melalui pelicinan atau parameter khusus untuk segmentasi lebih baik
Had & Pertimbangan
- Perdagangan Kegeneralan Model: Walaupun model generalis berfungsi secara meluas, bentuk sel atau keadaan pengimejan yang sangat luar biasa mungkin memerlukan latihan semula.
- Usaha Anotasi: Latihan dengan campur tangan manusia memerlukan pembetulan manual, yang boleh mengambil masa untuk set data besar.
- Kerumitan Pemasangan: Pemasangan GUI mungkin memerlukan penggunaan baris perintah, persekitaran conda, dan pengurusan kebergantungan Python — tidak selalu mudah untuk bukan pengaturcara.
- Hanya Desktop: Cellpose direka untuk penggunaan desktop; tiada aplikasi asli Android atau iOS tersedia.
Soalan Lazim
Tidak — Cellpose menyediakan model generalis pra-latih yang sering berfungsi dengan baik tanpa latihan semula. Walau bagaimanapun, untuk hasil optimum pada data khas atau luar biasa, anda boleh anotasi dan latih semula menggunakan ciri campur tangan manusia.
Boleh — ia menyokong 3D dengan menggunakan semula model 2D ("2.5D"), dan anda boleh memproses tumpukan volumetrik melalui GUI atau API.
GPU sangat disyorkan untuk inferens dan latihan lebih pantas, terutamanya pada set data besar atau 3D, tetapi Cellpose boleh dijalankan pada mesin hanya CPU dengan prestasi lebih perlahan.
Dalam GUI, tetapkan diameter sel anggaran secara manual atau biarkan Cellpose kalibrasi secara automatik. Anda boleh memperbaiki keputusan dan melatih semula jika segmentasi tidak optimum.
Boleh — versi terkini (Cellpose 3) termasuk model pemulihan imej untuk penyingkiran hingar, pengaburan, dan peningkatan resolusi bagi meningkatkan kualiti segmentasi sebelum pemprosesan.
StarDist
| Pembangun | Uwe Schmidt, Martin Weigert, Coleman Broaddus, dan Gene Myers |
| Platform Disokong |
|
| Sokongan Bahasa | Projek sumber terbuka dengan dokumentasi dan komuniti terutamanya dalam Bahasa Inggeris |
| Model Harga | Percuma dan sumber terbuka. Dilesenkan di bawah BSD-3-Clause |
Gambaran Keseluruhan
StarDist adalah alat pembelajaran mendalam untuk segmentasi instans dalam imej mikroskopi. Ia mewakili setiap objek (seperti nukleus sel) sebagai poligon bintang-konveks dalam 2D atau poliedra dalam 3D, membolehkan pengesanan dan pemisahan objek yang padat atau bertindih dengan tepat. Dengan seni bina yang kukuh, StarDist digunakan secara meluas untuk segmentasi automatik sel dan nukleus dalam mikroskopi fluoresen, histopatologi, dan aplikasi analisis bioimej lain.
Ciri Utama
Segmentasi instans yang sangat tepat menggunakan poligon bintang-konveks (2D) dan poliedra (3D) untuk pengesanan objek yang boleh dipercayai.
Model khusus untuk imej 2D dan data volumetrik 3D untuk analisis mikroskopi yang menyeluruh.
Model sedia guna untuk nukleus fluoresen, histologi bertatah H&E, dan senario pengimejan biasa lain.
Klasifikasikan objek yang dikesan ke dalam kelas berbeza (contohnya, jenis sel berlainan) dalam satu sesi segmentasi.
Integrasi lancar dengan ImageJ/Fiji, QuPath, dan napari untuk aliran kerja berasaskan GUI yang mudah diakses.
Penilaian segmentasi instans yang komprehensif termasuk ketepatan, panggilan balik, skor F1, dan kualiti panoptik.
Latar Belakang Teknikal
Asalnya diperkenalkan dalam kertas MICCAI 2018, inovasi teras StarDist adalah ramalan jarak radial sepanjang sinar tetap digabungkan dengan kebarangkalian objek untuk setiap piksel, membolehkan pembinaan semula bentuk bintang-konveks yang tepat. Pendekatan ini dengan boleh dipercayai memisahkan objek yang rapat bersentuhan yang sukar dipisahkan menggunakan kaedah berasaskan piksel tradisional atau kotak sempadan.
Pembangunan terkini telah mengembangkan StarDist ke imej histopatologi, membolehkan bukan sahaja segmentasi nukleus tetapi juga klasifikasi berbilang kelas objek yang dikesan. Kaedah ini mencapai prestasi teratas dalam cabaran seperti cabaran CoNIC (Pengenalpastian dan Pengiraan Nukleus Kolon).
Muat Turun atau Akses
Pemasangan & Persediaan
Pasang TensorFlow (versi 1.x atau 2.x) sebagai prasyarat untuk StarDist.
Gunakan pip untuk memasang pakej Python StarDist:
pip install stardistUntuk napari:
pip install stardist-napariUntuk QuPath: Pasang sambungan StarDist dengan menyeret fail .jar ke dalam QuPath.
Untuk ImageJ/Fiji: Gunakan pengurus pemalam terbina dalam atau pemasangan manual melalui menu pemalam.
Menjalankan Segmentasi
Muatkan model pra-latih, normalisasikan imej anda, dan jalankan ramalan:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Buka imej anda dalam napari, pilih pemalam StarDist, pilih model pra-latih atau tersuai, dan jalankan ramalan terus dari GUI.
Gunakan pemalam StarDist dari menu Pemalam untuk menerapkan model pada tumpukan imej anda dengan antara muka yang intuitif.
Selepas memasang sambungan, jalankan pengesanan StarDist melalui konsol skrip atau antara muka grafik QuPath untuk analisis histopatologi.
Latihan & Penalaan Halus
Cipta imej label kebenaran asas di mana setiap objek dilabelkan secara unik. Gunakan alat anotasi seperti LabKit, QuPath, atau Fiji untuk menyediakan set data anda.
Gunakan API Python StarDist untuk melatih model baru atau menala halus model sedia ada dengan data anotasi tersuai anda.
Pilihan Pasca-Pemprosesan
- Gunakan penindasan bukan maksimum (NMS) untuk menghapuskan bentuk calon yang berlebihan
- Gunakan StarDist OPP (Pemprosesan Pasca-Objek) untuk menggabungkan topeng bagi bentuk yang bukan bintang-konveks
Had & Pertimbangan
- Andaian bintang-konveks mungkin tidak memodelkan bentuk yang sangat tidak konveks atau sangat tidak teratur dengan sempurna
- Kerumitan pemasangan: pemasangan khusus memerlukan penyusun C++ yang serasi untuk membina sambungan
- Percepatan GPU bergantung pada versi TensorFlow, CUDA, dan cuDNN yang serasi
- Sesetengah pengguna melaporkan isu menjalankan pemalam ImageJ disebabkan konfigurasi Java
Soalan Lazim
StarDist berfungsi dengan pelbagai jenis imej termasuk fluoresen, medan terang, dan histopatologi (contohnya, H&E), terima kasih kepada model pra-latih yang fleksibel dan kebolehannya menyesuaikan diri dengan pelbagai modaliti pengimejan.
Boleh — StarDist menyokong segmentasi instans 3D menggunakan poliedra bintang-konveks untuk data volumetrik, meluaskan keupayaan 2D kepada analisis 3D penuh.
Tidak semestinya. Model pra-latih tersedia dan sering berfungsi dengan baik terus guna. Walau bagaimanapun, untuk data khusus atau baru, anotasi dan latihan model tersuai meningkatkan ketepatan dengan ketara.
StarDist berintegrasi dengan napari, ImageJ/Fiji, dan QuPath, membolehkan anda menjalankan segmentasi dari GUI tanpa pengaturcaraan. Ia juga menyokong penggunaan API Python secara langsung untuk aliran kerja lanjutan.
StarDist menyediakan fungsi terbina dalam untuk mengira metrik segmentasi instans biasa termasuk ketepatan, panggilan balik, skor F1, dan kualiti panoptik untuk menilai prestasi segmentasi.
SAM
Maklumat Aplikasi
| Pembangun | Meta AI Research (FAIR) |
| Peranti Disokong |
|
| Bahasa & Ketersediaan | Model asas sumber terbuka tersedia di seluruh dunia; dokumentasi dalam Bahasa Inggeris |
| Harga | Percuma — sumber terbuka di bawah lesen Meta melalui GitHub dan integrasi MIB |
Gambaran Keseluruhan Umum
SAM (Segment Anything Model) adalah model asas AI yang berkuasa dicipta oleh Meta yang membolehkan segmentasi interaktif dan automatik hampir mana-mana objek dalam imej. Menggunakan arahan seperti titik, kotak sempadan, atau topeng kasar, SAM menghasilkan topeng segmentasi tanpa memerlukan latihan semula khusus tugasan. Dalam penyelidikan mikroskopi, fleksibiliti SAM telah diadaptasi untuk segmentasi sel, pengesanan organel, dan analisis histopatologi, menawarkan penyelesaian skala untuk penyelidik yang memerlukan alat segmentasi serba guna yang boleh diarahkan.
Pengenalan Terperinci
Asalnya dilatih oleh Meta pada lebih 1 bilion topeng merentasi 11 juta imej, SAM direka sebagai model asas yang boleh diarahkan untuk segmentasi dengan prestasi "zero-shot" pada domain baru. Dalam penyelidikan imej perubatan, SAM telah dinilai untuk segmentasi patologi helaian penuh, pengesanan tumor, dan pengecaman nukleus sel. Namun, prestasinya pada contoh yang sangat padat—seperti nukleus sel—adalah bercampur: walaupun dengan arahan yang banyak (contohnya, 20 klik atau kotak), segmentasi zero-shot boleh menghadapi kesukaran dalam imej mikroskopi yang kompleks.
Untuk mengatasi had ini, adaptasi khusus domain telah muncul:
- SAMCell — Ditala semula pada set data mikroskopi besar untuk segmentasi zero-shot yang kuat merentasi pelbagai jenis sel tanpa latihan semula setiap eksperimen
- μSAM — Dilatih semula pada lebih 17,000 imej mikroskopi yang dianotasi secara manual untuk meningkatkan ketepatan pada struktur sel kecil
Ciri Utama
Interaksi fleksibel menggunakan titik, kotak, dan topeng untuk kawalan tepat.
Melakukan segmentasi tanpa penalaan semula pada domain imej baru.
Boleh diadaptasi untuk mikroskopi dan histopatologi melalui latihan semula beberapa contoh atau berasaskan arahan.
Tersedia dalam Microscopy Image Browser (MIB) dengan sokongan segmentasi 3D dan interpolasi.
IDCC-SAM membolehkan pengiraan sel automatik dalam imunositokimia tanpa anotasi manual.
Muat Turun atau Akses
Panduan Pengguna
- Buka Microscopy Image Browser dan navigasi ke panel segmentasi SAM
- Konfigurasikan interpreter Python dan pilih antara model SAM-1 atau SAM-2
- Untuk pecutan GPU, pilih "cuda" dalam persekitaran pelaksanaan (disyorkan untuk prestasi optimum)
- Arahan titik: Klik pada objek untuk menentukan benih positif; gunakan Shift + klik untuk mengembangkan dan Ctrl + klik untuk benih negatif
- Tumpukan 3D: Gunakan mod Interaktif 3D—klik pada satu irisan, gulung shift, dan interpolasi benih merentasi irisan
- Mod laras: Gantikan, tambah, tolak topeng, atau cipta lapisan baru mengikut keperluan
- Gunakan pilihan "Automatic everything" MIB dalam panel SAM-2 untuk mensegmentasi semua objek yang kelihatan dalam sesuatu kawasan
- Semak dan perbaiki topeng selepas segmentasi mengikut keperluan
- Gunakan saluran penalaan semula berasaskan arahan (contohnya, "All-in-SAM") untuk menghasilkan anotasi per piksel daripada arahan pengguna yang jarang
- Untuk pengiraan sel, gunakan IDCC-SAM, yang menggunakan SAM dalam saluran zero-shot dengan pemprosesan pasca
- Untuk segmentasi sel berketepatan tinggi, gunakan SAMCell, yang ditala semula pada imej sel mikroskopi
Had & Pertimbangan
- Prestasi zero-shot tidak konsisten pada struktur padat atau bertindih tanpa penalaan domain
- Kualiti segmentasi sangat bergantung pada reka bentuk dan strategi arahan
- GPU sangat disyorkan; inferens CPU sangat perlahan
- Menghadapi kesukaran dengan imej helaian penuh beresolusi sangat tinggi dan struktur tisu berbilang skala
- Penalaan semula atau adaptasi SAM untuk mikroskopi mungkin memerlukan kemahiran pembelajaran mesin
Soalan Lazim
Boleh—melalui adaptasi seperti SAMCell, yang menala semula SAM pada set data mikroskopi khusus untuk tugasan segmentasi sel.
Tidak selalu. Dengan IDCC-SAM, anda boleh melakukan pengiraan sel zero-shot tanpa anotasi manual.
Gunakan penalaan semula berasaskan arahan (contohnya, "All-in-SAM") atau versi mikroskopi terlatih seperti μSAM, yang dilatih pada lebih 17,000 imej mikroskopi yang dianotasi.
Walaupun boleh dijalankan pada CPU, GPU sangat disyorkan untuk kelajuan inferens praktikal dan segmentasi interaktif masa nyata.
Boleh—integrasi SAM-2 dalam MIB menyokong segmentasi 3D dengan interpolasi benih merentasi irisan untuk analisis volumetrik.
AxonDeepSeg
| Pembangun | Makmal NeuroPoly di Polytechnique Montréal dan Université de Montréal |
| Platform Disokong |
|
| Bahasa | Dokumentasi dalam Bahasa Inggeris; alat sumber terbuka digunakan secara global |
| Harga | Percuma dan sumber terbuka |
Gambaran Keseluruhan
AxonDeepSeg ialah alat berkuasa AI untuk segmentasi automatik akson dan mielin dalam imej mikroskopi. Menggunakan rangkaian neural konvolusi, ia memberikan segmentasi tiga kelas yang tepat (akson, mielin, latar belakang) merentas pelbagai modaliti pengimejan termasuk TEM, SEM, dan mikroskopi medan cerah. Dengan mengautomasikan pengukuran morfometrik seperti diameter akson, nisbah g, dan ketebalan mielin, AxonDeepSeg mempermudah analisis kuantitatif dalam penyelidikan neurosains, mengurangkan masa anotasi manual dengan ketara dan meningkatkan kebolehpercayaan.
Ciri Utama
Model sedia guna yang dioptimumkan untuk modaliti mikroskopi TEM, SEM, dan medan cerah.
Klasifikasi tepat kawasan akson, mielin, dan latar belakang dalam imej mikroskopi.
Pengiraan automatik diameter akson, nisbah g, ketebalan mielin, dan metrik ketumpatan.
Integrasi GUI Napari membolehkan penambahbaikan manual topeng segmentasi untuk ketepatan lebih baik.
Integrasi lancar ke dalam saluran tersuai untuk analisis tisu neural berskala besar.
Skrip ujian menyeluruh memastikan kebolehulangan dan keputusan segmentasi yang boleh dipercayai.
Butiran Teknikal
Dibangunkan oleh Makmal NeuroPoly, AxonDeepSeg memanfaatkan pembelajaran mendalam untuk memberikan segmentasi berketepatan tinggi bagi aplikasi neurosains. Model terlatih tersedia untuk pelbagai modaliti mikroskopi, memastikan kepelbagaian merentas teknik pengimejan. Alat ini berintegrasi dengan Napari, membolehkan pembetulan interaktif topeng segmentasi, yang meningkatkan ketepatan pada set data yang mencabar. AxonDeepSeg mengira metrik morfometrik utama, menyokong kajian berkapasiti tinggi struktur dan patologi tisu neural. Rangka kerja berasaskan Python membolehkan integrasi ke dalam saluran tersuai untuk analisis berskala besar morfologi akson dan mielin.
Muat Turun atau Akses
Pemasangan & Persediaan
Pastikan Python 3.8 atau lebih baru dipasang, kemudian pasang AxonDeepSeg dan Napari menggunakan pip:
pip install axondeepseg napariJalankan skrip ujian yang disediakan untuk mengesahkan semua komponen dipasang dan berfungsi dengan betul.
Import imej mikroskopi (TEM, SEM, atau medan cerah) ke dalam Napari atau persekitaran Python anda.
Pilih model terlatih yang sesuai untuk modaliti pengimejan anda dan jalankan segmentasi untuk menghasilkan topeng akson dan mielin.
Hitung secara automatik pengukuran morfometrik termasuk diameter akson, nisbah g, ketumpatan, dan ketebalan mielin, kemudian eksport keputusan dalam format CSV.
Gunakan GUI Napari untuk melaraskan topeng segmentasi secara manual jika perlu, menggabungkan atau memadam topeng untuk ketepatan lebih baik.
Pertimbangan Penting
- Prestasi mungkin menurun pada modaliti pengimejan baru atau tidak dilatih
- Pembetulan manual mungkin diperlukan untuk kawasan yang mencabar atau kompleks
- GPU disyorkan untuk pemprosesan lebih pantas set data besar; pemprosesan CPU juga disokong
Soalan Lazim
AxonDeepSeg menyokong TEM (Transmission Electron Microscopy), SEM (Scanning Electron Microscopy), dan mikroskopi medan cerah dengan model terlatih yang dioptimumkan untuk setiap modaliti.
Ya, AxonDeepSeg adalah sepenuhnya percuma dan sumber terbuka, tersedia untuk kegunaan akademik dan komersial.
Ya, AxonDeepSeg secara automatik mengira diameter akson, nisbah g, ketebalan mielin, dan metrik ketumpatan daripada imej yang telah disegmentasi.
GPU disyorkan untuk segmentasi lebih pantas set data besar, tetapi pemprosesan CPU juga disokong untuk analisis yang lebih kecil.
Ya, integrasi GUI Napari membolehkan pembetulan interaktif dan penambahbaikan topeng segmentasi untuk ketepatan lebih tinggi pada kawasan yang mencabar.
Ilastik
| Pembangun | Pasukan Ilastik di Makmal Biologi Molekul Eropah (EMBL) dan rakan akademik berkaitan |
| Platform Disokong |
|
| Bahasa | Bahasa Inggeris |
| Harga | Percuma dan sumber terbuka |
Gambaran Keseluruhan
Ilastik ialah alat berkuasa yang dipacu AI untuk segmentasi imej interaktif, pengelasan, dan analisis data mikroskopi. Menggunakan teknik pembelajaran mesin seperti pengelasan Random Forest, ia membolehkan penyelidik menyegmentasi piksel, mengelaskan objek, menjejaki sel dari masa ke masa, dan melakukan pengiraan ketumpatan dalam set data 2D dan 3D. Dengan antara muka intuitif dan maklum balas masa nyata, Ilastik mudah diakses oleh saintis tanpa kepakaran pengaturcaraan dan digunakan secara meluas dalam biologi sel, neurosains, dan pengimejan bioperubatan.
Ciri-ciri Utama
Maklum balas masa nyata semasa anda memberi anotasi kawasan contoh untuk hasil segmentasi segera.
Mengategorikan struktur yang disegmentasi berdasarkan ciri morfologi dan intensiti.
Menjejaki pergerakan dan pembahagian sel dalam eksperimen mikroskopi masa-laps 2D dan 3D.
Mengira kawasan sesak tanpa segmentasi eksplisit objek individu.
Segmentasi separa automatik untuk volum 3D yang kompleks dengan interaksi intuitif.
Memproses pelbagai imej secara automatik menggunakan mod baris perintah tanpa kepala.
Muat Turun
Panduan Mula
Muat turun Ilastik untuk sistem operasi anda dari laman web rasmi. Pakej ini termasuk semua kebergantungan Python yang diperlukan, jadi ikut arahan pemasangan untuk platform anda.
Buka Ilastik dan pilih aliran kerja analisis anda: Pengelasan Piksel, Pengelasan Objek, Penjejakan, atau Pengiraan Ketumpatan. Muatkan set data imej anda, yang boleh merangkumi imej berbilang saluran, 3D, atau masa-laps.
Label beberapa piksel atau objek contoh dalam imej anda. Pengelasan Random Forest Ilastik belajar daripada anotasi ini dan secara automatik meramalkan label di seluruh set data anda.
Gunakan model yang telah dilatih untuk menyegmentasi atau mengelaskan keseluruhan set data anda. Eksport keputusan sebagai imej berlabel, peta kebarangkalian, atau jadual kuantitatif untuk analisis dan visualisasi selanjutnya.
Gunakan mod tanpa kepala Ilastik untuk memproses pelbagai imej secara automatik tanpa campur tangan manual, sesuai untuk saluran analisis berskala besar.
Had & Pertimbangan
- Pelabelan interaktif boleh mengambil masa yang lama untuk set data yang sangat besar
- Ketepatan bergantung pada kualiti dan keterwakilan anotasi pengguna
- Keperluan memori — set data beresolusi sangat tinggi atau berbilang gigabait mungkin memerlukan RAM yang besar
- Data kompleks — pengelasan Random Forest mungkin kurang berprestasi berbanding rangkaian neural dalam data imej yang sangat berubah-ubah atau kompleks
Soalan Lazim
Ya, Ilastik menyokong sepenuhnya volum 3D dan eksperimen masa-laps untuk segmentasi, penjejakan, dan analisis kuantitatif merentas pelbagai titik masa.
Ya, Ilastik adalah sepenuhnya percuma dan sumber terbuka, tersedia untuk semua pengguna tanpa sekatan lesen.
Tidak, Ilastik menyediakan antara muka grafik yang intuitif dengan maklum balas masa nyata, menjadikannya mudah diakses oleh penyelidik tanpa kepakaran pengaturcaraan. Pengguna lanjutan juga boleh menggunakan pemprosesan batch melalui baris perintah.
Ya, aliran kerja penjejakan khusus membolehkan analisis pergerakan dan pembahagian sel dalam set data masa-laps 2D dan 3D dengan penjejakan garis keturunan automatik.
Output segmentasi boleh dieksport sebagai imej berlabel, peta kebarangkalian, atau jadual kuantitatif, membolehkan integrasi lancar dengan alat analisis dan perisian visualisasi seterusnya.
Alat ini merangkumi tahap pemula hingga pakar. Ramai yang percuma dan sumber terbuka, memudahkan aliran kerja AI yang boleh diulang dan dikongsi dalam komuniti penyelidikan.
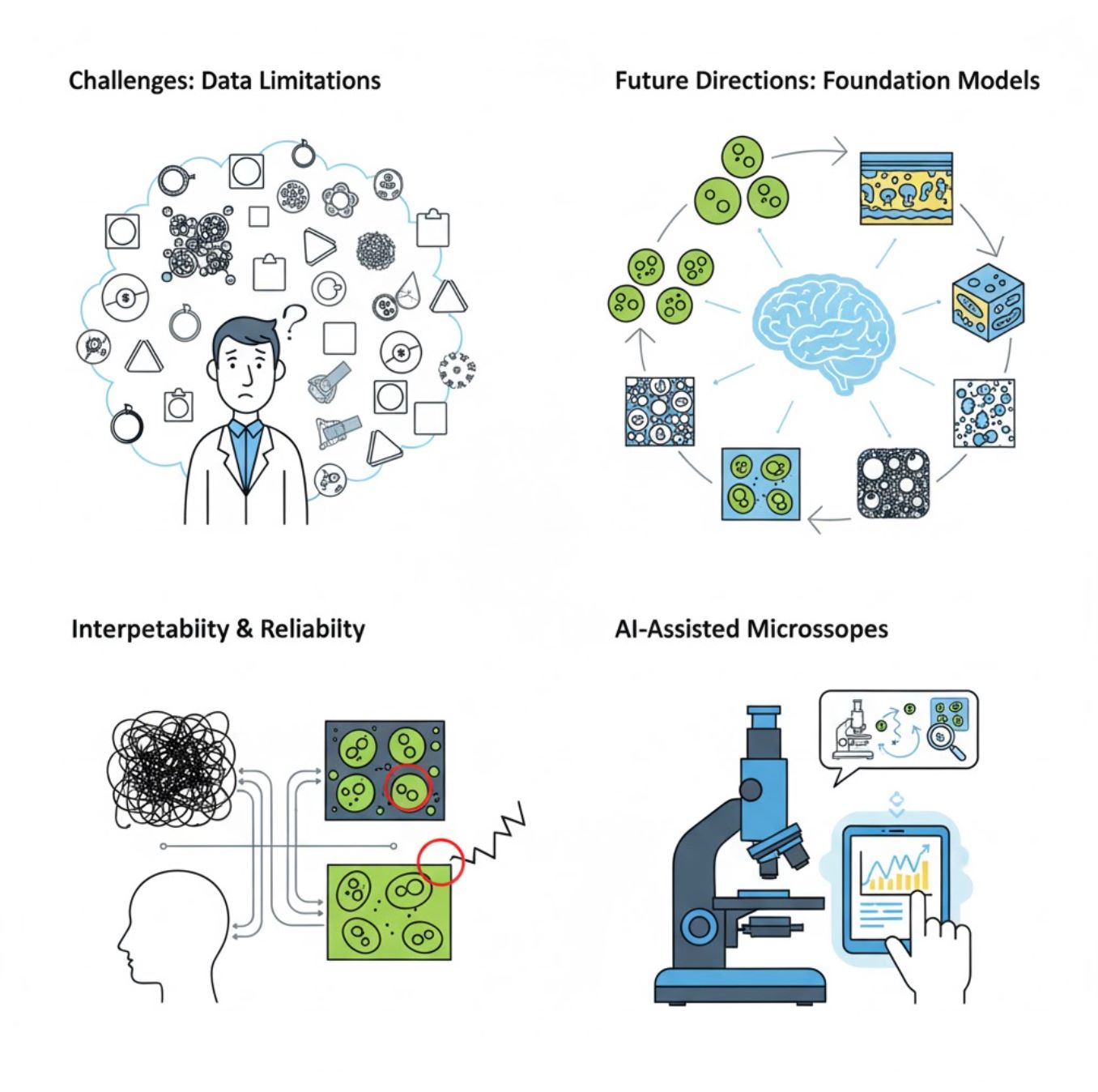
Cabaran dan Arah Masa Depan
Cabaran Semasa
Trend Terkini
Model Asas Visi
Sistem AI generasi akan datang menjanjikan pengurangan keperluan latihan khusus tugas.
- Model seperti SAM dan sistem berasaskan CLIP
- Satu AI mengendalikan banyak tugas mikroskopi
- Penerapan dan penyesuaian lebih pantas
Mikroskop Berbantu AI
Sistem mikroskopi sepenuhnya autonomi dan pintar semakin menjadi kenyataan.
- Kawalan bahasa semula jadi melalui LLM
- Gelung maklum balas automatik sepenuhnya
- Demokratisasi akses mikroskopi canggih
Pengajaran Utama
- AI dengan pantas mengubah pemprosesan imej mikroskop dengan ketepatan dan automasi yang dipertingkatkan
- Pembelajaran mendalam mengatasi pembelajaran mesin tradisional pada imej mikroskopi yang kompleks dan berubah-ubah
- CNN secara automatik belajar ciri berhierarki dari piksel mentah untuk analisis tahan lasak
- Aplikasi utama termasuk segmentasi, pengelasan, penjejakan, pengurangan hingar, dan pemerolehan automatik
- Keberhasilan bergantung pada data berkualiti dan pengesahan teliti oleh pakar
- Model asas visi dan mikroskop berbantu AI mewakili masa depan bidang ini
Dengan kemajuan berterusan dan usaha komuniti (alat sumber terbuka, set data dikongsi), AI akan semakin menjadi bahagian teras "mata" mikroskop, membantu saintis melihat yang tidak kelihatan.







No comments yet. Be the first to comment!