Τεχνητή Νοημοσύνη στην Επεξεργασία Εικόνων Μικροσκοπίου
Η τεχνητή νοημοσύνη φέρνει επανάσταση στην επεξεργασία εικόνων μικροσκοπίου με ισχυρές δυνατότητες όπως ακριβής τμηματοποίηση, μείωση θορύβου, υπερ-ανάλυση και αυτοματοποιημένη απόκτηση εικόνων. Αυτό το άρθρο αναδεικνύει βασικά εργαλεία τεχνητής νοημοσύνης και αναδυόμενες τάσεις στην επιστημονική έρευνα.
Οι τεχνικές τεχνητής νοημοσύνης φέρνουν επανάσταση στη μικροσκοπία βελτιστοποιώντας την απόκτηση εικόνων και αυτοματοποιώντας την ανάλυση. Στα σύγχρονα έξυπνα μικροσκόπια, τα μοντέλα τεχνητής νοημοσύνης μπορούν να ρυθμίζουν παραμέτρους απεικόνισης σε πραγματικό χρόνο (π.χ., εστίαση, φωτισμός) για να ελαχιστοποιούν το φωτοθάμπωμα και να ενισχύουν το σήμα. Παράλληλα, οι αλγόριθμοι βαθιάς μάθησης μπορούν να επεξεργάζονται πολύπλοκα δεδομένα εικόνας για να εξάγουν κρυφές βιολογικές πληροφορίες και ακόμη να συνδέουν εικόνες με άλλα δεδομένα (π.χ., γονιδιωματικά).
- 1. Μέθοδοι Τεχνητής Νοημοσύνης: Μηχανική Μάθηση vs Βαθιά Μάθηση
- 2. Οπτική Σύγκριση: Ροές Εργασίας ML vs DL
- 3. Βασικές Εφαρμογές Τεχνητής Νοημοσύνης στη Μικροσκοπία
- 4. Δημοφιλή Εργαλεία Τεχνητής Νοημοσύνης στην Επεξεργασία Εικόνων Μικροσκοπίου
- 5. Προκλήσεις και Μελλοντικές Κατευθύνσεις
- 6. Βασικά Συμπεράσματα
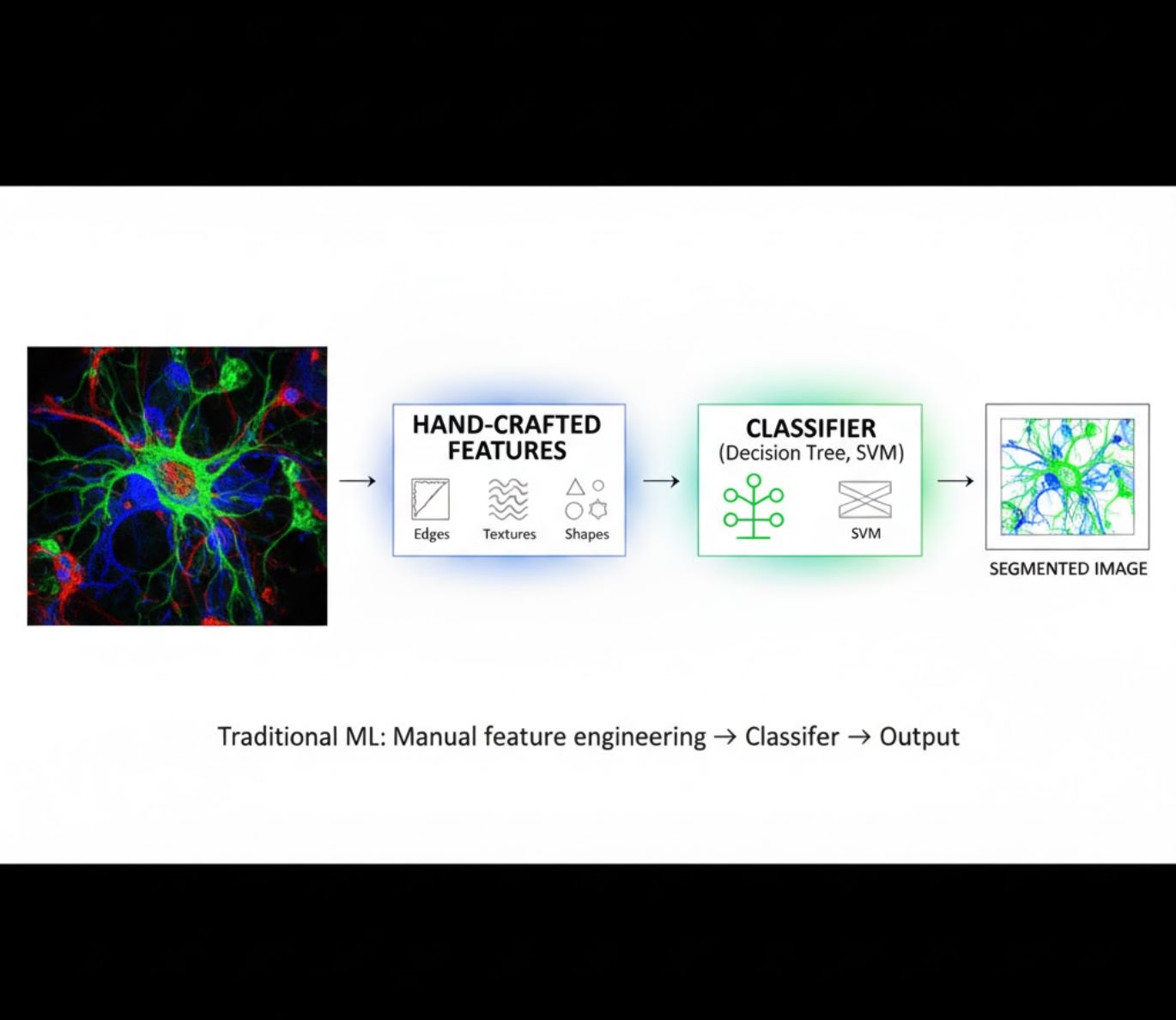
Μέθοδοι Τεχνητής Νοημοσύνης: Μηχανική Μάθηση vs Βαθιά Μάθηση
Οι μέθοδοι τεχνητής νοημοσύνης κυμαίνονται από την κλασική μηχανική μάθηση (ML) έως τη σύγχρονη βαθιά μάθηση (DL). Κάθε προσέγγιση έχει ξεχωριστά πλεονεκτήματα και περιορισμούς:
Χειροποίητα Χαρακτηριστικά
- Οι ερευνητές δημιουργούν χειροκίνητα χαρακτηριστικά εικόνας (άκρα, υφές, σχήματα)
- Τα χαρακτηριστικά τροφοδοτούνται σε ταξινομητές (δέντρα αποφάσεων, SVM)
- Γρήγορη εκπαίδευση
- Δυσκολεύεται με πολύπλοκες ή θορυβώδεις εικόνες
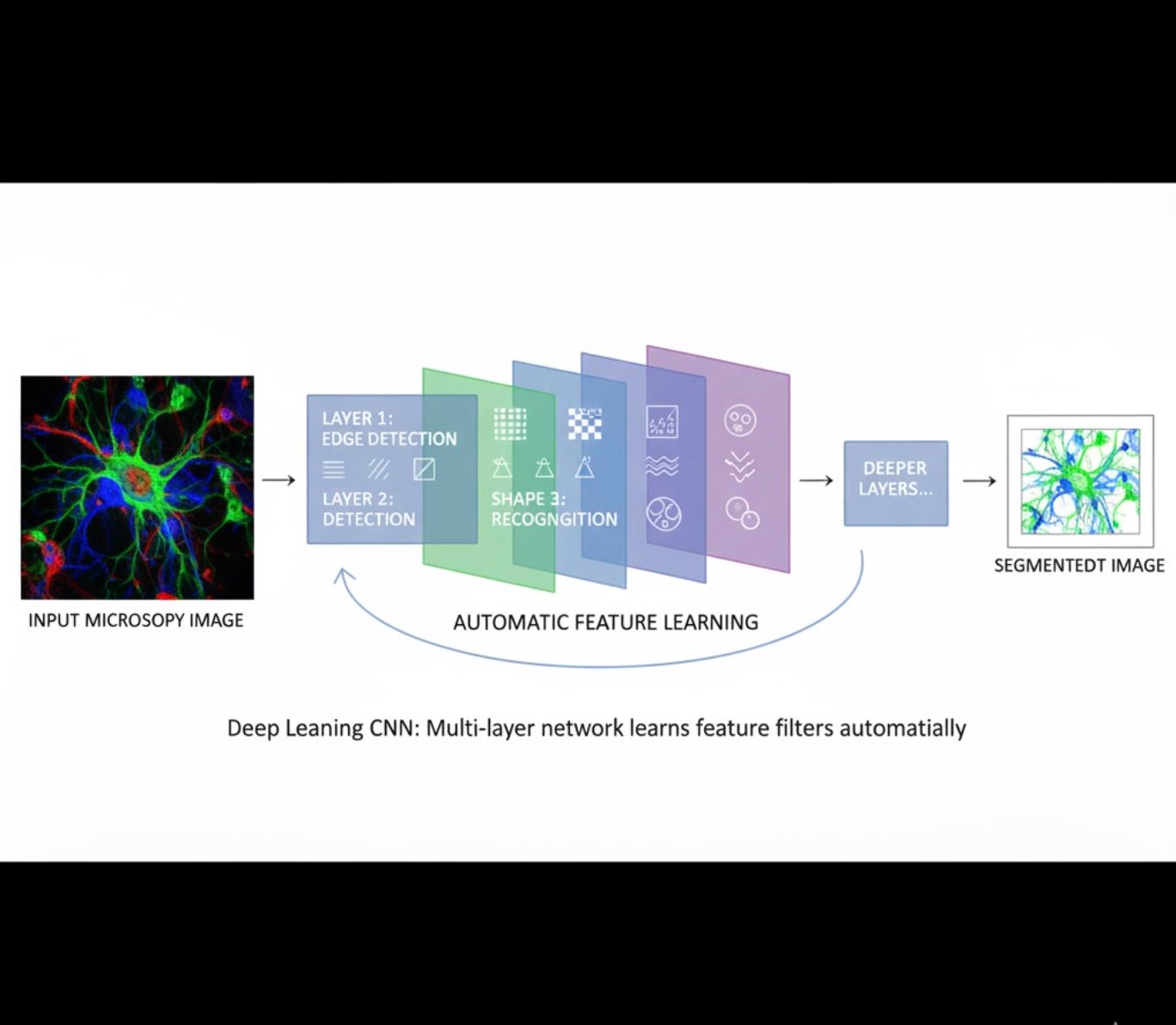
Αυτόματη Εκμάθηση Χαρακτηριστικών
- Πολυεπίπεδα νευρωνικά δίκτυα (CNNs) μαθαίνουν χαρακτηριστικά αυτόματα
- Εκμάθηση από ακατέργαστα pixels από άκρο σε άκρο
- Πολύ πιο ανθεκτική σε παραλλαγές
- Ανιχνεύει με ακρίβεια πολύπλοκες υφές και δομές
Πώς λειτουργούν τα CNNs: Τα συνελικτικά νευρωνικά δίκτυα εφαρμόζουν διαδοχικά φίλτρα σε εικόνες μικροσκοπίου, μαθαίνοντας να ανιχνεύουν απλά μοτίβα (άκρα) στα πρώτα στρώματα και σύνθετες δομές (σχήματα κυττάρων, υφές) στα βαθύτερα στρώματα. Αυτή η ιεραρχική μάθηση καθιστά τη βαθιά μάθηση εξαιρετικά ανθεκτική ακόμη και όταν τα προφίλ έντασης ποικίλλουν σημαντικά.
Οπτική Σύγκριση: Ροές Εργασίας ML vs DL
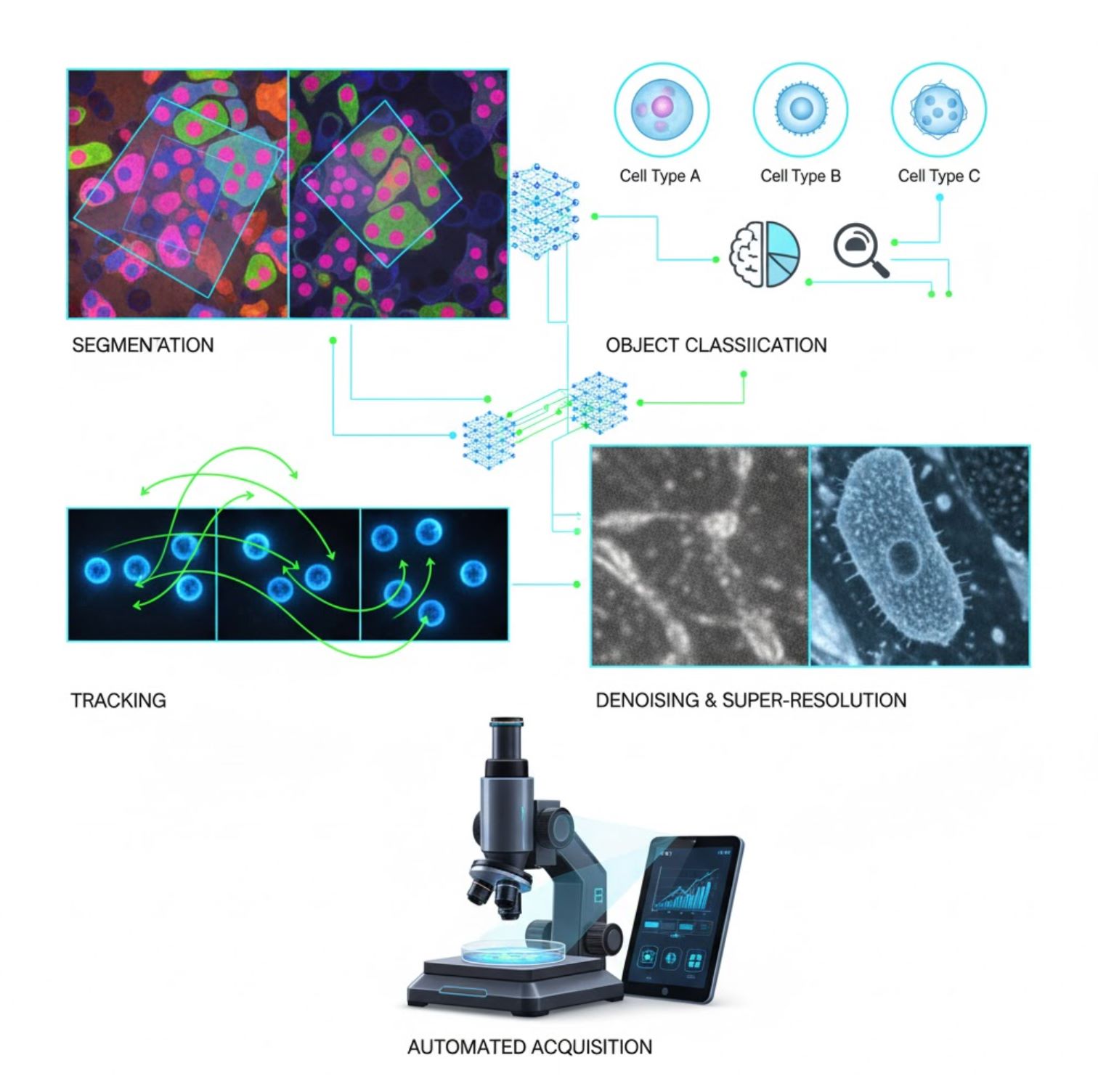
Βασικές Εφαρμογές Τεχνητής Νοημοσύνης στη Μικροσκοπία
Η τεχνητή νοημοσύνη είναι πλέον ενσωματωμένη σε πολλές εργασίες επεξεργασίας εικόνας κατά τη ροή εργασίας μικροσκοπίας:
Τμηματοποίηση
Διαίρεση εικόνων σε περιοχές (π.χ., αναγνώριση κάθε κυττάρου ή πυρήνα). Τα βαθιά δίκτυα όπως το U-Net διαπρέπουν σε αυτή την εργασία.
- Σημασιολογική τμηματοποίηση: Ετικέτες κατηγορίας ανά pixel
- Τμηματοποίηση αντικειμένων: Διαχωρισμός μεμονωμένων αντικειμένων
- Υψηλή ακρίβεια σε πυκνές ή σκοτεινές εικόνες
- Μοντέλα θεμελιώδους όρασης (π.χ., μSAM) πλέον προσαρμοσμένα για μικροσκοπία
Ταξινόμηση Αντικειμένων
Μετά την τμηματοποίηση, η τεχνητή νοημοσύνη ταξινομεί κάθε αντικείμενο με υψηλή ακρίβεια.
- Αναγνώριση τύπου κυττάρου
- Καθορισμός σταδίου μίτωσης
- Ανίχνευση δεικτών παθολογίας
- Διακρίνει λεπτές φαινοτυπικές διαφορές δύσκολες στην χειροκίνητη ποσοτικοποίηση
Παρακολούθηση
Στη μικροσκοπία χρονικής καθυστέρησης, η τεχνητή νοημοσύνη παρακολουθεί κύτταρα ή σωματίδια ανάμεσα σε καρέ με πρωτοφανή ακρίβεια.
- Η βαθιά μάθηση βελτιώνει δραματικά την ακρίβεια παρακολούθησης
- Επιτρέπει αξιόπιστη ανάλυση κινούμενων κυττάρων
- Καταγράφει δυναμικές βιολογικές διεργασίες
Αποθορυβοποίηση & Υπερ-Ανάλυση
Τα μοντέλα τεχνητής νοημοσύνης βελτιώνουν την ποιότητα εικόνας αφαιρώντας θόρυβο και θάμπωμα.
- Φυσικά ενημερωμένα βαθιά μοντέλα μαθαίνουν την οπτική του μικροσκοπίου
- Ανακατασκευάζουν πιο καθαρές, χωρίς σφάλματα εικόνες
- Υψηλότερη ανάλυση με λιγότερα σφάλματα σε σχέση με παραδοσιακές μεθόδους
Αυτοματοποιημένη Απόκτηση
Η τεχνητή νοημοσύνη καθοδηγεί το ίδιο το μικροσκόπιο σε πραγματικό χρόνο.
- Αναλύει ζωντανές εικόνες για να λαμβάνει έξυπνες αποφάσεις
- Ρυθμίζει αυτόματα την εστίαση και σκανάρει περιοχές ενδιαφέροντος
- Μειώνει τη φωτοτοξικότητα και εξοικονομεί χρόνο
- Επιτρέπει πειράματα υψηλής απόδοσης και προσαρμοστικής απεικόνισης
Δημοφιλή Εργαλεία Τεχνητής Νοημοσύνης στην Επεξεργασία Εικόνων Μικροσκοπίου
A rich ecosystem of tools supports AI in microscopy. Researchers have built both general-purpose and specialized software, many open-source:
Cellpose
| Δημιουργοί | Carsen Stringer και Marius Pachitariu (ομάδα έρευνας MouseLand) |
| Υποστηριζόμενες Πλατφόρμες |
Απαιτεί Python (εγκατάσταση μέσω pip/conda). Το GUI είναι διαθέσιμο μόνο σε επιτραπέζιους υπολογιστές. |
| Υποστήριξη Γλώσσας | Τεκμηρίωση στα Αγγλικά· ευρέως υιοθετημένο σε ερευνητικά εργαστήρια παγκοσμίως |
| Μοντέλο Τιμολόγησης | Δωρεάν και ανοιχτού κώδικα υπό άδεια BSD-3-Clause |
Επισκόπηση
Το Cellpose είναι ένα προηγμένο εργαλείο τμηματοποίησης βασισμένο σε βαθιά μάθηση, σχεδιασμένο για μικροσκοπικές εικόνες. Ως γενικός αλγόριθμος, τμηματοποιεί με ακρίβεια διάφορους τύπους κυττάρων (πυρήνες, κυτταρόπλασμα κ.ά.) σε διαφορετικές μεθόδους απεικόνισης χωρίς να απαιτείται επανεκπαίδευση του μοντέλου. Με δυνατότητες ανθρώπινης παρέμβασης, οι ερευνητές μπορούν να βελτιώσουν τα αποτελέσματα, να προσαρμόσουν το μοντέλο στα δεδομένα τους και να εφαρμόσουν το σύστημα σε ροές εργασίας 2D και 3D.
Κύρια Χαρακτηριστικά
Λειτουργεί άμεσα για μεγάλη ποικιλία τύπων κυττάρων, χρωστικών και μεθόδων απεικόνισης χωρίς προσαρμοσμένη εκπαίδευση.
Υποστηρίζει πλήρη 3D στοίβες χρησιμοποιώντας προσέγγιση "2.5D" που επαναχρησιμοποιεί 2D μοντέλα για όγκο δεδομένων.
Διορθώστε χειροκίνητα τα αποτελέσματα τμηματοποίησης και επανεκπαιδεύστε το μοντέλο στα δικά σας δεδομένα για βελτιωμένη ακρίβεια.
Πρόσβαση μέσω Python API, γραμμής εντολών ή γραφικής διεπαφής για ευέλικτες ροές εργασίας.
Δυνατότητες αποθορυβοποίησης, αποθόλωσης και αύξησης ανάλυσης για βελτίωση της ποιότητας εικόνας πριν την τμηματοποίηση.
Λήψη ή Πρόσβαση
Τεχνικό Υπόβαθρο
Το Cellpose παρουσιάστηκε σε μια σημαντική μελέτη από τους Stringer, Wang, Michaelos και Pachitariu, εκπαιδευμένο σε ένα μεγάλο και πολύ ποικίλο σύνολο δεδομένων που περιλαμβάνει πάνω από 70.000 τμηματοποιημένα αντικείμενα. Αυτή η ποικιλία επιτρέπει στο μοντέλο να γενικεύει σε διαφορετικά σχήματα, μεγέθη κυττάρων και ρυθμίσεις μικροσκοπίας, μειώνοντας σημαντικά την ανάγκη για προσαρμοσμένη εκπαίδευση στις περισσότερες περιπτώσεις. Για δεδομένα 3D, το Cellpose επαναχρησιμοποιεί έξυπνα το 2D μοντέλο του με προσέγγιση "2.5D", αποφεύγοντας την ανάγκη για πλήρως 3D επισημασμένα δεδομένα εκπαίδευσης, ενώ παρέχει όγκο τμηματοποίησης. Το Cellpose 2.0 εισήγαγε την επανεκπαίδευση με ανθρώπινη παρέμβαση, επιτρέποντας στους χρήστες να διορθώνουν χειροκίνητα τις προβλέψεις και να επανεκπαιδεύουν το μοντέλο στις δικές τους εικόνες για βελτιωμένη απόδοση σε συγκεκριμένα σύνολα δεδομένων.
Εγκατάσταση & Ρύθμιση
Δημιουργήστε ένα περιβάλλον Python χρησιμοποιώντας το conda:
conda create -n cellpose python=3.10Ενεργοποιήστε το περιβάλλον και εγκαταστήστε το Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeΞεκινώντας
Λειτουργία GUI
- Εκκινήστε το GUI εκτελώντας:
python -m cellpose - Σύρετε και αποθέστε αρχεία εικόνων (
.tif,.pngκ.ά.) στη διεπαφή - Επιλέξτε τύπο μοντέλου (π.χ. "cyto" για κυτταρόπλασμα ή "nuclei" για πυρήνες)
- Ορίστε την εκτιμώμενη διάμετρο κυττάρου ή αφήστε το Cellpose να αυτορυθμιστεί
- Κάντε κλικ για να ξεκινήσει η τμηματοποίηση και δείτε τα αποτελέσματα
Λειτουργία Python API
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Βελτίωση & Επανεκπαίδευση
- Μετά τη δημιουργία των μάσκων, διορθώστε την τμηματοποίηση στο GUI συγχωνεύοντας ή διαγράφοντας μάσκες χειροκίνητα
- Χρησιμοποιήστε τις ενσωματωμένες λειτουργίες εκπαίδευσης για επανεκπαίδευση με τα διορθωμένα παραδείγματα
- Βελτιωμένη απόδοση μοντέλου στο συγκεκριμένο σύνολο δεδομένων σας
Επεξεργασία 3D Δεδομένων
- Φορτώστε ένα TIFF πολλαπλών επιπέδων Z ή έναν όγκο δεδομένων
- Χρησιμοποιήστε τη σημαία
--Zstackστο GUI ή API για επεξεργασία ως 3D - Προαιρετικά βελτιώστε τις ροές 3D μέσω εξομάλυνσης ή εξειδικευμένων παραμέτρων για καλύτερη τμηματοποίηση
Περιορισμοί & Σκέψεις
- Ανταλλαγή Γενικότητας Μοντέλου: Παρότι το γενικό μοντέλο λειτουργεί ευρέως, ιδιαίτερα ασυνήθιστα σχήματα κυττάρων ή συνθήκες απεικόνισης μπορεί να απαιτούν επανεκπαίδευση.
- Προσπάθεια Επισημάνσεων: Η εκπαίδευση με ανθρώπινη παρέμβαση απαιτεί χειροκίνητες διορθώσεις, που μπορεί να είναι χρονοβόρες για μεγάλα σύνολα δεδομένων.
- Πολυπλοκότητα Εγκατάστασης: Η εγκατάσταση του GUI μπορεί να απαιτεί χρήση γραμμής εντολών, περιβάλλοντα conda και διαχείριση εξαρτήσεων Python — όχι πάντα απλή για μη προγραμματιστές.
- Μόνο για Επιτραπέζιους Υπολογιστές: Το Cellpose έχει σχεδιαστεί για χρήση σε επιτραπέζιους υπολογιστές· δεν υπάρχουν εγγενείς εφαρμογές για Android ή iOS.
Συχνές Ερωτήσεις
Όχι — το Cellpose παρέχει προεκπαιδευμένα, γενικά μοντέλα που συχνά λειτουργούν καλά χωρίς επανεκπαίδευση. Ωστόσο, για βέλτιστα αποτελέσματα σε ειδικά ή ασυνήθιστα δεδομένα, μπορείτε να επισημάνετε και να επανεκπαιδεύσετε χρησιμοποιώντας τις δυνατότητες ανθρώπινης παρέμβασης.
Ναι — υποστηρίζει 3D επαναχρησιμοποιώντας το 2D μοντέλο του (την αποκαλούμενη "2.5D" προσέγγιση), και μπορείτε να επεξεργαστείτε όγκους δεδομένων μέσω GUI ή API.
Συνιστάται έντονα GPU για ταχύτερη επεξεργασία και εκπαίδευση, ειδικά σε μεγάλα ή 3D σύνολα δεδομένων, αλλά το Cellpose μπορεί να τρέξει και σε μηχανές μόνο με CPU με μειωμένη απόδοση.
Στο GUI, ορίστε χειροκίνητα την εκτιμώμενη διάμετρο κυττάρου ή αφήστε το Cellpose να την αυτορυθμίσει. Μπορείτε να βελτιώσετε τα αποτελέσματα και να επανεκπαιδεύσετε αν η τμηματοποίηση δεν είναι βέλτιστη.
Ναι — οι νεότερες εκδόσεις (Cellpose 3) περιλαμβάνουν μοντέλα αποκατάστασης εικόνας για αποθορυβοποίηση, αποθόλωση και αύξηση ανάλυσης, βελτιώνοντας την ποιότητα της τμηματοποίησης πριν την επεξεργασία.
StarDist
| Δημιουργοί | Uwe Schmidt, Martin Weigert, Coleman Broaddus, και Gene Myers |
| Υποστηριζόμενες Πλατφόρμες |
|
| Υποστήριξη Γλώσσας | Ανοιχτού κώδικα έργο με τεκμηρίωση και κοινότητα κυρίως στα Αγγλικά |
| Μοντέλο Τιμολόγησης | Δωρεάν και ανοιχτού κώδικα. Αδειοδοτημένο υπό BSD-3-Clause |
Επισκόπηση
Το StarDist είναι ένα εργαλείο βαθιάς μάθησης για κατάτμηση αντικειμένων σε μικροσκοπικές εικόνες. Αναπαριστά κάθε αντικείμενο (όπως πυρήνες κυττάρων) ως αστέρας-κυρτό πολύγωνο σε 2D ή πολύεδρο σε 3D, επιτρέποντας ακριβή ανίχνευση και διαχωρισμό πυκνά συσκευασμένων ή επικαλυπτόμενων αντικειμένων. Με την ανθεκτική του αρχιτεκτονική, το StarDist χρησιμοποιείται ευρέως για αυτοματοποιημένη κατάτμηση κυττάρων και πυρήνων σε φθορίζουσα μικροσκοπία, ιστοπαθολογία και άλλες εφαρμογές βιοεικόνων.
Κύρια Χαρακτηριστικά
Εξαιρετικά ακριβής κατάτμηση αντικειμένων χρησιμοποιώντας αστέρες-κυρτά πολύγωνα (2D) και πολύεδρα (3D) για αξιόπιστη ανίχνευση αντικειμένων.
Αφιερωμένα μοντέλα για εικόνες 2D και τρισδιάστατα δεδομένα για ολοκληρωμένη ανάλυση μικροσκοπίας.
Έτοιμα προς χρήση μοντέλα για φθορίζοντες πυρήνες, ιστολογία με χρώση H&E και άλλα κοινά σενάρια απεικόνισης.
Ταξινόμηση ανιχνευμένων αντικειμένων σε διακριτές κατηγορίες (π.χ. διαφορετικοί τύποι κυττάρων) σε μία μόνο εκτέλεση κατάτμησης.
Αδιάλειπτη ενσωμάτωση με ImageJ/Fiji, QuPath και napari για προσβάσιμες ροές εργασίας με γραφικό περιβάλλον χρήστη.
Ολοκληρωμένη αξιολόγηση κατάτμησης αντικειμένων περιλαμβάνοντας ακρίβεια, ανάκληση, βαθμό F1 και πανοπτική ποιότητα.
Τεχνικό Υπόβαθρο
Αρχικά παρουσιάστηκε σε άρθρο MICCAI 2018, η βασική καινοτομία του StarDist είναι η πρόβλεψη ακτινικών αποστάσεων κατά μήκος σταθερών ακτίνων σε συνδυασμό με την πιθανότητα αντικειμένου για κάθε εικονοστοιχείο, επιτρέποντας ακριβή ανακατασκευή αστέρων-κυρτών σχημάτων. Αυτή η προσέγγιση διαχωρίζει αξιόπιστα αντικείμενα που αγγίζονται στενά και είναι δύσκολο να διαχωριστούν με παραδοσιακές μεθόδους βασισμένες σε εικονοστοιχεία ή πλαίσια οριοθέτησης.
Πρόσφατες εξελίξεις έχουν επεκτείνει το StarDist σε εικόνες ιστοπαθολογίας, επιτρέποντας όχι μόνο κατάτμηση πυρήνων αλλά και ταξινόμηση πολλαπλών κλάσεων των ανιχνευμένων αντικειμένων. Η μέθοδος πέτυχε κορυφαία απόδοση σε διαγωνισμούς όπως το CoNIC (Colon Nuclei Identification and Counting).
Λήψη ή Πρόσβαση
Εγκατάσταση & Ρύθμιση
Εγκαταστήστε το TensorFlow (έκδοση 1.x ή 2.x) ως προαπαιτούμενο για το StarDist.
Χρησιμοποιήστε pip για να εγκαταστήσετε το πακέτο StarDist Python:
pip install stardistΓια napari:
pip install stardist-napariΓια QuPath: Εγκαταστήστε την επέκταση StarDist σύροντας το αρχείο .jar μέσα στο QuPath.
Για ImageJ/Fiji: Χρησιμοποιήστε τον ενσωματωμένο διαχειριστή προσθέτων ή την χειροκίνητη εγκατάσταση μέσω του μενού προσθέτων.
Εκτέλεση Κατάτμησης
Φορτώστε ένα προεκπαιδευμένο μοντέλο, κανονικοποιήστε την εικόνα σας και εκτελέστε πρόβλεψη:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Ανοίξτε την εικόνα σας στο napari, επιλέξτε το πρόσθετο StarDist, διαλέξτε προεκπαιδευμένο ή προσαρμοσμένο μοντέλο και εκτελέστε πρόβλεψη απευθείας από το GUI.
Χρησιμοποιήστε το πρόσθετο StarDist από το μενού Προσθέτων για να εφαρμόσετε μοντέλο στο στοίβασμα εικόνων σας με φιλικό περιβάλλον.
Μετά την εγκατάσταση της επέκτασης, εκτελέστε ανίχνευση StarDist μέσω της κονσόλας σεναρίων ή του γραφικού περιβάλλοντος του QuPath για ανάλυση ιστοπαθολογίας.
Εκπαίδευση & Βελτιστοποίηση
Δημιουργήστε εικόνες ετικετών αληθινών δεδομένων όπου κάθε αντικείμενο επισημαίνεται μοναδικά. Χρησιμοποιήστε εργαλεία επισημάνσεων όπως LabKit, QuPath ή Fiji για να προετοιμάσετε το σύνολο δεδομένων σας.
Χρησιμοποιήστε το Python API του StarDist για να εκπαιδεύσετε νέο μοντέλο ή να βελτιστοποιήσετε υπάρχον με τα δικά σας επισημασμένα δεδομένα.
Επιλογές Μετα-Επεξεργασίας
- Εφαρμόστε καταστολή μη-μέγιστων (NMS) για να εξαλείψετε πλεονάζοντα υποψήφια σχήματα
- Χρησιμοποιήστε το StarDist OPP (Μετα-Επεξεργασία Αντικειμένων) για συγχώνευση μασκών σε μη αστέρες-κυρτά σχήματα
Περιορισμοί & Σκέψεις
- Η υπόθεση αστέρων-κυρτών μπορεί να μην μοντελοποιεί τέλεια πολύ μη-κυρτά ή πολύ ακανόνιστα σχήματα
- Πολυπλοκότητα εγκατάστασης: οι προσαρμοσμένες εγκαταστάσεις απαιτούν συμβατό μεταγλωττιστή C++ για κατασκευή επεκτάσεων
- Η επιτάχυνση GPU εξαρτάται από συμβατές εκδόσεις TensorFlow, CUDA και cuDNN
- Ορισμένοι χρήστες αναφέρουν προβλήματα εκτέλεσης του πρόσθετου ImageJ λόγω ρυθμίσεων Java
Συχνές Ερωτήσεις
Το StarDist λειτουργεί με ποικιλία τύπων εικόνων, συμπεριλαμβανομένων φθορισμού, φωτεινού πεδίου και ιστοπαθολογίας (π.χ. H&E), χάρη στα ευέλικτα προεκπαιδευμένα μοντέλα και την προσαρμοστικότητά του σε διαφορετικές απεικονιστικές μεθόδους.
Ναι — το StarDist υποστηρίζει τρισδιάστατη κατάτμηση αντικειμένων χρησιμοποιώντας αστέρες-κυρτά πολύεδρα για όγκους δεδομένων, επεκτείνοντας τις δυνατότητες 2D σε πλήρη 3D ανάλυση.
Όχι απαραίτητα. Υπάρχουν διαθέσιμα προεκπαιδευμένα μοντέλα που συχνά λειτουργούν καλά άμεσα. Ωστόσο, για εξειδικευμένα ή νέα δεδομένα, η επισημάνση και εκπαίδευση προσαρμοσμένων μοντέλων βελτιώνει σημαντικά την ακρίβεια.
Το StarDist ενσωματώνεται με napari, ImageJ/Fiji και QuPath, επιτρέποντάς σας να εκτελείτε κατάτμηση από γραφικό περιβάλλον χωρίς κωδικοποίηση. Υποστηρίζει επίσης άμεση χρήση Python API για προχωρημένες ροές εργασίας.
Το StarDist παρέχει ενσωματωμένες λειτουργίες για υπολογισμό κοινών μετρικών κατάτμησης αντικειμένων, όπως ακρίβεια, ανάκληση, βαθμό F1 και πανοπτική ποιότητα, για την αξιολόγηση της απόδοσης κατάτμησης.
SAM
Πληροφορίες Εφαρμογής
| Προγραμματιστής | Meta AI Research (FAIR) |
| Υποστηριζόμενες Συσκευές |
|
| Γλώσσα & Διαθεσιμότητα | Ανοιχτού κώδικα θεμελιώδες μοντέλο διαθέσιμο παγκοσμίως· τεκμηρίωση στα Αγγλικά |
| Τιμολόγηση | Δωρεάν — ανοιχτού κώδικα υπό την άδεια της Meta μέσω GitHub και ενσωμάτωσης στο MIB |
Γενική Επισκόπηση
Το SAM (Segment Anything Model) είναι ένα ισχυρό θεμελιώδες μοντέλο τεχνητής νοημοσύνης που δημιούργησε η Meta και επιτρέπει την αλληλεπιδραστική και αυτόματη τμηματοποίηση σχεδόν οποιουδήποτε αντικειμένου σε εικόνες. Χρησιμοποιώντας προτροπές όπως σημεία, πλαίσια οριοθέτησης ή αδρές μάσκες, το SAM παράγει μάσκες τμηματοποίησης χωρίς να απαιτείται επανεκπαίδευση για συγκεκριμένη εργασία. Στην έρευνα μικροσκοπίας, η ευελιξία του SAM έχει προσαρμοστεί για τμηματοποίηση κυττάρων, ανίχνευση οργανιδίων και ανάλυση ιστοπαθολογίας, προσφέροντας μια κλιμακούμενη λύση για ερευνητές που χρειάζονται ένα εργαλείο τμηματοποίησης γενικής χρήσης με δυνατότητα προτροπής.
Λεπτομερής Εισαγωγή
Αρχικά εκπαιδευμένο από τη Meta σε πάνω από 1 δισεκατομμύριο μάσκες σε 11 εκατομμύρια εικόνες, το SAM σχεδιάστηκε ως θεμελιώδες μοντέλο με δυνατότητα προτροπής για τμηματοποίηση με απόδοση "μηδενικού βολής" σε νέους τομείς. Στην έρευνα ιατρικής απεικόνισης, το SAM έχει αξιολογηθεί για τμηματοποίηση ολόκληρων διαφανειών παθολογίας, ανίχνευση όγκων και αναγνώριση πυρήνων κυττάρων. Ωστόσο, η απόδοσή του σε πυκνά συσκευασμένες περιπτώσεις — όπως οι πυρήνες κυττάρων — είναι μικτή: ακόμη και με εκτεταμένες προτροπές (π.χ. 20 κλικ ή πλαίσια), η τμηματοποίηση μηδενικού βολής μπορεί να δυσκολευτεί σε σύνθετες μικροσκοπικές εικόνες.
Για να αντιμετωπιστεί αυτός ο περιορισμός, έχουν αναπτυχθεί προσαρμογές συγκεκριμένου τομέα:
- SAMCell — Εκπαιδευμένο εκ νέου σε μεγάλα σύνολα δεδομένων μικροσκοπίας για ισχυρή τμηματοποίηση μηδενικού βολής σε διάφορους τύπους κυττάρων χωρίς επανεκπαίδευση ανά πείραμα
- μSAM — Επανεκπαιδευμένο σε πάνω από 17.000 χειροκίνητα σχολιασμένες μικροσκοπικές εικόνες για βελτίωση της ακρίβειας σε μικρές κυτταρικές δομές
Κύρια Χαρακτηριστικά
Ευέλικτη αλληλεπίδραση με χρήση σημείων, πλαισίων και μασκών για ακριβή έλεγχο.
Εκτελεί τμηματοποίηση χωρίς επανεκπαίδευση σε νέους τομείς εικόνων.
Προσαρμόσιμο για μικροσκοπία και ιστοπαθολογία μέσω λίγων δειγμάτων ή επανεκπαίδευσης με προτροπές.
Διαθέσιμο στο Microscopy Image Browser (MIB) με υποστήριξη 3D και ενδιάμεσης τμηματοποίησης.
Το IDCC-SAM επιτρέπει αυτόματη καταμέτρηση κυττάρων σε ανοσοκυτταροχημεία χωρίς χειροκίνητο σχολιασμό.
Λήψη ή Πρόσβαση
Οδηγός Χρήστη
- Ανοίξτε το Microscopy Image Browser και μεταβείτε στον πίνακα τμηματοποίησης SAM
- Ρυθμίστε τον διερμηνέα Python και επιλέξτε μεταξύ των μοντέλων SAM-1 ή SAM-2
- Για επιτάχυνση με GPU, επιλέξτε "cuda" στο περιβάλλον εκτέλεσης (συνιστάται για βέλτιστη απόδοση)
- Προτροπές σημείων: Κάντε κλικ σε ένα αντικείμενο για να ορίσετε θετικό σπόρο· χρησιμοποιήστε Shift + κλικ για επέκταση και Ctrl + κλικ για αρνητικούς σπόρους
- 3D στοίβες: Χρησιμοποιήστε τη λειτουργία Interactive 3D — κάντε κλικ σε μία τομή, μετακινηθείτε με shift-scroll και παρεμβάλετε σπόρους μεταξύ τομών
- Λειτουργία προσαρμογής: Αντικαταστήστε, προσθέστε, αφαιρέστε μάσκες ή δημιουργήστε νέο επίπεδο ανάλογα με τις ανάγκες
- Χρησιμοποιήστε την επιλογή "Αυτόματο τα πάντα" του MIB στον πίνακα SAM-2 για να τμηματοποιήσετε όλα τα ορατά αντικείμενα σε μια περιοχή
- Ελέγξτε και βελτιώστε τις μάσκες μετά την τμηματοποίηση, αν χρειάζεται
- Χρησιμοποιήστε ροές εργασίας επανεκπαίδευσης με βάση προτροπές (π.χ. "All-in-SAM") για δημιουργία σχολιασμών σε επίπεδο pixel από αραιές προτροπές χρήστη
- Για καταμέτρηση κυττάρων, εφαρμόστε το IDCC-SAM, που χρησιμοποιεί το SAM σε ροή μηδενικού βολής με μετα-επεξεργασία
- Για υψηλή ακρίβεια στην τμηματοποίηση κυττάρων, χρησιμοποιήστε το SAMCell, εκπαιδευμένο εκ νέου σε εικόνες μικροσκοπίας κυττάρων
Περιορισμοί & Σκέψεις
- Η απόδοση μηδενικού βολής είναι ασυνεπής σε πυκνές ή επικαλυπτόμενες δομές χωρίς εκπαίδευση τομέα
- Η ποιότητα της τμηματοποίησης εξαρτάται σημαντικά από το σχεδιασμό και τη στρατηγική προτροπών
- Συνιστάται έντονα GPU· η εκτέλεση σε CPU είναι πολύ αργή
- Δυσκολίες με εικόνες ολόκληρης διαφάνειας πολύ υψηλής ανάλυσης και πολυκλιμακωτές δομές ιστών
- Η επανεκπαίδευση ή προσαρμογή του SAM για μικροσκοπία μπορεί να απαιτεί γνώσεις μηχανικής μάθησης
Συχνές Ερωτήσεις
Ναι — μέσω προσαρμογών όπως το SAMCell, που εκπαιδεύει εκ νέου το SAM σε σύνολα δεδομένων μικροσκοπίας ειδικά για εργασίες τμηματοποίησης κυττάρων.
Όχι πάντα. Με το IDCC-SAM, μπορείτε να κάνετε καταμέτρηση κυττάρων μηδενικού βολής χωρίς χειροκίνητο σχολιασμό.
Χρησιμοποιήστε επανεκπαίδευση με βάση προτροπές (π.χ. "All-in-SAM") ή προεκπαιδευμένες εκδόσεις μικροσκοπίας όπως το μSAM, που έχει εκπαιδευτεί σε πάνω από 17.000 σχολιασμένες μικροσκοπικές εικόνες.
Παρότι είναι δυνατή η εκτέλεση σε CPU, η χρήση GPU συνιστάται έντονα για πρακτική ταχύτητα εκτέλεσης και αλληλεπιδραστική τμηματοποίηση σε πραγματικό χρόνο.
Ναι — η ενσωμάτωση SAM-2 στο MIB υποστηρίζει τρισδιάστατη τμηματοποίηση με παρεμβολή σπόρων μεταξύ τομών για ογκομετρική ανάλυση.
AxonDeepSeg
| Δημιουργός | Εργαστήριο NeuroPoly στο Polytechnique Montréal και Université de Montréal |
| Υποστηριζόμενες Πλατφόρμες |
|
| Γλώσσα | Τεκμηρίωση στα Αγγλικά· εργαλείο ανοιχτού κώδικα με παγκόσμια χρήση |
| Τιμολόγηση | Δωρεάν και ανοιχτού κώδικα |
Επισκόπηση
Το AxonDeepSeg είναι ένα εργαλείο με τεχνητή νοημοσύνη για αυτόματο τεμαχισμό αξόνων και μυελίνης σε μικροσκοπικές εικόνες. Χρησιμοποιώντας συνελικτικά νευρωνικά δίκτυα, παρέχει ακριβή τρισδιάστατο τεμαχισμό τριών κατηγοριών (άξονας, μυελίνη, φόντο) σε πολλαπλές μεθόδους απεικόνισης, όπως TEM, SEM και μικροσκόπιο φωτεινού πεδίου. Με την αυτοματοποίηση μορφομετρικών μετρήσεων όπως διάμετρος άξονα, λόγος g και πάχος μυελίνης, το AxonDeepSeg απλοποιεί την ποσοτική ανάλυση στην έρευνα νευροεπιστημών, μειώνοντας σημαντικά τον χρόνο χειροκίνητης σήμανσης και βελτιώνοντας την αναπαραγωγιμότητα.
Κύρια Χαρακτηριστικά
Έτοιμα προς χρήση μοντέλα βελτιστοποιημένα για τις μεθόδους TEM, SEM και μικροσκόπιο φωτεινού πεδίου.
Ακριβής ταξινόμηση περιοχών άξονα, μυελίνης και φόντου σε μικροσκοπικές εικόνες.
Αυτόματος υπολογισμός διαμέτρου άξονα, λόγου g, πάχους μυελίνης και μετρικών πυκνότητας.
Η ενσωμάτωση με το Napari GUI επιτρέπει χειροκίνητη βελτίωση των μάσκων τεμαχισμού για μεγαλύτερη ακρίβεια.
Ενσωματώνεται ομαλά σε προσαρμοσμένες ροές εργασίας για ανάλυση νευρικού ιστού μεγάλης κλίμακας.
Πλήρεις δοκιμαστικές εντολές εξασφαλίζουν αναπαραγωγιμότητα και αξιόπιστα αποτελέσματα τεμαχισμού.
Τεχνικές Λεπτομέρειες
Αναπτυγμένο από το εργαστήριο NeuroPoly, το AxonDeepSeg αξιοποιεί τη βαθιά μάθηση για να προσφέρει τεμαχισμό υψηλής ακρίβειας σε εφαρμογές νευροεπιστημών. Διατίθενται προεκπαιδευμένα μοντέλα για διαφορετικές μικροσκοπικές μεθόδους, εξασφαλίζοντας ευελιξία σε τεχνικές απεικόνισης. Το εργαλείο ενσωματώνεται με το Napari, επιτρέποντας διαδραστικές διορθώσεις μάσκας τεμαχισμού, που βελτιώνουν την ακρίβεια σε απαιτητικά σύνολα δεδομένων. Το AxonDeepSeg υπολογίζει βασικές μορφομετρικές μετρικές, υποστηρίζοντας μελέτες υψηλής απόδοσης της δομής και παθολογίας νευρικού ιστού. Το πλαίσιο του βασίζεται σε Python, επιτρέποντας ενσωμάτωση σε προσαρμοσμένες ροές εργασίας για ανάλυση μεγάλης κλίμακας της μορφολογίας αξόνων και μυελίνης.
Λήψη ή Πρόσβαση
Εγκατάσταση & Ρύθμιση
Βεβαιωθείτε ότι είναι εγκατεστημένη η Python 3.8 ή νεότερη έκδοση, στη συνέχεια εγκαταστήστε το AxonDeepSeg και το Napari μέσω pip:
pip install axondeepseg napariΕκτελέστε τα παρεχόμενα σενάρια δοκιμών για να επιβεβαιώσετε ότι όλα τα στοιχεία είναι σωστά εγκατεστημένα και λειτουργούν.
Εισάγετε μικροσκοπικές εικόνες (TEM, SEM ή φωτεινού πεδίου) στο Napari ή στο περιβάλλον Python σας.
Επιλέξτε το κατάλληλο προεκπαιδευμένο μοντέλο για τη μέθοδο απεικόνισης και εκτελέστε τον τεμαχισμό για να δημιουργήσετε μάσκες άξονα και μυελίνης.
Υπολογίστε αυτόματα μορφομετρικές μετρήσεις όπως διάμετρο άξονα, λόγο g, πυκνότητα και πάχος μυελίνης, και εξάγετε τα αποτελέσματα σε μορφή CSV.
Χρησιμοποιήστε το περιβάλλον Napari GUI για χειροκίνητη διόρθωση των μάσκων τεμαχισμού όπου χρειάζεται, συγχωνεύοντας ή διαγράφοντας μάσκες για βελτιωμένη ακρίβεια.
Σημαντικές Παρατηρήσεις
- Η απόδοση μπορεί να μειωθεί σε νέες ή μη εκπαιδευμένες μεθόδους απεικόνισης
- Ενδέχεται να απαιτούνται χειροκίνητες διορθώσεις σε απαιτητικές ή σύνθετες περιοχές
- Συνιστάται GPU για ταχύτερη επεξεργασία μεγάλων συνόλων δεδομένων· υποστηρίζεται και επεξεργασία με CPU
Συχνές Ερωτήσεις
Το AxonDeepSeg υποστηρίζει τις μεθόδους TEM (Μεταφορική Ηλεκτρονική Μικροσκοπία), SEM (Σαρωτική Ηλεκτρονική Μικροσκοπία) και μικροσκόπιο φωτεινού πεδίου με προεκπαιδευμένα μοντέλα βελτιστοποιημένα για κάθε μέθοδο.
Ναι, το AxonDeepSeg είναι εντελώς δωρεάν και ανοιχτού κώδικα, διαθέσιμο για ακαδημαϊκή και εμπορική χρήση.
Ναι, το AxonDeepSeg υπολογίζει αυτόματα διάμετρο άξονα, λόγο g, πάχος μυελίνης και μετρικές πυκνότητας από τις τεμαχισμένες εικόνες.
Η χρήση GPU συνιστάται για ταχύτερο τεμαχισμό μεγάλων συνόλων δεδομένων, αλλά υποστηρίζεται και επεξεργασία με CPU για μικρότερες αναλύσεις.
Ναι, η ενσωμάτωση με το περιβάλλον Napari GUI επιτρέπει διαδραστικές διορθώσεις και βελτιώσεις των μάσκων τεμαχισμού για μεγαλύτερη ακρίβεια σε απαιτητικές περιοχές.
Ilastik
| Δημιουργός | Η ομάδα Ilastik στο Ευρωπαϊκό Εργαστήριο Μοριακής Βιολογίας (EMBL) και συνεργαζόμενοι ακαδημαϊκοί φορείς |
| Υποστηριζόμενες Πλατφόρμες |
|
| Γλώσσα | Αγγλικά |
| Τιμολόγηση | Δωρεάν και ανοιχτού κώδικα |
Επισκόπηση
Το Ilastik είναι ένα ισχυρό εργαλείο με τεχνητή νοημοσύνη για διαδραστικό διαχωρισμό εικόνων, ταξινόμηση και ανάλυση δεδομένων μικροσκοπίας. Χρησιμοποιώντας τεχνικές μηχανικής μάθησης όπως οι ταξινομητές Random Forest, επιτρέπει στους ερευνητές να διαχωρίζουν εικονοστοιχεία, να ταξινομούν αντικείμενα, να παρακολουθούν κύτταρα με την πάροδο του χρόνου και να εκτελούν καταμέτρηση πυκνότητας σε 2D και 3D σύνολα δεδομένων. Με τη διαισθητική διεπαφή και την ανατροφοδότηση σε πραγματικό χρόνο, το Ilastik είναι προσβάσιμο σε επιστήμονες χωρίς γνώσεις προγραμματισμού και χρησιμοποιείται ευρέως στη βιολογία κυττάρου, τη νευροεπιστήμη και την ιατρική απεικόνιση.
Κύρια Χαρακτηριστικά
Ανατροφοδότηση σε πραγματικό χρόνο καθώς επισημαίνετε αντιπροσωπευτικές περιοχές για άμεσα αποτελέσματα διαχωρισμού.
Κατηγοριοποιήστε διαχωρισμένες δομές βάσει μορφολογικών και εντατικών χαρακτηριστικών.
Παρακολουθήστε την κίνηση και διαίρεση κυττάρων σε 2D και 3D πειράματα χρονικής λήψης μικροσκοπίας.
Ποσοτικοποιήστε πυκνές περιοχές χωρίς ρητό διαχωρισμό μεμονωμένων αντικειμένων.
Ημι-αυτόματος διαχωρισμός για σύνθετους 3D όγκους με διαισθητική αλληλεπίδραση.
Επεξεργαστείτε πολλαπλές εικόνες αυτόματα χρησιμοποιώντας λειτουργία χωρίς γραφικό περιβάλλον.
Λήψη
Οδηγός Έναρξης
Κατεβάστε το Ilastik για το λειτουργικό σας σύστημα από την επίσημη ιστοσελίδα. Το πακέτο περιλαμβάνει όλες τις απαραίτητες εξαρτήσεις Python, οπότε ακολουθήστε τις οδηγίες εγκατάστασης για την πλατφόρμα σας.
Ανοίξτε το Ilastik και επιλέξτε τη ροή εργασίας ανάλυσης: Ταξινόμηση Εικονοστοιχείων, Ταξινόμηση Αντικειμένων, Παρακολούθηση ή Καταμέτρηση Πυκνότητας. Φορτώστε το σύνολο εικόνων σας, που μπορεί να περιλαμβάνει πολυκαναλικές, 3D ή χρονικές εικόνες.
Επισημάνετε μερικά αντιπροσωπευτικά εικονοστοιχεία ή αντικείμενα στις εικόνες σας. Ο ταξινομητής Random Forest του Ilastik μαθαίνει από αυτές τις επισημάνσεις και προβλέπει αυτόματα ετικέτες σε ολόκληρο το σύνολο δεδομένων.
Εφαρμόστε το εκπαιδευμένο μοντέλο για να διαχωρίσετε ή να ταξινομήσετε ολόκληρο το σύνολο δεδομένων σας. Εξάγετε τα αποτελέσματα ως επισημασμένες εικόνες, χάρτες πιθανοτήτων ή ποσοτικούς πίνακες για περαιτέρω ανάλυση και οπτικοποίηση.
Χρησιμοποιήστε τη λειτουργία χωρίς γραφικό περιβάλλον του Ilastik για αυτόματη επεξεργασία πολλαπλών εικόνων χωρίς χειροκίνητη παρέμβαση, ιδανική για μεγάλης κλίμακας αναλυτικές ροές εργασίας.
Περιορισμοί & Σκέψεις
- Η διαδραστική επισήμανση μπορεί να είναι χρονοβόρα για πολύ μεγάλα σύνολα δεδομένων
- Η ακρίβεια εξαρτάται από την ποιότητα και αντιπροσωπευτικότητα των επισημάνσεων του χρήστη
- Απαιτήσεις μνήμης — πολύ υψηλής ανάλυσης ή πολυ-γιγαμπαϊτικά σύνολα δεδομένων μπορεί να απαιτούν σημαντική RAM
- Σύνθετα δεδομένα — οι ταξινομητές Random Forest μπορεί να υστερούν σε σχέση με βαθιά νευρωνικά δίκτυα σε πολύ μεταβλητά ή σύνθετα δεδομένα απεικόνισης
Συχνές Ερωτήσεις
Ναι, το Ilastik υποστηρίζει πλήρως 3D όγκους και πειράματα χρονικής λήψης για διαχωρισμό, παρακολούθηση και ποσοτική ανάλυση σε πολλαπλά χρονικά σημεία.
Ναι, το Ilastik είναι εντελώς δωρεάν και ανοιχτού κώδικα, διαθέσιμο σε όλους τους χρήστες χωρίς περιορισμούς αδειοδότησης.
Όχι, το Ilastik παρέχει μια διαισθητική γραφική διεπαφή με ανατροφοδότηση σε πραγματικό χρόνο, καθιστώντας το προσβάσιμο σε ερευνητές χωρίς γνώσεις προγραμματισμού. Προχωρημένοι χρήστες μπορούν επίσης να χρησιμοποιήσουν τη μαζική επεξεργασία μέσω γραμμής εντολών.
Ναι, η ειδική ροή εργασίας παρακολούθησης επιτρέπει την ανάλυση κίνησης και διαίρεσης κυττάρων σε 2D και 3D χρονικά σύνολα δεδομένων με αυτόματη παρακολούθηση γενεαλογίας.
Τα αποτελέσματα διαχωρισμού μπορούν να εξαχθούν ως επισημασμένες εικόνες, χάρτες πιθανοτήτων ή ποσοτικοί πίνακες, επιτρέποντας ομαλή ενσωμάτωση με εργαλεία ανάλυσης και λογισμικό οπτικοποίησης.
Αυτά τα εργαλεία καλύπτουν επίπεδα από αρχάριους έως ειδικούς. Πολλά είναι δωρεάν και ανοιχτού κώδικα, διευκολύνοντας αναπαραγώγιμες και κοινόχρηστες ροές εργασίας τεχνητής νοημοσύνης στην ερευνητική κοινότητα.
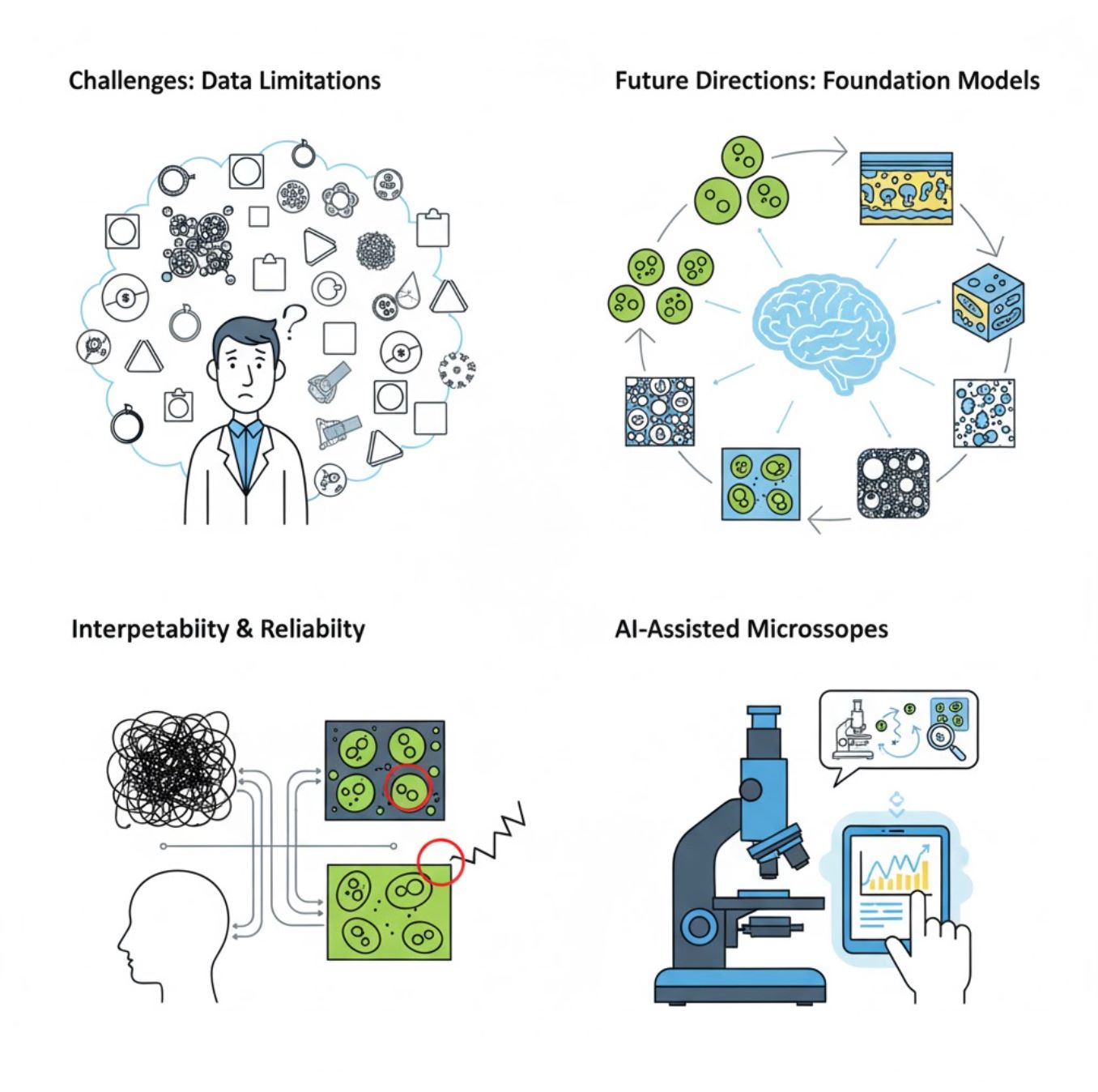
Προκλήσεις και Μελλοντικές Κατευθύνσεις
Τρέχουσες Προκλήσεις
Αναδυόμενες Τάσεις
Μοντέλα Θεμελιώδους Όρασης
Τα συστήματα τεχνητής νοημοσύνης επόμενης γενιάς υπόσχονται να μειώσουν την ανάγκη για εκπαίδευση ειδική σε κάθε εργασία.
- Μοντέλα όπως το SAM και συστήματα βασισμένα σε CLIP
- Ένα AI που διαχειρίζεται πολλές εργασίες μικροσκοπίας
- Ταχύτερη ανάπτυξη και προσαρμογή
Μικροσκόπια με Υποστήριξη Τεχνητής Νοημοσύνης
Πλήρως αυτόνομα και έξυπνα συστήματα μικροσκοπίας γίνονται πραγματικότητα.
- Έλεγχος με φυσική γλώσσα μέσω μεγάλων γλωσσικών μοντέλων (LLMs)
- Πλήρως αυτοματοποιημένοι βρόχοι ανατροφοδότησης
- Δημοκρατικοποιεί την πρόσβαση σε προηγμένη μικροσκοπία
Βασικά Συμπεράσματα
- Η τεχνητή νοημοσύνη μετασχηματίζει γρήγορα την επεξεργασία εικόνων μικροσκοπίου με βελτιωμένη ακρίβεια και αυτοματοποίηση
- Η βαθιά μάθηση υπερτερεί της παραδοσιακής μηχανικής μάθησης σε πολύπλοκες, μεταβλητές εικόνες μικροσκοπίας
- Τα CNNs μαθαίνουν αυτόματα ιεραρχικά χαρακτηριστικά από ακατέργαστα pixels για ανθεκτική ανάλυση
- Βασικές εφαρμογές περιλαμβάνουν τμηματοποίηση, ταξινόμηση, παρακολούθηση, αποθορυβοποίηση και αυτοματοποιημένη απόκτηση
- Η επιτυχία εξαρτάται από ποιοτικά δεδομένα και προσεκτική επικύρωση από ειδικούς
- Τα μοντέλα θεμελιώδους όρασης και τα μικροσκόπια με υποστήριξη τεχνητής νοημοσύνης αντιπροσωπεύουν το μέλλον του πεδίου
Με συνεχή πρόοδο και προσπάθειες της κοινότητας (εργαλεία ανοιχτού κώδικα, κοινά σύνολα δεδομένων), η τεχνητή νοημοσύνη θα γίνει ολοένα και περισσότερο βασικό μέρος του "ματιού" του μικροσκοπίου, βοηθώντας τους επιστήμονες να δουν το αόρατο.







No comments yet. Be the first to comment!