KI in der Mikroskop-Bildverarbeitung
KI revolutioniert die Mikroskop-Bildverarbeitung mit leistungsstarken Funktionen wie präziser Segmentierung, Rauschreduzierung, Superauflösung und automatisierter Bildaufnahme. Dieser Artikel stellt wichtige KI-Werkzeuge und aufkommende Trends in der wissenschaftlichen Forschung vor.
KI-Techniken revolutionieren die Mikroskopie durch Optimierung der Bildaufnahme und Automatisierung der Analyse. In modernen intelligenten Mikroskopen können KI-Module Bildgebungsparameter in Echtzeit anpassen (z. B. Fokus, Beleuchtung), um Photobleichen zu minimieren und das Signal zu verbessern. Gleichzeitig können Deep-Learning-Algorithmen komplexe Bilddaten durchforsten, um verborgene biologische Erkenntnisse zu gewinnen und sogar Bilder mit anderen Daten (z. B. Genomik) zu verknüpfen.
KI-Methoden: Maschinelles Lernen vs. Deep Learning
KI-Methoden reichen vom klassischen Maschinellen Lernen (ML) bis zum modernen Deep Learning (DL). Jeder Ansatz hat eigene Stärken und Schwächen:
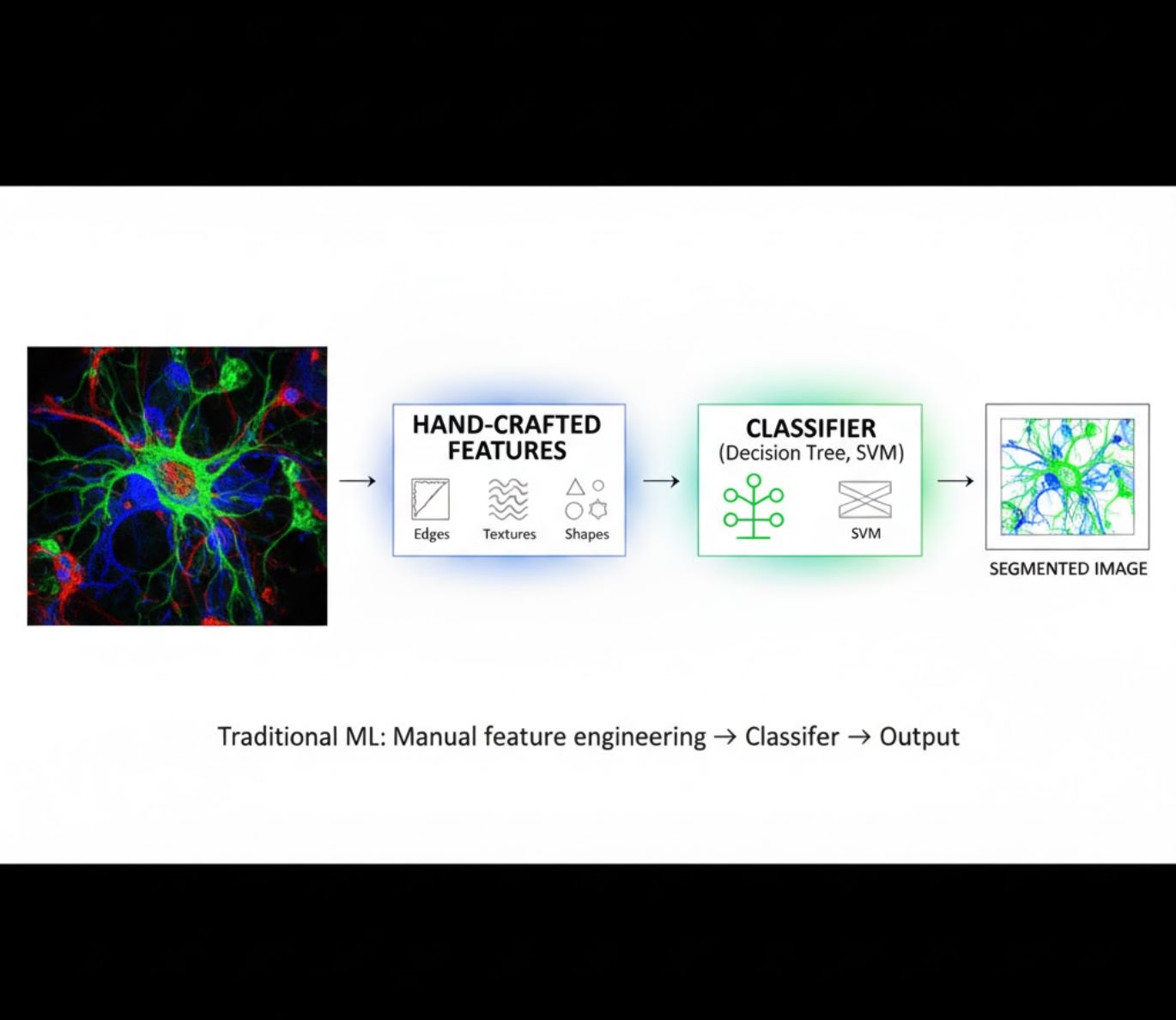
Handgefertigte Merkmale
- Forschende erstellen manuell Bildmerkmale (Kanten, Texturen, Formen)
- Merkmale werden an Klassifikatoren (Entscheidungsbäume, SVM) übergeben
- Schnelles Training
- Schwierig bei komplexen oder verrauschten Bildern
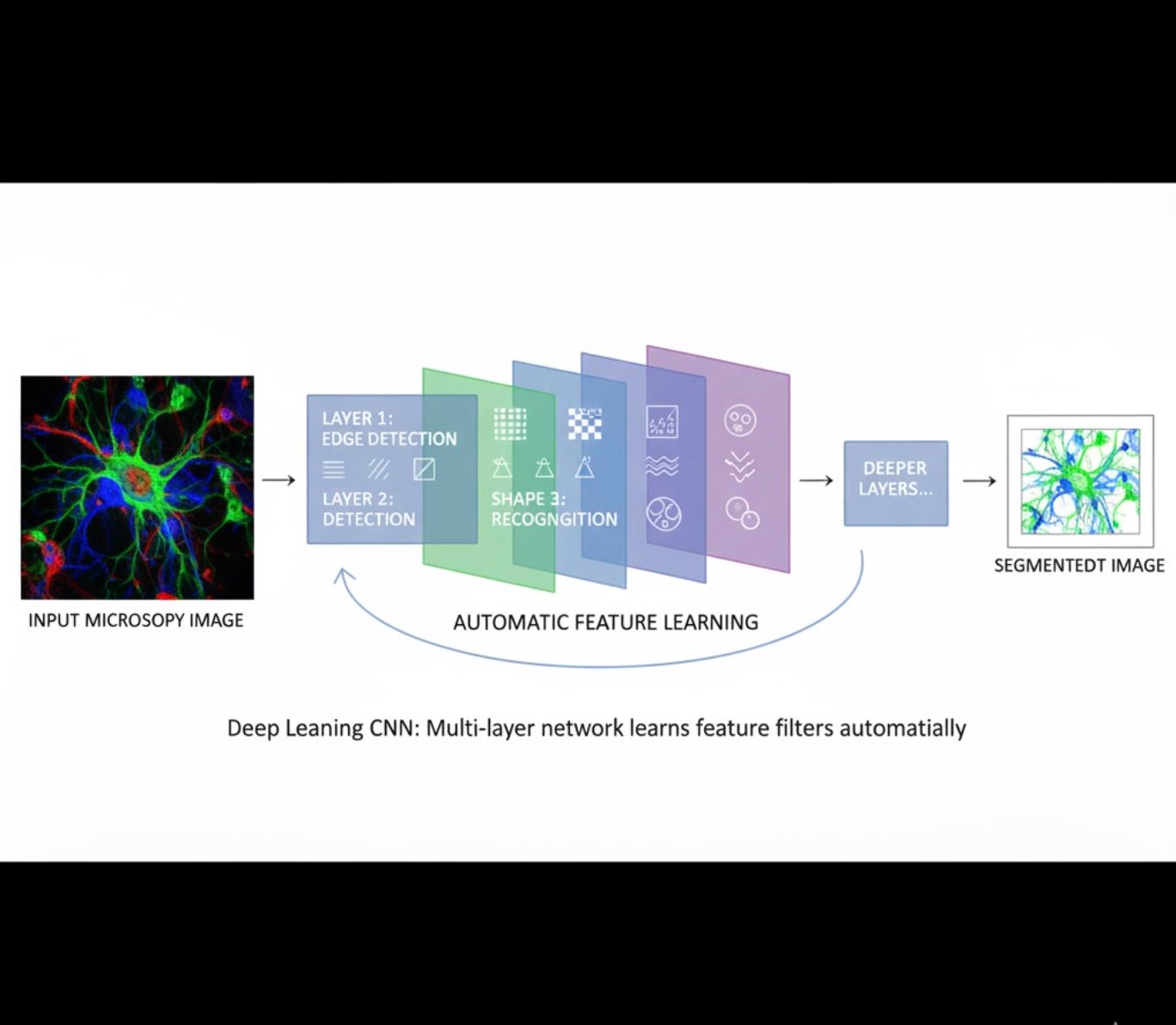
Automatisches Merkmalslernen
- Mehrschichtige neuronale Netze (CNNs) lernen Merkmale automatisch
- End-to-End-Lernen direkt von Rohpixeln
- Deutlich robuster gegenüber Variationen
- Erfasst zuverlässig komplexe Texturen und Strukturen
Funktionsweise von CNNs: Convolutional Neural Networks wenden aufeinanderfolgende Filter auf Mikroskopbilder an, lernen einfache Muster (Kanten) in frühen Schichten und komplexe Strukturen (Zellformen, Texturen) in tieferen Schichten zu erkennen. Dieses hierarchische Lernen macht DL besonders robust, selbst wenn Intensitätsprofile stark variieren.
Visueller Vergleich: ML- vs. DL-Pipelines
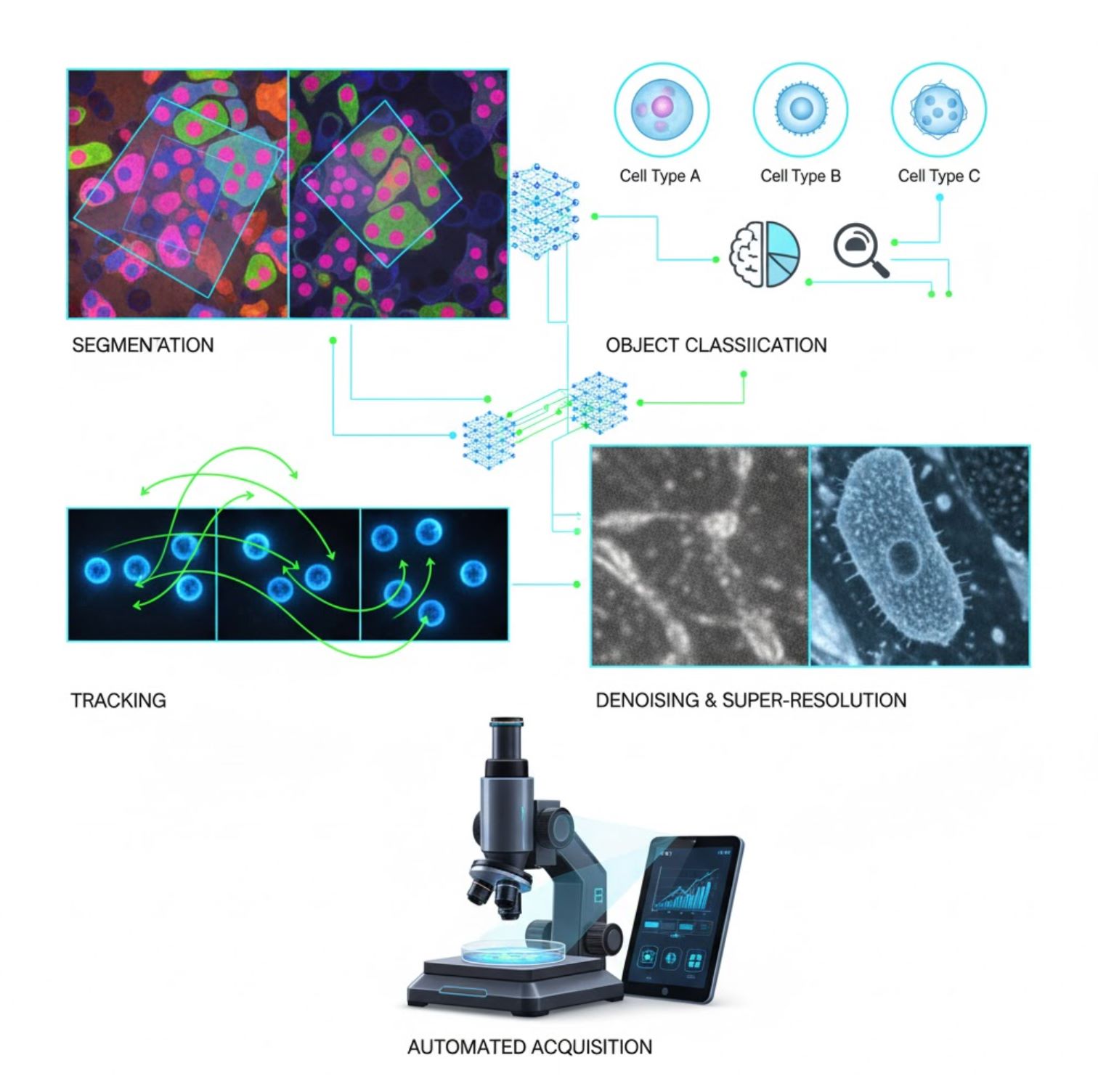
Wichtige KI-Anwendungen in der Mikroskopie
KI ist heute in vielen bildverarbeitenden Aufgaben im Mikroskopie-Workflow integriert:
Segmentierung
Aufteilung von Bildern in Regionen (z. B. Identifikation jeder Zelle oder jedes Zellkerns). Tiefe Netzwerke wie U-Net sind darin besonders leistungsfähig.
- Semantische Segmentierung: Pixelgenaue Klassenzuordnung
- Instanzsegmentierung: Trennung einzelner Objekte
- Hohe Genauigkeit bei überfüllten oder dunklen Bildern
- Vision-Foundation-Modelle (z. B. μSAM) sind jetzt für Mikroskopie adaptiert
Objektklassifikation
Nach der Segmentierung klassifiziert KI jedes Objekt mit hoher Präzision.
- Zelltypbestimmung
- Bestimmung des Mitose-Stadiums
- Erkennung pathologischer Indikatoren
- Unterscheidet subtile Phänotypen, die manuell schwer quantifizierbar sind
Tracking
In der Zeitraffer-Mikroskopie verfolgt KI Zellen oder Partikel über Bildfolgen mit beispielloser Genauigkeit.
- Deep Learning verbessert die Tracking-Genauigkeit dramatisch
- Ermöglicht zuverlässige Analyse bewegter Zellen
- Erfasst dynamische biologische Prozesse
Rauschunterdrückung & Superauflösung
KI-Modelle verbessern die Bildqualität durch Entfernung von Rauschen und Unschärfe.
- Physikalisch informierte Deep-Modelle lernen Mikroskopoptik
- Rekonstruiert schärfere, artefaktfreie Bilder
- Höhere Auflösung mit weniger Artefakten im Vergleich zu traditionellen Methoden
Automatisierte Aufnahme
KI steuert das Mikroskop selbst in Echtzeit.
- Analysiert Live-Bilder, um intelligente Entscheidungen zu treffen
- Passt Fokus automatisch an und scannt interessante Bereiche
- Reduziert Phototoxizität und spart Zeit
- Ermöglicht Hochdurchsatz- und adaptive Bildgebungs-Experimente
Beliebte KI-Werkzeuge in der Mikroskop-Bildverarbeitung
<ITEM_DESCRIPTION>Ein umfangreiches Ökosystem an Werkzeugen unterstützt KI in der Mikroskopie. Forscher haben sowohl allgemein einsetzbare als auch spezialisierte Software entwickelt, viele davon Open-Source:</ITEM_DESCRIPTION>
Cellpose
| Entwickler | Carsen Stringer und Marius Pachitariu (MouseLand-Forschungsgruppe) |
| Unterstützte Plattformen |
Erfordert Python (pip/conda Installation). GUI nur auf Desktop verfügbar. |
| Sprachunterstützung | Englische Dokumentation; weltweit in Forschungslaboren etabliert |
| Preismodell | Kostenlos und Open Source unter BSD-3-Clause Lizenz |
Überblick
Cellpose ist ein fortschrittliches, auf Deep Learning basierendes Segmentierungswerkzeug für Mikroskopiebilder. Als generischer Algorithmus segmentiert es präzise verschiedene Zelltypen (Kerne, Zytoplasma usw.) über unterschiedliche Bildgebungsverfahren hinweg, ohne dass ein Nachtraining des Modells nötig ist. Mit Mensch-in-der-Schleife-Funktionalitäten können Forschende Ergebnisse verfeinern, das Modell an eigene Daten anpassen und das System für 2D- und 3D-Bildgebungs-Workflows einsetzen.
Hauptmerkmale
Funktioniert sofort für eine Vielzahl von Zelltypen, Färbungen und Bildgebungsverfahren ohne individuelles Training.
Unterstützt vollständige 3D-Stapel mit einem „2,5D“-Ansatz, der 2D-Modelle für volumetrische Daten wiederverwendet.
Manuelle Korrektur der Segmentierungsergebnisse und Nachtraining des Modells mit eigenen Daten für bessere Genauigkeit.
Zugriff über Python-API, Kommandozeilenschnittstelle oder grafische Benutzeroberfläche für flexible Arbeitsabläufe.
Rauschunterdrückung, Entschärfung und Hochskalierung zur Verbesserung der Bildqualität vor der Segmentierung.
Download oder Zugriff
Technischer Hintergrund
Cellpose wurde in einer wegweisenden Studie von Stringer, Wang, Michaelos und Pachitariu vorgestellt und an einem großen, sehr vielfältigen Datensatz mit über 70.000 segmentierten Objekten trainiert. Diese Vielfalt ermöglicht es dem Modell, über Zellformen, Größen und Mikroskopieeinstellungen hinweg zu generalisieren und reduziert den Bedarf an individuellem Training in den meisten Anwendungsfällen erheblich. Für 3D-Daten nutzt Cellpose clever sein 2D-Modell in einer „2,5D“-Manier, wodurch vollständig 3D-annotierte Trainingsdaten entfallen, während dennoch volumetrische Segmentierung möglich ist. Cellpose 2.0 führte das Mensch-in-der-Schleife-Nachtraining ein, das Nutzern erlaubt, Vorhersagen manuell zu korrigieren und das Modell mit eigenen Bildern für bessere Leistung auf spezifischen Datensätzen nachzutrainieren.
Installation & Einrichtung
Richten Sie eine Python-Umgebung mit conda ein:
conda create -n cellpose python=3.10Aktivieren Sie die Umgebung und installieren Sie Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeErste Schritte
GUI-Modus
- Starten Sie die GUI mit:
python -m cellpose - Ziehen Sie Bilddateien (
.tif,.pngusw.) per Drag & Drop in die Oberfläche - Wählen Sie den Modelltyp (z. B. „cyto“ für Zytoplasma oder „nuclei“ für Zellkerne)
- Geben Sie den geschätzten Zell-Durchmesser an oder lassen Sie Cellpose automatisch kalibrieren
- Klicken Sie zum Start der Segmentierung und zur Anzeige der Ergebnisse
Python-API-Modus
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Verfeinern & Nachtrainieren
- Nach der Maskenerstellung korrigieren Sie die Segmentierung in der GUI durch manuelles Zusammenführen oder Löschen von Masken
- Nutzen Sie die integrierten Trainingsfunktionen, um mit korrigierten Beispielen nachzutrainieren
- Verbesserte Modellleistung für Ihren spezifischen Datensatz
3D-Daten verarbeiten
- Laden Sie einen Multi-Z-TIFF oder volumetrischen Stapel
- Verwenden Sie das
--Zstack-Flag in GUI oder API, um als 3D zu verarbeiten - Optional können Sie 3D-Flows durch Glättung oder spezielle Parameter für bessere Segmentierung verfeinern
Einschränkungen & Hinweise
- Modell-Generizität vs. Spezialisierung: Obwohl das generische Modell breit einsetzbar ist, können sehr ungewöhnliche Zellformen oder Bildgebungsbedingungen ein Nachtraining erfordern.
- Annotierungsaufwand: Mensch-in-der-Schleife-Training erfordert manuelle Korrekturen, was bei großen Datensätzen zeitintensiv sein kann.
- Installationskomplexität: Die GUI-Installation kann Kommandozeilen-Nutzung, conda-Umgebungen und Verwaltung von Python-Abhängigkeiten erfordern – nicht immer einfach für Nicht-Programmierer.
- Nur Desktop: Cellpose ist für den Desktop-Einsatz konzipiert; native Android- oder iOS-Apps sind nicht verfügbar.
Häufig gestellte Fragen
Nein – Cellpose stellt vortrainierte, generische Modelle bereit, die oft ohne Nachtraining gut funktionieren. Für optimale Ergebnisse bei speziellen oder ungewöhnlichen Daten können Sie jedoch annotieren und mit den Mensch-in-der-Schleife-Funktionen nachtrainieren.
Ja – es unterstützt 3D durch Wiederverwendung seines 2D-Modells (sogenanntes „2,5D“) und Sie können volumetrische Stapel über GUI oder API verarbeiten.
Eine GPU wird für schnellere Inferenz und Training, insbesondere bei großen oder 3D-Datensätzen, stark empfohlen, aber Cellpose kann auch auf reinen CPU-Maschinen mit geringerer Leistung laufen.
In der GUI geben Sie den geschätzten Zell-Durchmesser manuell ein oder lassen Cellpose ihn automatisch kalibrieren. Sie können Ergebnisse verfeinern und bei nicht optimaler Segmentierung nachtrainieren.
Ja – neuere Versionen (Cellpose 3) enthalten Bildrestaurierungsmodelle für Rauschunterdrückung, Entschärfung und Hochskalierung, um die Segmentierungsqualität vor der Verarbeitung zu verbessern.
StarDist
| Entwickler | Uwe Schmidt, Martin Weigert, Coleman Broaddus und Gene Myers |
| Unterstützte Plattformen |
|
| Sprachunterstützung | Open-Source-Projekt mit Dokumentation und Community hauptsächlich in Englisch |
| Preismodell | Kostenlos und Open Source. Lizenziert unter BSD-3-Clause |
Überblick
StarDist ist ein Deep-Learning-Werkzeug zur Instanzsegmentierung in Mikroskopiebildern. Es stellt jedes Objekt (z. B. Zellkerne) als sternkonvexes Polygon in 2D oder Polyeder in 3D dar, was eine präzise Erkennung und Trennung dicht gepackter oder überlappender Objekte ermöglicht. Mit seiner robusten Architektur wird StarDist weit verbreitet für die automatisierte Zell- und Kernsegmentierung in der Fluoreszenzmikroskopie, Histopathologie und anderen Bio-Bildanalyse-Anwendungen eingesetzt.
Hauptmerkmale
Hochpräzise Instanzsegmentierung mit sternkonvexen Polygonen (2D) und Polyedern (3D) für zuverlässige Objekterkennung.
Spezialisierte Modelle für 2D-Bilder und 3D-volumetrische Daten für umfassende Mikroskopieanalysen.
Einsatzbereite Modelle für fluoreszierende Zellkerne, H&E-gefärbte Histologie und andere gängige Bildgebungsszenarien.
Klassifizieren Sie erkannte Objekte in verschiedene Klassen (z. B. unterschiedliche Zelltypen) in einem einzigen Segmentierungslauf.
Nahtlose Integration mit ImageJ/Fiji, QuPath und napari für zugängliche GUI-basierte Workflows.
Umfassende Evaluierung der Instanzsegmentierung inklusive Präzision, Recall, F1-Score und panoptischer Qualität.
Technischer Hintergrund
Ursprünglich in einem MICCAI-2018-Paper vorgestellt, besteht die Kerninnovation von StarDist in der Vorhersage radialer Abstände entlang fester Strahlen kombiniert mit Objektwahrscheinlichkeit für jeden Pixel, was eine genaue Rekonstruktion sternkonvexer Formen ermöglicht. Dieser Ansatz segmentiert zuverlässig eng aneinander liegende Objekte, die mit traditionellen pixelbasierten oder Bounding-Box-Methoden schwer zu trennen sind.
Neuere Entwicklungen haben StarDist auf Histopathologiebilder ausgeweitet, wodurch nicht nur die Kernsegmentierung, sondern auch die Mehrklassenklassifikation erkannter Objekte möglich ist. Die Methode erzielte Spitzenleistungen bei Herausforderungen wie der CoNIC (Colon Nuclei Identification and Counting) Challenge.
Download oder Zugriff
Installation & Einrichtung
Installieren Sie TensorFlow (Version 1.x oder 2.x) als Voraussetzung für StarDist.
Verwenden Sie pip, um das StarDist Python-Paket zu installieren:
pip install stardistFür napari:
pip install stardist-napariFür QuPath: Installieren Sie die StarDist-Erweiterung, indem Sie die .jar-Datei in QuPath ziehen.
Für ImageJ/Fiji: Verwenden Sie den integrierten Plugin-Manager oder die manuelle Installation über das Plugins-Menü.
Segmentierung ausführen
Laden Sie ein vortrainiertes Modell, normalisieren Sie Ihr Bild und führen Sie die Vorhersage aus:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Öffnen Sie Ihr Bild in napari, wählen Sie das StarDist-Plugin, wählen Sie ein vortrainiertes oder eigenes Modell und führen Sie die Vorhersage direkt über die GUI aus.
Verwenden Sie das StarDist-Plugin im Plugins-Menü, um ein Modell auf Ihren Bildstapel mit einer intuitiven Oberfläche anzuwenden.
Nach der Installation der Erweiterung führen Sie die StarDist-Erkennung über die Skriptkonsole oder grafische Oberfläche von QuPath für die Histopathologieanalyse aus.
Training & Feinabstimmung
Erstellen Sie Ground-Truth-Labelbilder, in denen jedes Objekt eindeutig beschriftet ist. Verwenden Sie Annotationswerkzeuge wie LabKit, QuPath oder Fiji zur Vorbereitung Ihres Datensatzes.
Nutzen Sie die StarDist Python-API, um ein neues Modell zu trainieren oder ein bestehendes mit Ihren eigenen annotierten Daten fein abzustimmen.
Nachbearbeitungsoptionen
- Wenden Sie Non-Maximum Suppression (NMS) an, um redundante Kandidatenformen zu eliminieren
- Verwenden Sie StarDist OPP (Object Post-Processing), um Masken für nicht sternkonvexe Formen zusammenzuführen
Einschränkungen & Überlegungen
- Die Annahme der Sternkonvexität modelliert sehr nicht-konvexe oder stark unregelmäßige Formen möglicherweise nicht perfekt
- Installationskomplexität: Benutzerdefinierte Installationen benötigen einen kompatiblen C++-Compiler zum Erstellen von Erweiterungen
- GPU-Beschleunigung hängt von kompatiblen Versionen von TensorFlow, CUDA und cuDNN ab
- Einige Nutzer berichten von Problemen bei der Ausführung des ImageJ-Plugins aufgrund von Java-Konfigurationen
Häufig gestellte Fragen
StarDist funktioniert mit verschiedenen Bildtypen, darunter Fluoreszenz, Hellfeld und Histopathologie (z. B. H&E), dank seiner flexiblen vortrainierten Modelle und Anpassungsfähigkeit an unterschiedliche Bildgebungsmodalitäten.
Ja — StarDist unterstützt 3D-Instanzsegmentierung mit sternkonvexen Polyedern für volumetrische Daten und erweitert die 2D-Fähigkeiten auf vollständige 3D-Analysen.
Nicht unbedingt. Vortrainierte Modelle sind verfügbar und funktionieren oft direkt einsatzbereit gut. Für spezialisierte oder neue Daten verbessert das Annotieren und Trainieren eigener Modelle jedoch die Genauigkeit deutlich.
StarDist integriert sich in napari, ImageJ/Fiji und QuPath, sodass Sie Segmentierungen über eine GUI ohne Programmierung durchführen können. Außerdem unterstützt es die direkte Nutzung der Python-API für fortgeschrittene Workflows.
StarDist bietet eingebaute Funktionen zur Berechnung gängiger Instanzsegmentierungsmetriken wie Präzision, Recall, F1-Score und panoptische Qualität zur Bewertung der Segmentierungsleistung.
SAM
Anwendungsinformationen
| Entwickler | Meta AI Research (FAIR) |
| Unterstützte Geräte |
|
| Sprache & Verfügbarkeit | Open-Source-Grundlagenmodell weltweit verfügbar; Dokumentation in Englisch |
| Preisgestaltung | Kostenlos — Open Source unter Meta-Lizenz via GitHub und MIB-Integration |
Allgemeiner Überblick
SAM (Segment Anything Model) ist ein leistungsstarkes KI-Grundlagenmodell von Meta, das interaktive und automatische Segmentierung nahezu beliebiger Objekte in Bildern ermöglicht. Mithilfe von Eingaben wie Punkten, Begrenzungsrahmen oder groben Masken erzeugt SAM Segmentierungsmasken, ohne dass ein aufgabenspezifisches Nachtraining erforderlich ist. In der Mikroskopieforschung wurde SAMs Flexibilität für Zellsegmentierung, Organellenerkennung und histopathologische Analyse angepasst und bietet eine skalierbare Lösung für Forscher, die ein promptfähiges, universelles Segmentierungswerkzeug benötigen.
Ausführliche Einführung
Ursprünglich von Meta auf über 1 Milliarde Masken über 11 Millionen Bilder trainiert, wurde SAM als promptfähiges Grundlagenmodell für Segmentierung mit „Zero-Shot“-Leistung auf neuen Domänen entwickelt. In der medizinischen Bildgebung wurde SAM für die Segmentierung ganzer Pathologie-Schnitte, Tumorerkennung und Zellkernidentifikation evaluiert. Die Leistung bei dicht gepackten Instanzen – wie Zellkernen – ist jedoch gemischt: Selbst mit umfangreichen Eingaben (z. B. 20 Klicks oder Rahmen) kann die Zero-Shot-Segmentierung bei komplexen Mikroskopiebildern Schwierigkeiten haben.
Zur Überwindung dieser Einschränkung entstanden domänenspezifische Anpassungen:
- SAMCell — Feinabgestimmt auf großen Mikroskopiedatensätzen für starke Zero-Shot-Segmentierung verschiedener Zelltypen ohne experimentenspezifisches Nachtraining
- μSAM — Nachtrainiert auf über 17.000 manuell annotierten Mikroskopiebildern zur Verbesserung der Genauigkeit bei kleinen zellulären Strukturen
Hauptmerkmale
Flexible Interaktion mit Punkten, Rahmen und Masken für präzise Steuerung.
Segmentierung ohne Feinabstimmung auf neuen Bilddomänen.
Anpassbar für Mikroskopie und Histopathologie durch Few-Shot- oder promptbasiertes Nachtraining.
Verfügbar im Microscopy Image Browser (MIB) mit Unterstützung für 3D- und interpolierte Segmentierung.
IDCC-SAM ermöglicht automatische Zellzählung in der Immunzytochemie ohne manuelle Annotation.
Download oder Zugriff
Benutzerhandbuch
- Öffnen Sie den Microscopy Image Browser und navigieren Sie zum SAM-Segmentierungsfenster
- Konfigurieren Sie den Python-Interpreter und wählen Sie zwischen den Modellen SAM-1 oder SAM-2
- Für GPU-Beschleunigung wählen Sie „cuda“ in der Ausführungsumgebung (empfohlen für optimale Leistung)
- Punkt-Eingaben: Klicken Sie auf ein Objekt, um einen positiven Startpunkt zu definieren; verwenden Sie Shift + Klick zum Erweitern und Strg + Klick für negative Startpunkte
- 3D-Stapel: Nutzen Sie den interaktiven 3D-Modus – klicken Sie auf eine Schicht, scrollen Sie mit Shift und interpolieren Sie Startpunkte über Schichten hinweg
- Modus anpassen: Masken ersetzen, hinzufügen, subtrahieren oder bei Bedarf eine neue Ebene erstellen
- Verwenden Sie die Option „Automatisch alles“ im SAM-2-Panel von MIB, um alle sichtbaren Objekte in einem Bereich zu segmentieren
- Überprüfen und verfeinern Sie Masken nach der Segmentierung bei Bedarf
- Nutzen Sie promptbasierte Feinabstimmungs-Pipelines (z. B. „All-in-SAM“), um Pixel-Annotationen aus spärlichen Benutzereingaben zu generieren
- Für Zellzählung wenden Sie IDCC-SAM an, das SAM in einer Zero-Shot-Pipeline mit Nachbearbeitung verwendet
- Für hochpräzise Zellsegmentierung verwenden Sie SAMCell, feinabgestimmt auf Mikroskopiezellbilder
Einschränkungen & Überlegungen
- Zero-Shot-Leistung inkonsistent bei dichten oder überlappenden Strukturen ohne Domänenanpassung
- Segmentierungsqualität stark abhängig von Eingabegestaltung und Strategie
- GPU wird dringend empfohlen; CPU-Inferenz ist sehr langsam
- Probleme bei sehr hochauflösenden Ganzschnittbildern und multiskaligen Gewebestrukturen
- Feinabstimmung oder Anpassung von SAM für Mikroskopie erfordert möglicherweise Kenntnisse im maschinellen Lernen
Häufig gestellte Fragen
Ja – durch Anpassungen wie SAMCell, das SAM speziell für Zellsegmentierungsaufgaben auf Mikroskopiedatensätzen feinabstimmt.
Nicht immer. Mit IDCC-SAM können Sie Zero-Shot-Zellzählung ohne manuelle Annotation durchführen.
Verwenden Sie promptbasierte Feinabstimmung (z. B. „All-in-SAM“) oder vortrainierte Mikroskopie-Versionen wie μSAM, das auf über 17.000 annotierten Mikroskopiebildern trainiert wurde.
Obwohl eine Ausführung auf der CPU möglich ist, wird eine GPU dringend empfohlen für praktische Inferenzgeschwindigkeit und Echtzeit-interaktive Segmentierung.
Ja – die SAM-2-Integration in MIB unterstützt 3D-Segmentierung mit Seed-Interpolation über Schichten hinweg für volumetrische Analysen.
AxonDeepSeg
| Entwickler | NeuroPoly Lab an der Polytechnique Montréal und der Université de Montréal |
| Unterstützte Plattformen |
|
| Sprache | Englische Dokumentation; Open-Source-Tool mit weltweiter Nutzung |
| Preisgestaltung | Kostenlos und Open-Source |
Überblick
AxonDeepSeg ist ein KI-gestütztes Werkzeug zur automatischen Segmentierung von Axonen und Myelin in Mikroskopiebildern. Mithilfe konvolutionaler neuronaler Netze liefert es eine präzise Segmentierung in drei Klassen (Axon, Myelin, Hintergrund) über mehrere Bildgebungsmodalitäten hinweg, darunter TEM, SEM und Hellfeldmikroskopie. Durch die Automatisierung morphometrischer Messungen wie Axondurchmesser, g-Ratio und Myelindicke vereinfacht AxonDeepSeg die quantitative Analyse in der neurowissenschaftlichen Forschung, reduziert die manuelle Annotationszeit erheblich und verbessert die Reproduzierbarkeit.
Hauptmerkmale
Einsatzbereite Modelle, optimiert für TEM, SEM und Hellfeldmikroskopie.
Präzise Klassifizierung von Axon-, Myelin- und Hintergrundbereichen in Mikroskopiebildern.
Automatische Berechnung von Axondurchmesser, g-Ratio, Myelindicke und Dichte-Metriken.
Napari-GUI-Integration ermöglicht manuelle Verfeinerung der Segmentierungsmasken für höhere Genauigkeit.
Nahtlose Integration in individuelle Pipelines für groß angelegte Analysen neuronalen Gewebes.
Umfassende Testscripte gewährleisten Reproduzierbarkeit und zuverlässige Segmentierungsergebnisse.
Technische Details
Entwickelt vom NeuroPoly Lab nutzt AxonDeepSeg Deep Learning, um hochpräzise Segmentierungen für neurowissenschaftliche Anwendungen zu liefern. Vortrainierte Modelle stehen für verschiedene Mikroskopiemodalitäten zur Verfügung und gewährleisten Vielseitigkeit über Bildgebungstechniken hinweg. Das Tool integriert sich mit Napari, was interaktive Korrekturen der Segmentierungsmasken erlaubt und die Genauigkeit bei herausfordernden Datensätzen verbessert. AxonDeepSeg berechnet wichtige morphometrische Metriken und unterstützt Hochdurchsatzstudien zur Struktur und Pathologie neuronalen Gewebes. Das Python-basierte Framework ermöglicht die Integration in individuelle Pipelines für groß angelegte Analysen von Axon- und Myelinmorphologie.
Download oder Zugriff
Installation & Einrichtung
Stellen Sie sicher, dass Python 3.8 oder höher installiert ist, und installieren Sie dann AxonDeepSeg und Napari mit pip:
pip install axondeepseg napariFühren Sie die bereitgestellten Testscripte aus, um zu bestätigen, dass alle Komponenten korrekt installiert sind und funktionieren.
Importieren Sie Mikroskopiebilder (TEM, SEM oder Hellfeld) in Napari oder Ihre Python-Umgebung.
Wählen Sie das passende vortrainierte Modell für Ihre Bildmodalität und führen Sie die Segmentierung aus, um Axon- und Myelinmasken zu erzeugen.
Berechnen Sie automatisch morphometrische Messwerte wie Axondurchmesser, g-Ratio, Dichte und Myelindicke und exportieren Sie die Ergebnisse im CSV-Format.
Nutzen Sie die Napari-GUI, um Segmentierungsmasken manuell anzupassen, indem Sie Masken zusammenführen oder löschen, um die Genauigkeit zu verbessern.
Wichtige Hinweise
- Die Leistung kann bei neuen oder untrainierten Bildgebungsmodalitäten abnehmen
- Manuelle Korrekturen können bei schwierigen oder komplexen Bereichen erforderlich sein
- GPU wird für schnellere Verarbeitung großer Datensätze empfohlen; CPU-Verarbeitung wird ebenfalls unterstützt
Häufig gestellte Fragen
AxonDeepSeg unterstützt TEM (Transmissionselektronenmikroskopie), SEM (Rasterelektronenmikroskopie) und Hellfeldmikroskopie mit vortrainierten Modellen, die für jede Modalität optimiert sind.
Ja, AxonDeepSeg ist vollständig kostenlos und Open-Source, verfügbar für akademische und kommerzielle Nutzung.
Ja, AxonDeepSeg berechnet automatisch Axondurchmesser, g-Ratio, Myelindicke und Dichte-Metriken aus segmentierten Bildern.
Eine GPU wird für schnellere Segmentierung großer Datensätze empfohlen, aber die Verarbeitung auf der CPU wird für kleinere Analysen ebenfalls unterstützt.
Ja, die Integration der Napari-GUI ermöglicht interaktive Korrekturen und Verfeinerungen der Segmentierungsmasken für höhere Genauigkeit bei schwierigen Bereichen.
Ilastik
| Entwickler | Ilastik-Team am Europäischen Laboratorium für Molekularbiologie (EMBL) und assoziierte akademische Partner |
| Unterstützte Plattformen |
|
| Sprache | Englisch |
| Preisgestaltung | Kostenlos und Open-Source |
Überblick
Ilastik ist ein leistungsstarkes, KI-gesteuertes Werkzeug für interaktive Bildsegmentierung, Klassifikation und Analyse von Mikroskopiedaten. Mithilfe von maschinellen Lernverfahren wie Random-Forest-Klassifikatoren ermöglicht es Forschern, Pixel zu segmentieren, Objekte zu klassifizieren, Zellen über die Zeit zu verfolgen und Dichtezählungen in 2D- und 3D-Datensätzen durchzuführen. Mit seiner intuitiven Benutzeroberfläche und dem Echtzeit-Feedback ist Ilastik für Wissenschaftler ohne Programmierkenntnisse zugänglich und wird in Zellbiologie, Neurowissenschaften und biomedizinischer Bildgebung weit verbreitet eingesetzt.
Hauptfunktionen
Echtzeit-Feedback, während Sie repräsentative Regionen annotieren, für sofortige Segmentierungsergebnisse.
Kategorisieren Sie segmentierte Strukturen basierend auf morphologischen und Intensitätsmerkmalen.
Verfolgen Sie Zellbewegungen und Zellteilungen in 2D- und 3D-Zeitreihen-Mikroskopieexperimenten.
Quantifizieren Sie überfüllte Bereiche ohne explizite Segmentierung einzelner Objekte.
Semi-automatische Segmentierung komplexer 3D-Volumen mit intuitiver Interaktion.
Verarbeiten Sie mehrere Bilder automatisch im headless Kommandozeilenmodus.
Download
Erste Schritte Anleitung
Laden Sie Ilastik für Ihr Betriebssystem von der offiziellen Webseite herunter. Das Paket enthält alle notwendigen Python-Abhängigkeiten, folgen Sie den Installationsanweisungen für Ihre Plattform.
Öffnen Sie Ilastik und wählen Sie Ihren Analyse-Workflow: Pixelklassifikation, Objektklassifikation, Tracking oder Dichtezählung. Laden Sie Ihren Bilddatensatz, der Mehrkanal-, 3D- oder Zeitreihenbilder enthalten kann.
Markieren Sie einige repräsentative Pixel oder Objekte in Ihren Bildern. Der Random-Forest-Klassifikator von Ilastik lernt aus diesen Annotationen und sagt automatisch Labels für den gesamten Datensatz voraus.
Wenden Sie das trainierte Modell an, um Ihren gesamten Datensatz zu segmentieren oder zu klassifizieren. Exportieren Sie Ergebnisse als beschriftete Bilder, Wahrscheinlichkeitskarten oder quantitative Tabellen für die Weiterverarbeitung und Visualisierung.
Nutzen Sie den headless Modus von Ilastik, um mehrere Bilder automatisch ohne manuelle Eingriffe zu verarbeiten – ideal für groß angelegte Analyse-Pipelines.
Einschränkungen & Überlegungen
- Interaktive Beschriftung kann bei sehr großen Datensätzen zeitaufwändig sein
- Genauigkeit hängt ab von der Qualität und Repräsentativität der Nutzerannotationen
- Speicherbedarf — sehr hochauflösende oder multi-Gigabyte-Datensätze können erheblichen RAM erfordern
- Komplexe Daten — Random-Forest-Klassifikatoren können bei stark variierenden oder komplexen Bilddaten gegenüber tiefen neuronalen Netzen unterlegen sein
Häufig gestellte Fragen
Ja, Ilastik unterstützt vollständig 3D-Volumen und Zeitreihenexperimente für Segmentierung, Tracking und quantitative Analyse über mehrere Zeitpunkte.
Ja, Ilastik ist komplett kostenlos und Open-Source, für alle Nutzer ohne Lizenzbeschränkungen verfügbar.
Nein, Ilastik bietet eine intuitive grafische Benutzeroberfläche mit Echtzeit-Feedback, die es Forschern ohne Programmierkenntnisse zugänglich macht. Fortgeschrittene Nutzer können auch die Kommandozeilen-Stapelverarbeitung nutzen.
Ja, der dedizierte Tracking-Workflow ermöglicht die Analyse von Zellbewegungen und Zellteilungen in 2D- und 3D-Zeitreihendatensätzen mit automatischer Linienverfolgung.
Segmentierungsergebnisse können als beschriftete Bilder, Wahrscheinlichkeitskarten oder quantitative Tabellen exportiert werden, was eine nahtlose Integration mit nachgelagerten Analysewerkzeugen und Visualisierungssoftware ermöglicht.
Diese Werkzeuge decken Anfänger- bis Expertenlevel ab. Viele sind kostenlos und Open Source, was reproduzierbare und teilbare KI-Workflows in der Forschungsgemeinschaft fördert.
Herausforderungen und Zukunftsperspektiven
Aktuelle Herausforderungen
Aufkommende Trends
Vision-Foundation-Modelle
KI-Systeme der nächsten Generation versprechen, den Bedarf an aufgabenspezifischem Training zu reduzieren.
- Modelle wie SAM und CLIP-basierte Systeme
- Eine KI für viele Mikroskopie-Aufgaben
- Schnellere Implementierung und Anpassung
KI-unterstützte Mikroskope
Vollautonome und intelligente Mikroskopiesysteme werden Realität.
- Natürliche Sprachsteuerung via LLMs
- Vollautomatische Feedback-Schleifen
- Demokratisierung des Zugangs zu fortschrittlicher Mikroskopie
Wichtige Erkenntnisse
- KI transformiert die Mikroskop-Bildverarbeitung schnell mit verbesserter Genauigkeit und Automatisierung
- Deep Learning übertrifft traditionelles Maschinelles Lernen bei komplexen, variablen Mikroskopiebildern
- CNNs lernen automatisch hierarchische Merkmale aus Rohpixeln für robuste Analysen
- Wichtige Anwendungen sind Segmentierung, Klassifikation, Tracking, Rauschunterdrückung und automatisierte Aufnahme
- Erfolg hängt von hochwertigen Daten und sorgfältiger Validierung durch Experten ab
- Vision-Foundation-Modelle und KI-unterstützte Mikroskope repräsentieren die Zukunft des Feldes
Mit fortschreitenden Entwicklungen und gemeinschaftlichen Anstrengungen (Open-Source-Tools, geteilte Datensätze) wird KI zunehmend zum Kern des „Auges“ des Mikroskops und hilft Wissenschaftlern, das Unsichtbare sichtbar zu machen.







No comments yet. Be the first to comment!