顯微鏡影像處理中的人工智能
人工智能正以精確分割、降噪、超高解析度及自動影像擷取等強大功能,革新顯微鏡影像處理。本文重點介紹科學研究中重要的人工智能工具及新興趨勢。
人工智能技術正透過優化影像擷取及自動化分析,革新顯微鏡領域。在現代智慧顯微鏡中,人工智能模組可即時調整成像參數(如焦距、照明),以減少光漂白並提升訊號。同時,深度學習演算法能從複雜的影像資料中篩選出隱藏的生物學見解,甚至將影像與其他資料(如基因組學)連結。
人工智能方法:機器學習與深度學習
人工智能方法涵蓋經典的機器學習(ML)及現代的深度學習(DL),各有其優勢與限制:
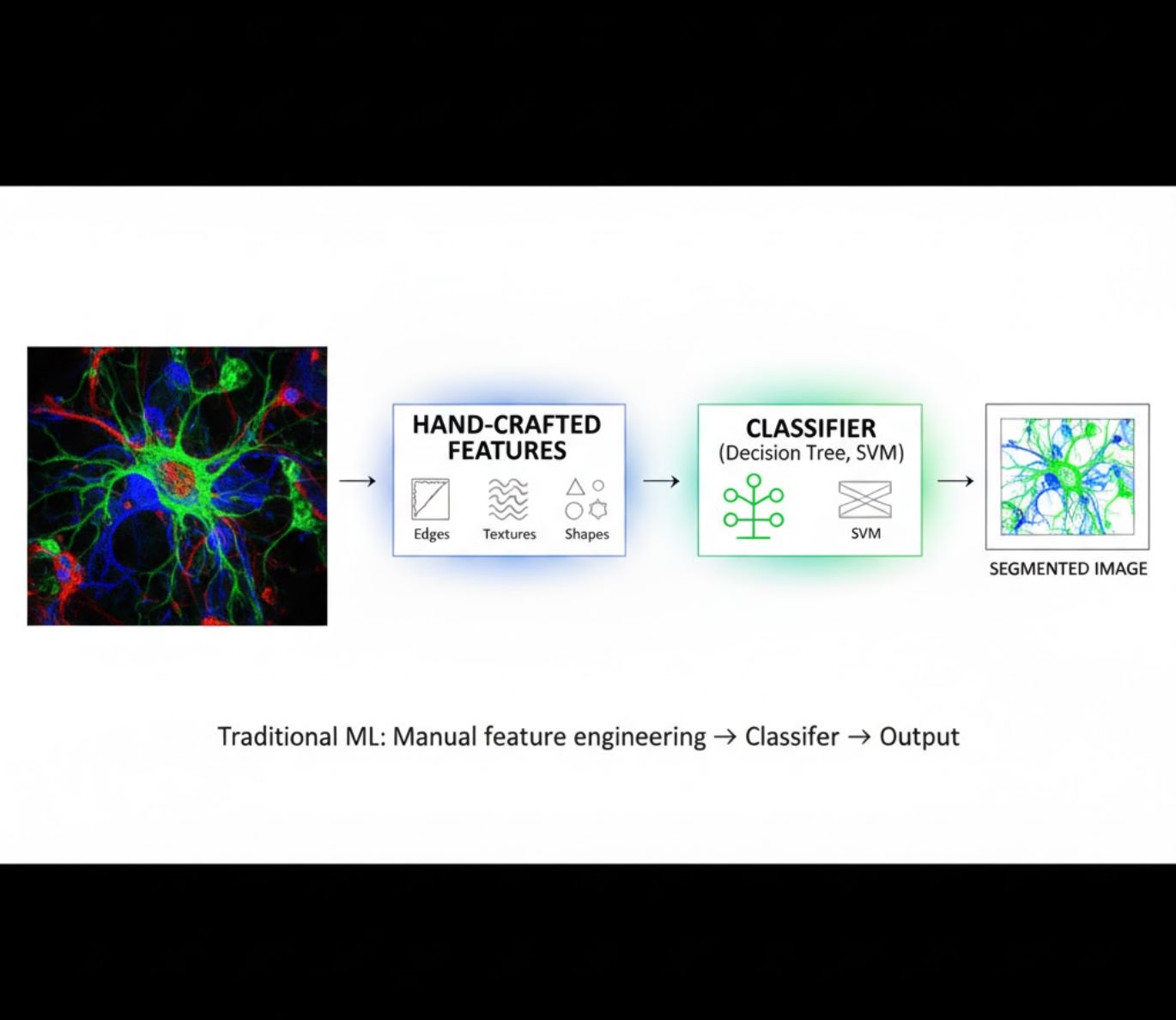
手工特徵設計
- 研究人員手動設計影像特徵(邊緣、紋理、形狀)
- 將特徵輸入分類器(決策樹、支持向量機)
- 訓練速度快
- 對複雜或噪聲影像表現較差
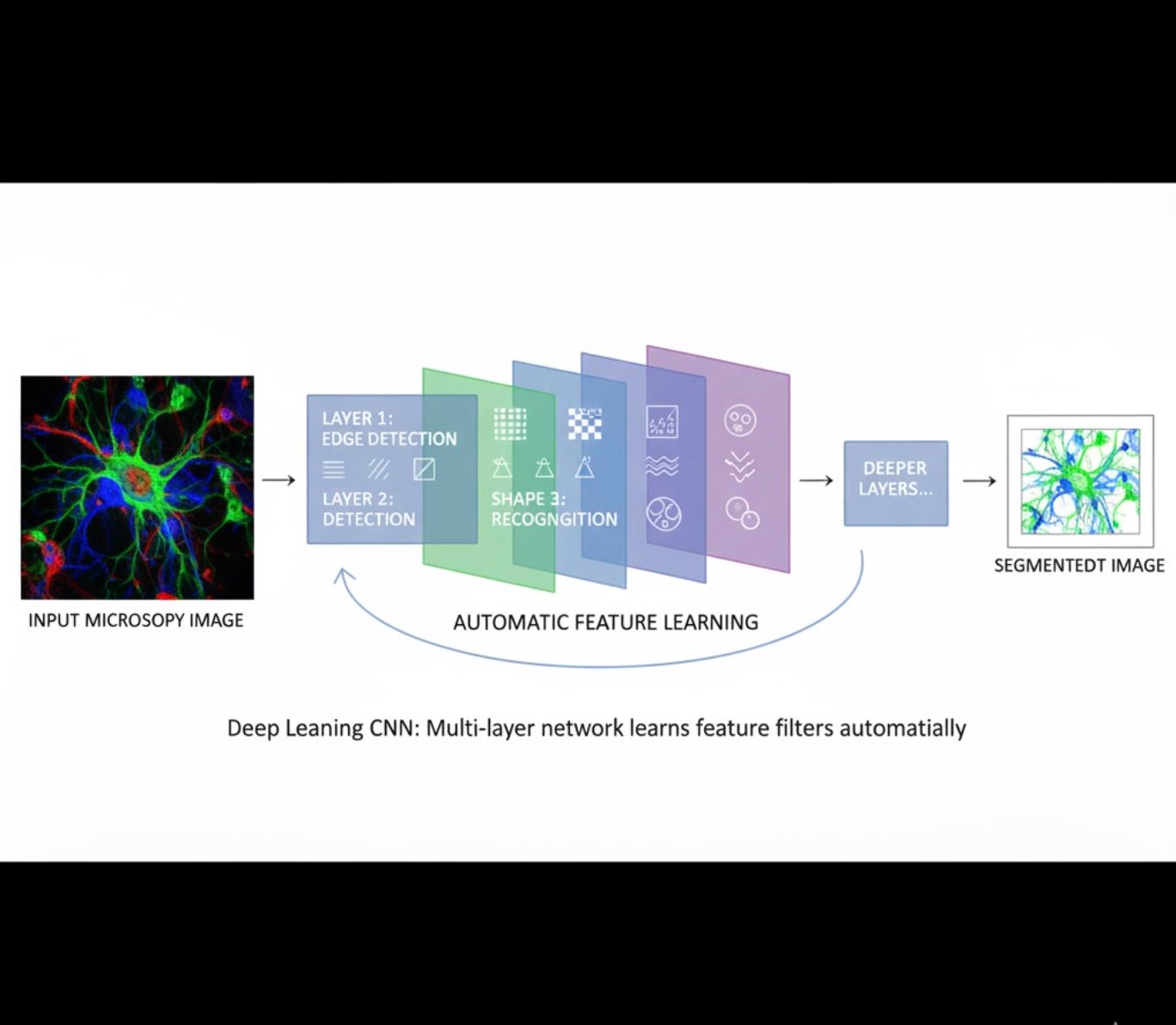
自動特徵學習
- 多層神經網絡(CNN)自動學習特徵
- 從原始像素端到端學習
- 對變異更具魯棒性
- 可靠捕捉複雜紋理與結構
CNN運作原理:卷積神經網絡對顯微鏡影像施加連續濾波器,早期層學習偵測簡單模式(邊緣),深層則捕捉複雜結構(細胞形狀、紋理)。這種分層學習使深度學習在強度變化大時仍具高度魯棒性。
視覺比較:機器學習與深度學習流程
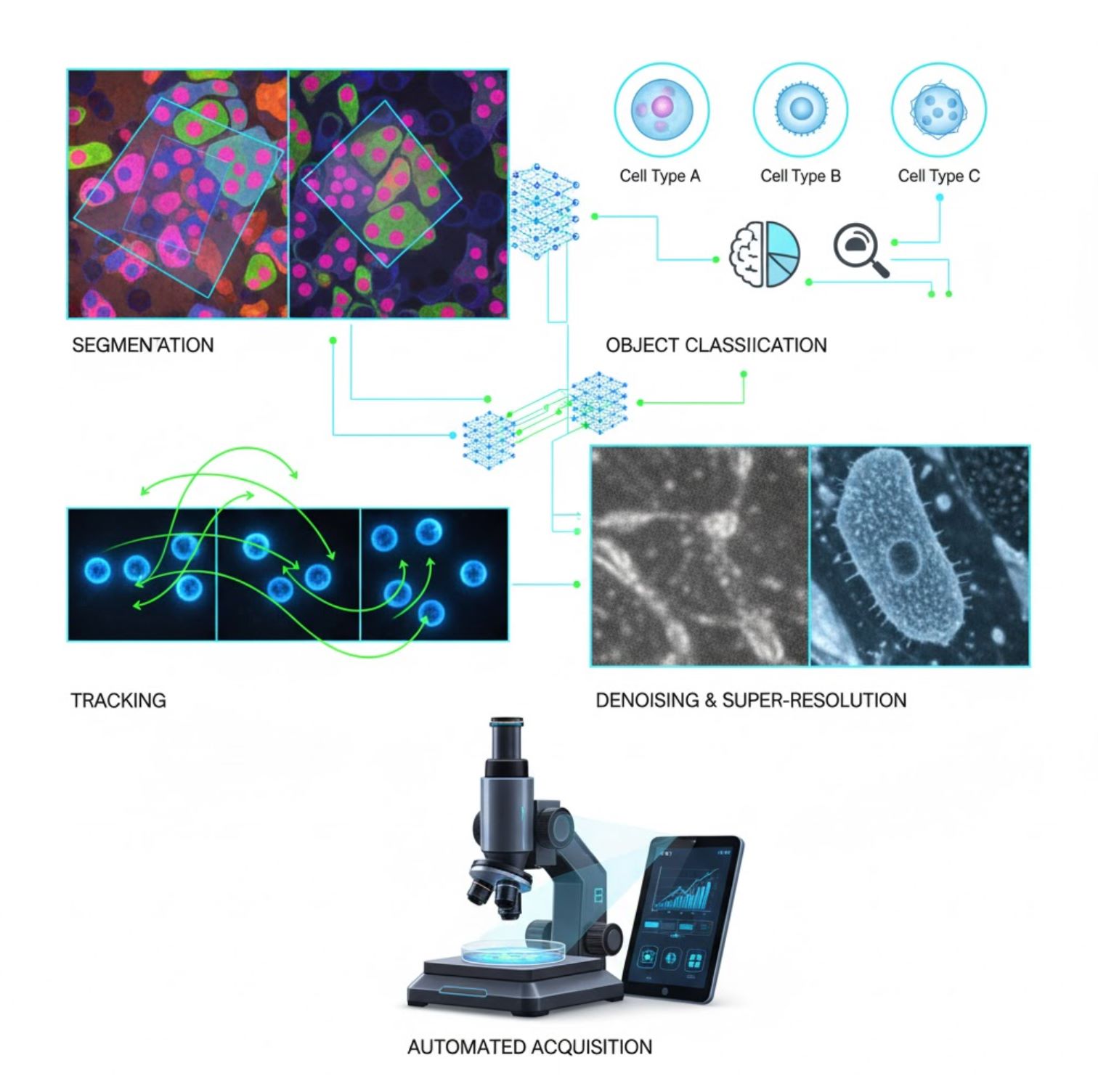
顯微鏡中人工智能的主要應用
人工智能現已嵌入顯微鏡影像處理的多個任務:
分割
將影像劃分為不同區域(如識別每個細胞或細胞核)。深度網絡如U-Net在此任務表現優異。
- 語義分割:每像素分類標籤
- 實例分割:區分個別物體
- 在擁擠或暗淡影像中仍具高準確度
- 視覺基礎模型(如μSAM)現已適用於顯微鏡
物體分類
分割後,人工智能可高精度分類每個物體。
- 細胞類型識別
- 有絲分裂階段判定
- 病理指標檢測
- 辨識難以手動量化的細微表型
追蹤
在時間序列顯微鏡中,人工智能能以前所未有的準確度追蹤細胞或粒子。
- 深度學習大幅提升追蹤準確性
- 實現對移動細胞的可靠分析
- 捕捉動態生物過程
降噪與超高解析度
人工智能模型透過去除噪聲與模糊,提升影像品質。
- 物理知識導向的深度模型學習顯微鏡光學
- 重建更銳利、無偽影的影像
- 相較傳統方法,解析度更高且偽影更少
自動擷取
人工智能實時引導顯微鏡操作。
- 分析即時影像做出智能決策
- 自動調整焦距並掃描感興趣區域
- 減少光毒性並節省時間
- 實現高通量及自適應成像實驗
顯微鏡影像處理中熱門的人工智能工具
<div i='1'>一個豐富的工具生態系統支持顯微鏡中的人工智能。研究人員已開發出多種通用及專用軟件,其中許多是開源的:</div>
Cellpose
| 開發者 | Carsen Stringer 與 Marius Pachitariu(MouseLand 研究團隊) |
| 支援平台 |
需安裝 Python(pip/conda)。圖形介面僅限桌面使用。 |
| 語言支援 | 英文文件;全球研究實驗室廣泛採用 |
| 收費模式 | 免費且開源,採 BSD-3-Clause 授權條款 |
概覽
Cellpose 是一款先進的深度學習分割工具,專為顯微鏡影像設計。作為通用演算法,它能準確分割多種細胞類型(細胞核、細胞質等)及不同成像模式,無需重新訓練。具備人機互動功能,研究人員可優化結果,調整模型以適應自訂數據,並應用於 2D 及 3D 影像流程。
主要功能
開箱即用,適用多種細胞類型、染色及成像模式,無需自訂訓練。
支持使用「2.5D」方法處理完整 3D 堆疊,重用 2D 模型進行體積資料分割。
手動修正分割結果,並在自訂數據上再訓練模型以提升準確度。
提供 Python API、命令列介面及圖形介面,靈活支援不同工作流程。
具備去噪、去模糊及升頻功能,提升分割前的影像品質。
下載或存取
技術背景
Cellpose 由 Stringer、Wang、Michaelos 與 Pachitariu 在一項開創性研究中提出,訓練於一個包含超過 70,000 個分割物件 的大型多樣化數據集。此多樣性使模型能跨越細胞形狀、大小及顯微鏡設定進行泛化,大幅減少多數應用場景下的自訂訓練需求。對於 3D 數據,Cellpose 巧妙地以「2.5D」方式重用 2D 模型,避免完全 3D 標註訓練數據的需求,同時提供體積分割。Cellpose 2.0 引入了 人機互動式再訓練,允許用戶手動修正預測並在自有影像上再訓練,以提升特定數據集的表現。
安裝與設定
使用 conda 建立 Python 環境:
conda create -n cellpose python=3.10啟動環境並安裝 Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellpose快速入門
圖形介面模式
- 執行以下指令啟動 GUI:
python -m cellpose - 將影像檔案(
.tif、.png等)拖放至介面 - 選擇模型類型(例如「cyto」代表細胞質,「nuclei」代表細胞核)
- 設定估計細胞直徑,或讓 Cellpose 自動校準
- 點擊開始分割並查看結果
Python API 模式
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)優化與再訓練
- 生成遮罩後,在 GUI 中手動合併或刪除遮罩以修正分割
- 使用內建訓練功能,基於修正範例進行再訓練
- 提升模型在特定數據集上的表現
處理 3D 數據
- 載入多層 Z 軸 TIFF 或體積堆疊
- 在 GUI 或 API 中使用
--Zstack參數以 3D 模式處理 - 可選擇透過平滑或專用參數優化 3D 流以提升分割效果
限制與注意事項
- 模型通用性折衷:通用模型適用範圍廣,但對極端異常細胞形狀或成像條件可能需重新訓練。
- 標註工作量:人機互動式訓練需手動修正,對大型數據集耗時較長。
- 安裝複雜度:GUI 安裝可能需使用命令列、conda 環境及管理 Python 依賴,對非程式使用者不一定直觀。
- 僅限桌面:Cellpose 設計為桌面使用,無原生 Android 或 iOS 應用程式。
常見問題
不需要 — Cellpose 提供預訓練的通用模型,通常無需再訓練即可良好運作。但若需在特殊或異常數據上達到最佳效果,可利用人機互動功能進行標註與再訓練。
能 — 它透過重用 2D 模型的「2.5D」方法支持 3D,並可在 GUI 或 API 中處理體積堆疊。
建議使用 GPU 以加快推論與訓練速度,尤其是大型或 3D 數據集,但 Cellpose 亦可在僅 CPU 的機器上運行,性能較慢。
在 GUI 中可手動設定估計細胞直徑,或讓 Cellpose 自動校準。若分割效果不理想,可進一步優化結果並再訓練。
可以 — 新版本(Cellpose 3)包含影像修復模型,具備去噪、去模糊及升頻功能,提升分割前的影像品質。
StarDist
| 開發者 | Uwe Schmidt、Martin Weigert、Coleman Broaddus 及 Gene Myers |
| 支援平台 |
|
| 語言支援 | 開源專案,文件與社群主要以英文為主 |
| 收費模式 | 免費且開源。授權採用 BSD-3-Clause |
概覽
StarDist 是一款用於顯微鏡影像中實例分割的深度學習工具。它將每個物體(如細胞核)表示為 2D 的星形凸多邊形或 3D 的多面體,能準確偵測並分離密集或重疊的物體。憑藉其穩健架構,StarDist 廣泛應用於螢光顯微鏡、組織病理學及其他生物影像分析的自動細胞與細胞核分割。
主要特點
利用星形凸多邊形(2D)及多面體(3D)實現高度精確的實例分割,確保物體偵測可靠。
專為 2D 影像及 3D 體積資料設計的模型,提供全面的顯微鏡分析。
提供螢光細胞核、H&E 染色組織學及其他常見影像場景的即用型模型。
在單次分割中將偵測到的物體分類至不同類別(例如不同細胞類型)。
與 ImageJ/Fiji、QuPath 及 napari 無縫整合,提供易用的 GUI 工作流程。
包含精確度、召回率、F1 分數及全景品質等全面的實例分割評估指標。
技術背景
StarDist 最初於 2018 年 MICCAI 論文中提出,其核心創新在於沿固定射線預測放射距離並結合每像素的物體概率,從而準確重建星形凸形狀。此方法能可靠分割緊密接觸且難以用傳統像素或邊界框方法分離的物體。
近期發展擴展至組織病理學影像,不僅能分割細胞核,還能對偵測物體進行多類別分類。該方法在 CoNIC(結腸細胞核識別與計數)挑戰賽中取得頂尖表現。
下載或存取
安裝與設定
安裝 TensorFlow(版本 1.x 或 2.x)作為 StarDist 的前置條件。
使用 pip 安裝 StarDist Python 套件:
pip install stardistnapari:
pip install stardist-napariQuPath: 將 .jar 檔案拖曳至 QuPath 以安裝 StarDist 擴充功能。
ImageJ/Fiji: 使用內建插件管理器或透過插件選單手動安裝。
執行分割
載入預訓練模型,標準化影像,並執行預測:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)在 napari 中開啟影像,選擇 StarDist 插件,挑選預訓練或自訂模型,並直接從 GUI 執行預測。
從插件選單使用 StarDist 插件,透過直覺介面對影像堆疊套用模型。
安裝擴充功能後,可透過 QuPath 的腳本控制台或圖形介面執行 StarDist 偵測,適用於組織病理分析。
訓練與微調
建立真實標註標籤影像,確保每個物體有唯一標記。可使用 LabKit、QuPath 或 Fiji 等標註工具準備資料集。
使用 StarDist Python API 以自訂標註資料訓練新模型或微調現有模型。
後處理選項
- 應用非極大值抑制(NMS)以消除冗餘候選形狀
- 使用 StarDist OPP(物體後處理)合併非星形凸形狀的遮罩
限制與注意事項
- 星形凸假設可能無法完美模擬高度非凸或極不規則形狀
- 安裝複雜度:自訂安裝需相容的 C++ 編譯器以編譯擴充功能
- GPU 加速依賴相容的 TensorFlow、CUDA 及 cuDNN 版本
- 部分用戶反映因 Java 設定問題導致 ImageJ 插件運行異常
常見問題
StarDist 支援多種影像類型,包括螢光、明場及組織病理學(如 H&E),得益於其靈活的預訓練模型及對不同影像模式的適應性。
可以 — StarDist 支援使用星形凸多面體進行 3D 實例分割,將 2D 功能擴展至完整 3D 分析。
不一定。已有預訓練模型且通常可直接使用。但對於專門或新穎資料,自行標註並訓練自訂模型能顯著提升準確度。
StarDist 可整合於 napari、ImageJ/Fiji 及 QuPath,讓您無需編碼即可透過 GUI 執行分割。也支援直接使用 Python API 以進行進階工作流程。
StarDist 內建計算常用實例分割指標的功能,包括精確度、召回率、F1 分數及全景品質,以評估分割效能。
SAM
應用資訊
| 開發者 | Meta AI 研究院(FAIR) |
| 支援裝置 |
|
| 語言及可用性 | 全球開源基礎模型;文件以英文提供 |
| 價格 | 免費 — 透過 Meta 授權於 GitHub 及 MIB 整合提供開源 |
概覽
SAM(Segment Anything Model)是由Meta打造的強大AI基礎模型,能夠互動式及自動分割幾乎任何圖像中的物體。透過點、邊界框或粗略遮罩等提示,SAM可生成分割遮罩,無需特定任務的重新訓練。在顯微鏡研究中,SAM的靈活性被用於細胞分割、細胞器偵測及組織病理分析,為需要可提示、通用分割工具的研究人員提供可擴展的解決方案。
詳細介紹
SAM最初由Meta在超過10億個遮罩及1100萬張圖像上訓練,設計為可提示的基礎分割模型,具備對新領域的「零樣本」性能。在醫學影像研究中,SAM已被評估用於全片病理分割、腫瘤偵測及細胞核識別。然而,對於密集排列的實例(如細胞核),其表現參差不齊:即使使用大量提示(例如20次點擊或框選),零樣本分割在複雜顯微鏡圖像中仍可能遇到困難。
為解決此限制,出現了領域特定的調整版本:
- SAMCell — 在大型顯微鏡數據集上微調,實現跨多種細胞類型的強大零樣本分割,無需每次實驗重新訓練
- μSAM — 在超過17,000張手動標註的顯微鏡圖像上重新訓練,以提升對小型細胞結構的準確度
主要特點
使用點、框及遮罩靈活互動,精確控制分割結果。
無需在新影像領域微調即可執行分割。
可透過少量樣本或提示微調,適用於顯微鏡及組織病理影像。
於 Microscopy Image Browser(MIB)中提供,支援3D及插值分割。
IDCC-SAM實現免疫細胞化學中無需手動標註的自動細胞計數。
下載或存取
使用指南
- 開啟 Microscopy Image Browser,前往 SAM 分割面板
- 設定 Python 解譯器,並選擇 SAM-1 或 SAM-2 模型
- 若需 GPU 加速,請在執行環境中選擇「cuda」(建議以獲得最佳效能)
- 點提示:點擊物體以定義正向種子;使用 Shift + 點擊 擴展,Ctrl + 點擊 設定負向種子
- 3D 堆疊:使用互動式3D模式—點擊單張切片,Shift滾動滑鼠,並在切片間插值種子
- 調整模式:根據需要替換、添加、減去遮罩,或建立新圖層
- 在 SAM-2 面板中使用 MIB 的「自動全部」選項,分割區域內所有可見物體
- 分割後檢視並必要時微調遮罩
- 使用基於提示的微調流程(如「All-in-SAM」)從稀疏用戶提示生成像素級標註
- 細胞計數可應用 IDCC-SAM,利用 SAM 進行零樣本流程並搭配後處理
- 高精度細胞分割則使用在顯微鏡細胞影像上微調的 SAMCell
限制與注意事項
- 未經領域調整,零樣本性能在密集或重疊結構上不穩定
- 分割品質嚴重依賴提示設計與策略
- 強烈建議使用GPU;CPU推理速度非常慢
- 對超高解析度全片影像及多尺度組織結構處理存在困難
- 微調或調整SAM於顯微鏡影像可能需要機器學習專業知識
常見問題
可以—透過如SAMCell等調整版本,專門在顯微鏡數據集上微調以執行細胞分割任務。
不一定。使用IDCC-SAM,可在無需手動標註的情況下進行零樣本細胞計數。
可使用基於提示的微調(如「All-in-SAM」)或採用預訓練的顯微鏡版本,如在超過17,000張標註顯微鏡圖像上訓練的μSAM。
雖然CPU可用,但強烈建議使用GPU以獲得實用的推理速度及即時互動分割體驗。
可以—MIB的SAM-2整合支援3D分割,並可在切片間插值種子以進行體積分析。
AxonDeepSeg
| 開發者 | 蒙特利爾理工學院及蒙特利爾大學 NeuroPoly 實驗室 |
| 支援平台 |
|
| 語言 | 英文文件;全球開源工具 |
| 價格 | 免費且開源 |
概覽
AxonDeepSeg 是一款 AI 驅動的工具,用於自動分割顯微鏡圖像中的軸突與髓鞘。透過卷積神經網絡,提供準確的三類分割(軸突、髓鞘、背景),支援多種成像模式,包括 TEM、SEM 及明場顯微鏡。自動化形態計量測量如軸突直徑、g 比率及髓鞘厚度,助力神經科學研究中的定量分析,大幅減少手動標註時間並提升重現性。
主要功能
針對 TEM、SEM 及明場顯微鏡模式優化的即用型模型。
精確分類顯微鏡圖像中的軸突、髓鞘及背景區域。
自動計算軸突直徑、g 比率、髓鞘厚度及密度指標。
整合 Napari GUI,支援手動微調分割遮罩以提升準確度。
無縫整合至自訂流程,適用於大規模神經組織分析。
完整測試腳本確保重現性及分割結果可靠性。
技術細節
AxonDeepSeg 由 NeuroPoly 實驗室開發,利用深度學習技術提供高精度神經科學應用分割。針對不同顯微鏡模式提供預訓練模型,確保多種成像技術的適用性。工具整合 Napari,允許互動式修正分割遮罩,提升複雜數據集的準確度。AxonDeepSeg 可計算關鍵形態計量指標,支持神經組織結構及病理的高通量研究。其基於 Python 的框架便於整合至大規模軸突與髓鞘形態分析的自訂流程。
下載或存取
安裝與設定
確保已安裝 Python 3.8 或以上版本,然後使用 pip 安裝 AxonDeepSeg 與 Napari:
pip install axondeepseg napari執行提供的測試腳本,確認所有組件均已正確安裝並正常運作。
將顯微鏡圖像(TEM、SEM 或明場)匯入 Napari 或您的 Python 環境。
選擇適合您成像模式的預訓練模型,執行分割以產生軸突與髓鞘遮罩。
自動計算形態計量數據,包括軸突直徑、g 比率、密度及髓鞘厚度,並以 CSV 格式匯出結果。
使用 Napari GUI 手動調整分割遮罩,合併或刪除遮罩以提升準確度。
重要注意事項
- 對新穎或未訓練的成像模式,效能可能下降
- 複雜或具挑戰性的區域可能需手動修正
- 建議使用 GPU 加速大型數據集處理;亦支援 CPU 處理
常見問題
AxonDeepSeg 支援透射電子顯微鏡(TEM)、掃描電子顯微鏡(SEM)及明場顯微鏡,並針對各模式提供優化的預訓練模型。
是的,AxonDeepSeg 完全免費且開源,適用於學術及商業用途。
可以,AxonDeepSeg 可自動從分割圖像計算軸突直徑、g 比率、髓鞘厚度及密度指標。
建議使用 GPU 以加快大型數據集的分割速度,但 CPU 也支援較小規模的分析。
可以,Napari GUI 整合支援互動式修正與微調分割遮罩,提升複雜區域的準確度。
Ilastik
| 開發者 | 歐洲分子生物學實驗室(EMBL)Ilastik 團隊及相關學術合作夥伴 |
| 支援平台 |
|
| 語言 | 英文 |
| 價格 | 免費且開源 |
概覽
Ilastik 是一款強大的 AI 驅動工具,用於互動式影像分割、分類及顯微鏡資料分析。透過隨機森林分類器等機器學習技術,研究人員可對像素進行分割、物件分類、細胞追蹤,並在 2D 及 3D 資料集中執行密度計數。憑藉直觀介面與即時反饋,Ilastik 讓無程式設計經驗的科學家也能輕鬆使用,廣泛應用於細胞生物學、神經科學及生物醫學影像領域。
主要功能
標註代表區域時提供即時反饋,快速獲得分割結果。
根據形態及強度特徵對分割結構進行分類。
追蹤 2D 及 3D 時間序列顯微鏡實驗中的細胞移動與分裂。
在無需明確分割個別物件的情況下,量化擁擠區域的數量。
針對複雜 3D 體積提供半自動分割,操作直觀。
使用無頭命令列模式自動處理多張影像。
下載
入門指南
從官方網站下載適用於您的作業系統的 Ilastik。套件包含所有必要的 Python 依賴,請依照平台指示完成安裝。
開啟 Ilastik,選擇分析工作流程:像素分類、物件分類、追蹤或密度計數。載入您的影像資料集,支援多通道、3D 或時間序列影像。
在影像中標註少量代表性像素或物件。Ilastik 的隨機森林分類器會從標註中學習,自動預測整個資料集的標籤。
將訓練好的模型應用於整個資料集的分割或分類。匯出標記影像、機率圖或量化表格,方便後續分析與視覺化。
利用 Ilastik 的無頭模式,自動處理多張影像,適合大規模分析流程。
限制與注意事項
- 互動式標註對於超大型資料集可能耗時
- 準確度依賴用戶標註的品質與代表性
- 記憶體需求 — 超高解析度或多 GB 資料集可能需大量 RAM
- 複雜資料 — 隨機森林分類器在高度變異或複雜影像資料上可能不及深度神經網絡
常見問題
能,Ilastik 完全支援 3D 體積及時間序列實驗,用於分割、追蹤及多時間點的定量分析。
是的,Ilastik 完全免費且開源,所有用戶均無需授權限制。
不需要,Ilastik 提供直觀的圖形介面與即時反饋,讓無程式設計經驗的研究人員也能輕鬆使用。進階用戶亦可使用命令列批次處理功能。
能,專用的追蹤工作流程可分析 2D 及 3D 時間序列資料中的細胞移動與分裂,並自動追蹤細胞系譜。
分割結果可匯出為標記影像、機率圖或量化表格,方便與後續分析工具及視覺化軟件無縫整合。
這些工具涵蓋初學者至專家級別。許多為免費且開源,促進研究社群中可重現及共享的人工智能工作流程。
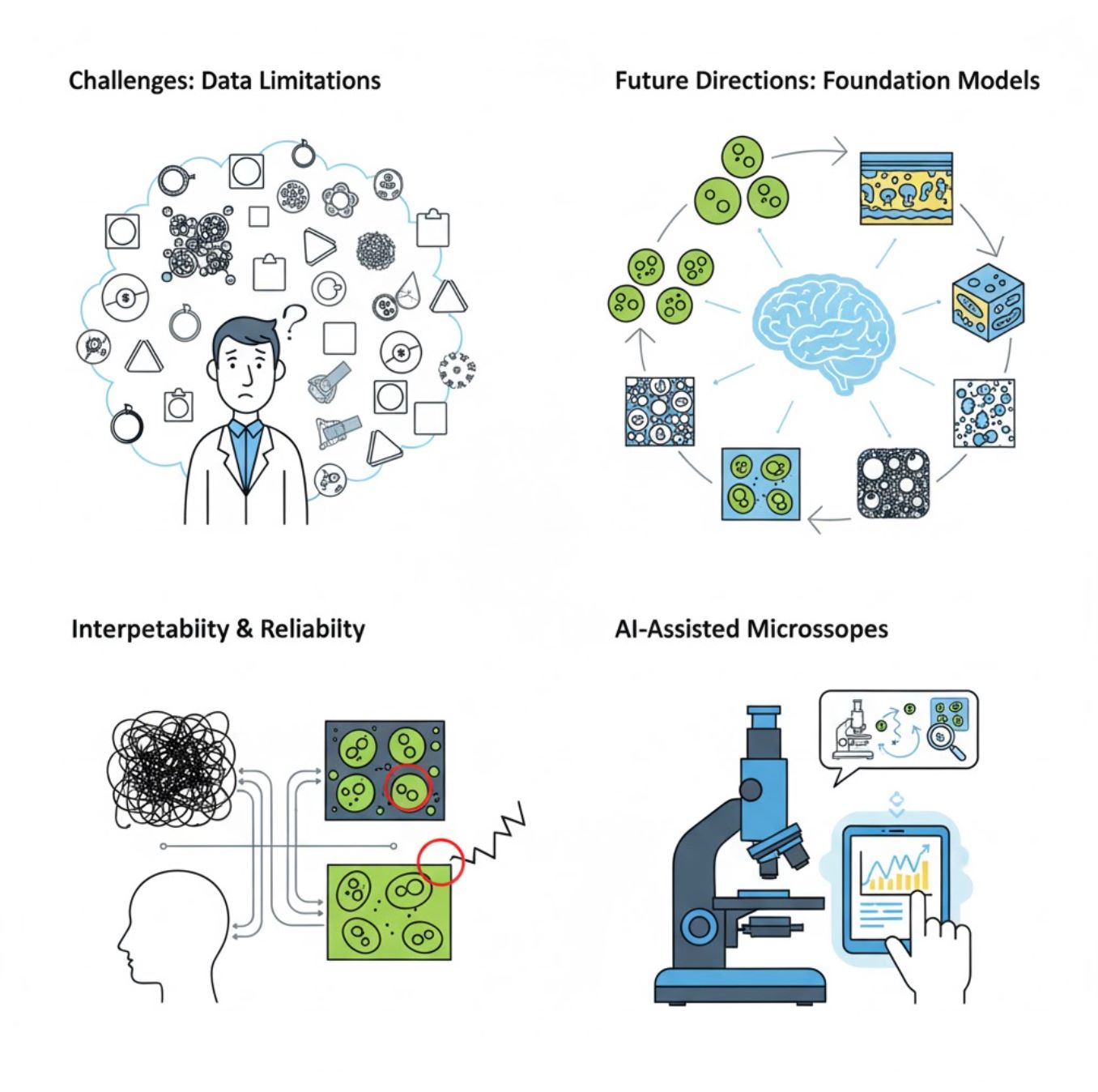
挑戰與未來方向
當前挑戰
新興趨勢
視覺基礎模型
下一代人工智能系統有望減少對特定任務訓練的依賴。
- 如SAM及基於CLIP的系統
- 一套人工智能處理多種顯微鏡任務
- 部署與適應更快速
人工智能輔助顯微鏡
全自動且智慧的顯微鏡系統正逐漸成真。
- 透過大型語言模型實現自然語言控制
- 完全自動化的反饋迴路
- 普及先進顯微鏡技術的使用
主要結論
- 人工智能正迅速改變顯微鏡影像處理,提升準確度與自動化
- 深度學習在複雜且多變的顯微影像中勝過傳統機器學習
- CNN自動從原始像素學習分層特徵,實現穩健分析
- 主要應用包括分割、分類、追蹤、降噪及自動擷取
- 成功依賴高品質資料及專家嚴謹驗證
- 視覺基礎模型與人工智能輔助顯微鏡代表未來發展方向
隨著持續進步與社群努力(開源工具、共享資料集),人工智能將日益成為顯微鏡的「眼睛」核心,助科學家洞察未見之物。







No comments yet. Be the first to comment!