L'IA nell'elaborazione delle immagini al microscopio
L'IA sta rivoluzionando l'elaborazione delle immagini al microscopio con potenti capacità come la segmentazione precisa, la riduzione del rumore, la super-risoluzione e l'acquisizione automatizzata delle immagini. Questo articolo evidenzia gli strumenti essenziali di IA e le tendenze emergenti nella ricerca scientifica.
Le tecniche di IA stanno rivoluzionando la microscopia ottimizzando l'acquisizione delle immagini e automatizzando l'analisi. Nei moderni microscopi intelligenti, i moduli di IA possono regolare i parametri di imaging in tempo reale (ad esempio, messa a fuoco, illuminazione) per minimizzare il fotobleaching e migliorare il segnale. Nel frattempo, gli algoritmi di deep learning possono analizzare dati complessi di immagini per estrarre intuizioni biologiche nascoste e persino collegare le immagini ad altri dati (ad esempio, genomica).
Metodi di IA: Machine Learning vs Deep Learning
I metodi di IA spaziano dal classico machine learning (ML) al moderno deep learning (DL). Ogni approccio ha punti di forza e limiti distinti:
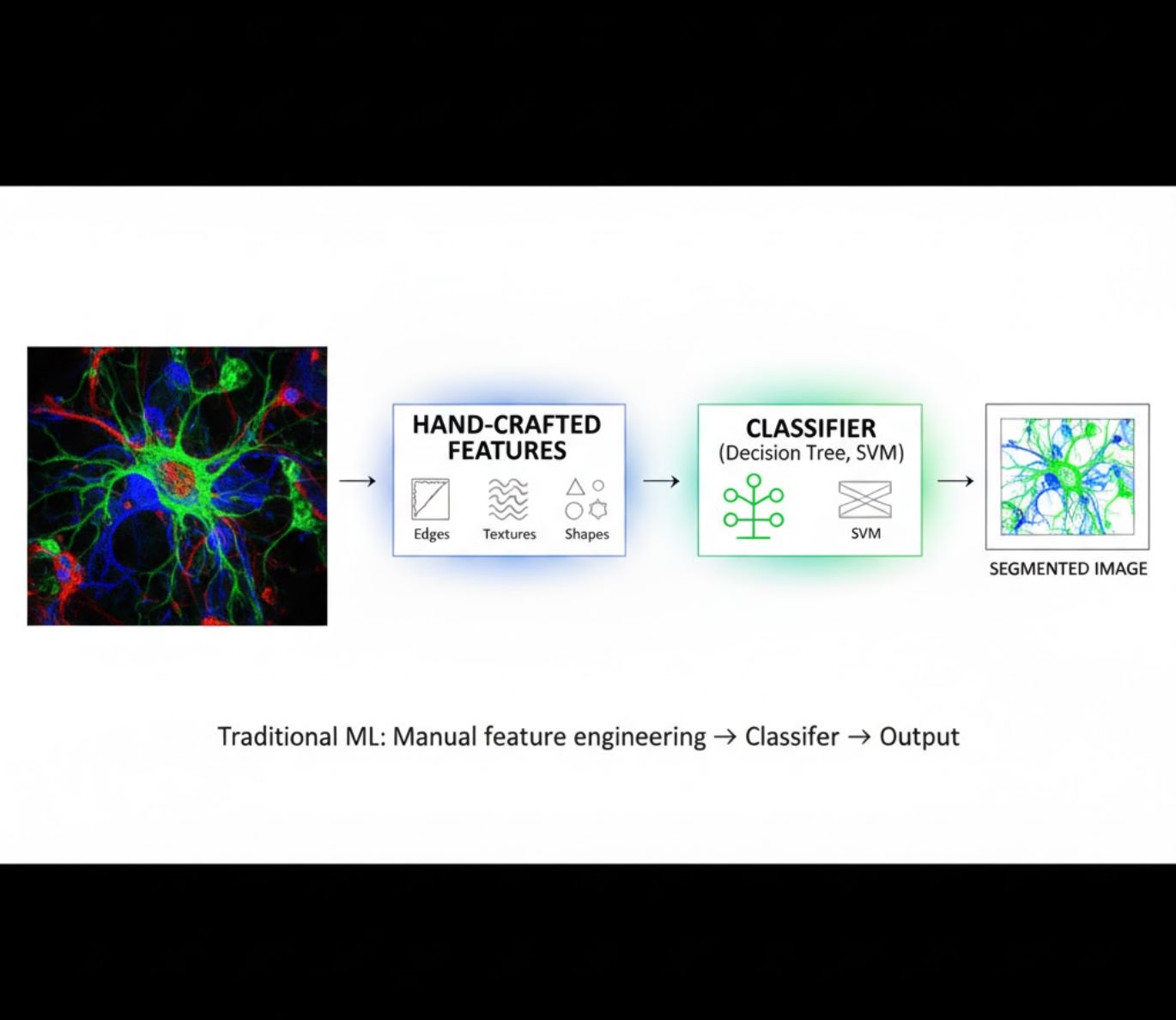
Caratteristiche Artigianali
- I ricercatori creano manualmente caratteristiche delle immagini (bordi, texture, forme)
- Le caratteristiche sono fornite a classificatori (alberi decisionali, SVM)
- Veloce da addestrare
- Difficoltà con immagini complesse o rumorose
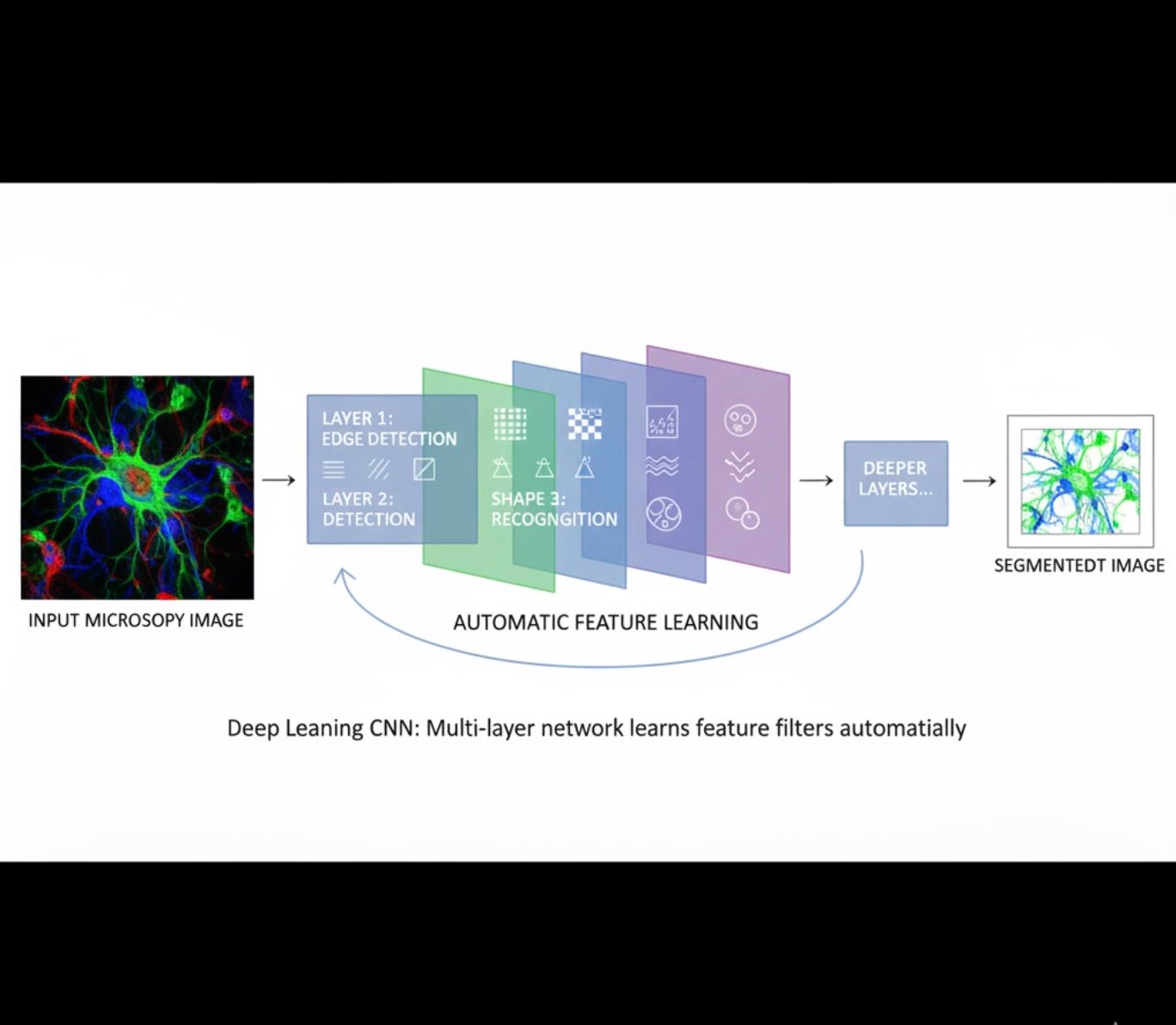
Apprendimento Automatico delle Caratteristiche
- Reti neurali multilivello (CNN) apprendono automaticamente le caratteristiche
- Apprendimento end-to-end dai pixel grezzi
- Molto più robusto alle variazioni
- Cattura texture e strutture complesse in modo affidabile
Come funzionano le CNN: Le reti neurali convoluzionali applicano filtri successivi alle immagini microscopiche, imparando a rilevare pattern semplici (bordi) negli strati iniziali e strutture complesse (forme cellulari, texture) negli strati più profondi. Questo apprendimento gerarchico rende il DL eccezionalmente robusto anche quando i profili di intensità variano significativamente.
Confronto Visivo: Pipeline ML vs DL
Applicazioni Chiave dell'IA in Microscopia
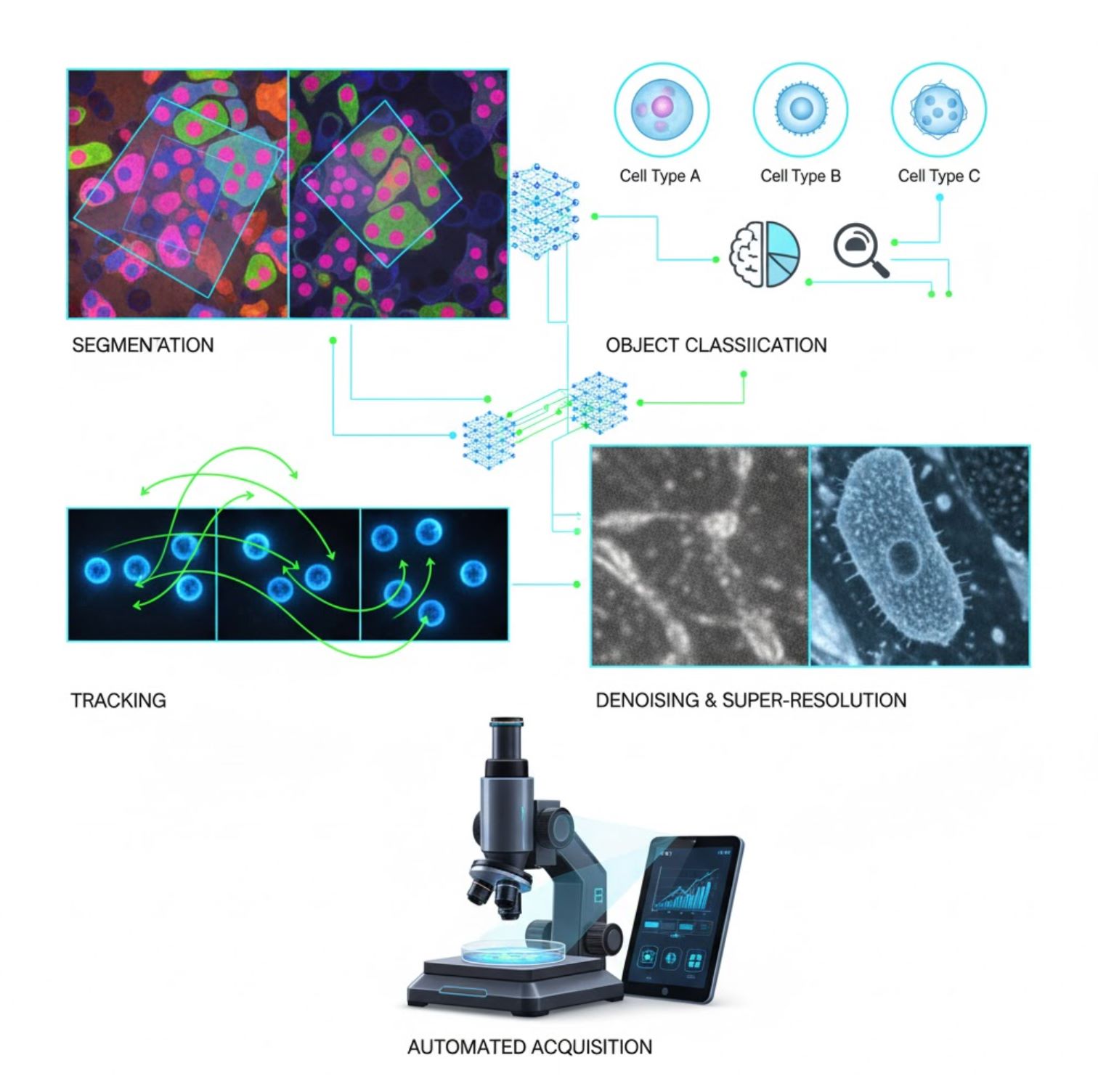
L'IA è ora integrata in molte attività di elaborazione immagini lungo il flusso di lavoro microscopico:
Segmentazione
Suddivisione delle immagini in regioni (ad esempio, identificazione di ogni cellula o nucleo). Le reti profonde come U-Net eccellono in questo compito.
- Segmentazione semantica: etichette di classe per pixel
- Segmentazione per istanza: separazione di oggetti individuali
- Alta precisione su immagini affollate o poco luminose
- Modelli foundation per la visione (es. μSAM) ora adattati alla microscopia
Classificazione degli Oggetti
Dopo la segmentazione, l'IA classifica ogni oggetto con alta precisione.
- Identificazione del tipo cellulare
- Determinazione della fase mitotica
- Rilevamento di indicatori patologici
- Distingue fenotipi sottili difficili da quantificare manualmente
Tracciamento
Nella microscopia a intervalli temporali, l'IA traccia cellule o particelle attraverso i fotogrammi con precisione senza precedenti.
- Il deep learning migliora drasticamente la precisione del tracciamento
- Consente analisi affidabili di cellule in movimento
- Cattura processi biologici dinamici
Riduzione del Rumore e Super-Risoluzione
I modelli di IA migliorano la qualità dell'immagine rimuovendo rumore e sfocature.
- Modelli profondi informati dalla fisica apprendono l'ottica del microscopio
- Ricostruiscono immagini più nitide e prive di artefatti
- Risoluzione superiore con meno artefatti rispetto ai metodi tradizionali
Acquisizione Automatizzata
L'IA guida il microscopio stesso in tempo reale.
- Analizza immagini live per prendere decisioni intelligenti
- Regola automaticamente la messa a fuoco e scansiona aree di interesse
- Riduce la fototossicità e risparmia tempo
- Consente esperimenti di imaging ad alto rendimento e adattativi
Strumenti Popolari di IA nell'Elaborazione delle Immagini al Microscopio
Un ecosistema ricco di strumenti supporta l'IA in microscopia. I ricercatori hanno sviluppato sia software di uso generale sia soluzioni specializzate, molte delle quali open-source:
Cellpose
| Sviluppatori | Carsen Stringer e Marius Pachitariu (gruppo di ricerca MouseLand) |
| Piattaforme supportate |
Richiede Python (installazione via pip/conda). GUI disponibile solo su desktop. |
| Supporto linguistico | Documentazione in inglese; adottato globalmente in laboratori di ricerca in tutto il mondo |
| Modello di prezzo | Gratuito e open-source sotto licenza BSD-3-Clause |
Panoramica
Cellpose è uno strumento avanzato di segmentazione basato su deep learning progettato per immagini microscopiche. Come algoritmo generalista, segmenta con precisione diversi tipi cellulari (nuclei, citoplasma, ecc.) attraverso differenti modalità di imaging senza richiedere riaddestramento del modello. Con funzionalità di intervento umano, i ricercatori possono perfezionare i risultati, adattare il modello ai propri dati e applicare il sistema sia a flussi di lavoro 2D che 3D.
Caratteristiche principali
Funziona immediatamente per una vasta gamma di tipi cellulari, colorazioni e modalità di imaging senza necessità di addestramento personalizzato.
Supporta pile 3D complete usando un approccio "2.5D" che riutilizza modelli 2D per dati volumetrici.
Correggi manualmente i risultati della segmentazione e riaddestra il modello sui tuoi dati personalizzati per una maggiore accuratezza.
Accesso tramite API Python, interfaccia a riga di comando o interfaccia grafica per flussi di lavoro flessibili.
Funzionalità di denoising, deblurring e upsampling per migliorare la qualità delle immagini prima della segmentazione.
Scarica o accedi
Background tecnico
Cellpose è stato introdotto in uno studio fondamentale di Stringer, Wang, Michaelos e Pachitariu, addestrato su un dataset ampio e altamente variegato contenente oltre 70.000 oggetti segmentati. Questa diversità permette al modello di generalizzare tra forme cellulari, dimensioni e impostazioni microscopiche, riducendo significativamente la necessità di addestramento personalizzato nella maggior parte dei casi. Per i dati 3D, Cellpose riutilizza astutamente il suo modello 2D in modo "2.5D", evitando la necessità di dati di addestramento completamente annotati in 3D pur offrendo segmentazione volumetrica. Cellpose 2.0 ha introdotto il riaddestramento con intervento umano, consentendo agli utenti di correggere manualmente le previsioni e riaddestrare sulle proprie immagini per migliorare le prestazioni su dataset specifici.
Installazione e configurazione
Configura un ambiente Python usando conda:
conda create -n cellpose python=3.10Attiva l'ambiente e installa Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposePer iniziare
Modalità GUI
- Avvia la GUI eseguendo:
python -m cellpose - Trascina e rilascia i file immagine (
.tif,.png, ecc.) nell'interfaccia - Seleziona il tipo di modello (es. "cyto" per citoplasma o "nuclei" per nuclei)
- Imposta il diametro cellulare stimato o lascia che Cellpose si auto-calibri
- Clicca per avviare la segmentazione e visualizzare i risultati
Modalità API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Perfeziona e riaddestra
- Dopo aver generato le maschere, correggi la segmentazione nella GUI unendo o eliminando maschere manualmente
- Usa le funzioni di training integrate per riaddestrare sugli esempi corretti
- Migliora le prestazioni del modello sul tuo dataset specifico
Elabora dati 3D
- Carica un TIFF multi-Z o una pila volumetrica
- Usa il flag
--Zstacknella GUI o API per elaborare in 3D - Opzionalmente perfeziona i flussi 3D tramite smoothing o parametri specializzati per una segmentazione migliore
Limitazioni e considerazioni
- Compromesso sulla generalità del modello: Sebbene il modello generalista funzioni ampiamente, forme cellulari o condizioni di imaging molto insolite possono richiedere riaddestramento.
- Sforzo di annotazione: Il training con intervento umano richiede correzioni manuali, che possono essere dispendiose in termini di tempo per dataset grandi.
- Complessità di installazione: L’installazione della GUI può richiedere l’uso della riga di comando, ambienti conda e la gestione delle dipendenze Python — non sempre semplice per chi non è programmatore.
- Solo desktop: Cellpose è progettato per uso desktop; non sono disponibili applicazioni native per Android o iOS.
Domande frequenti
No — Cellpose fornisce modelli generalisti pre-addestrati che spesso funzionano bene senza riaddestramento. Tuttavia, per risultati ottimali su dati speciali o insoliti, è possibile annotare e riaddestrare usando le funzionalità con intervento umano.
Sì — supporta il 3D riutilizzando il modello 2D (cosiddetto "2.5D"), e puoi elaborare pile volumetriche tramite GUI o API.
Una GPU è fortemente consigliata per inferenza e training più rapidi, specialmente su dataset grandi o 3D, ma Cellpose può funzionare anche su macchine solo CPU con prestazioni più lente.
Nella GUI, imposta manualmente il diametro cellulare stimato o lascia che Cellpose lo calibri automaticamente. Puoi perfezionare i risultati e riaddestrare se la segmentazione non è ottimale.
Sì — le versioni più recenti (Cellpose 3) includono modelli di restauro immagini per denoising, deblurring e upsampling per migliorare la qualità della segmentazione prima dell’elaborazione.
StarDist
| Sviluppatori | Uwe Schmidt, Martin Weigert, Coleman Broaddus e Gene Myers |
| Piattaforme Supportate |
|
| Supporto Linguistico | Progetto open source con documentazione e comunità principalmente in inglese |
| Modello di Prezzo | Gratuito e open source. Licenziato sotto BSD-3-Clause |
Panoramica
StarDist è uno strumento di deep learning per la segmentazione delle istanze in immagini microscopiche. Rappresenta ogni oggetto (come nuclei cellulari) come un poligono star-convesso in 2D o un poliedro in 3D, permettendo un rilevamento e una separazione accurata di oggetti densi o sovrapposti. Grazie alla sua architettura robusta, StarDist è ampiamente utilizzato per la segmentazione automatica di cellule e nuclei in microscopia a fluorescenza, istopatologia e altre applicazioni di bioimmagini.
Caratteristiche Principali
Segmentazione delle istanze altamente accurata usando poligoni star-convessi (2D) e poliedri (3D) per un rilevamento affidabile degli oggetti.
Modelli dedicati per immagini 2D e dati volumetrici 3D per un’analisi microscopica completa.
Modelli pronti all’uso per nuclei fluorescenti, istologia colorata H&E e altri scenari comuni di imaging.
Classifica gli oggetti rilevati in classi distinte (es. diversi tipi cellulari) in una singola esecuzione di segmentazione.
Integrazione fluida con ImageJ/Fiji, QuPath e napari per flussi di lavoro GUI accessibili.
Valutazione completa della segmentazione delle istanze, inclusi precisione, richiamo, F1 score e qualità panottica.
Contesto Tecnico
Introdotto originariamente in un articolo MICCAI 2018, l’innovazione principale di StarDist è la previsione delle distanze radiali lungo raggi fissi combinata con la probabilità dell’oggetto per ogni pixel, permettendo una ricostruzione accurata di forme star-convesse. Questo approccio segmenta in modo affidabile oggetti molto vicini difficili da separare con metodi tradizionali basati su pixel o bounding box.
Sviluppi recenti hanno esteso StarDist alle immagini istopatologiche, consentendo non solo la segmentazione dei nuclei ma anche la classificazione multi-classe degli oggetti rilevati. Il metodo ha ottenuto prestazioni di punta in competizioni come la sfida CoNIC (Colon Nuclei Identification and Counting).
Scarica o Accedi
Installazione e Configurazione
Installa TensorFlow (versione 1.x o 2.x) come prerequisito per StarDist.
Usa pip per installare il pacchetto Python StarDist:
pip install stardistPer napari:
pip install stardist-napariPer QuPath: Installa l’estensione StarDist trascinando il file .jar in QuPath.
Per ImageJ/Fiji: Usa il gestore plugin integrato o l’installazione manuale tramite il menu plugin.
Esecuzione della Segmentazione
Carica un modello pre-addestrato, normalizza l’immagine ed esegui la predizione:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Apri la tua immagine in napari, seleziona il plugin StarDist, scegli un modello pre-addestrato o personalizzato ed esegui la predizione direttamente dall’interfaccia grafica.
Usa il plugin StarDist dal menu Plugin per applicare un modello sul tuo stack di immagini con un’interfaccia intuitiva.
Dopo aver installato l’estensione, esegui il rilevamento StarDist tramite la console di scripting o l’interfaccia grafica di QuPath per l’analisi istopatologica.
Addestramento e Messa a Punto
Crea immagini di etichettatura ground-truth dove ogni oggetto è univocamente etichettato. Usa strumenti di annotazione come LabKit, QuPath o Fiji per preparare il tuo dataset.
Usa l’API Python di StarDist per addestrare un nuovo modello o affinare uno esistente con i tuoi dati annotati personalizzati.
Opzioni di Post-Processing
- Applica la soppressione non massima (NMS) per eliminare forme candidate ridondanti
- Usa StarDist OPP (Object Post-Processing) per unire maschere di forme non star-convesse
Limitazioni e Considerazioni
- L’assunzione star-convessa potrebbe non modellare perfettamente forme altamente non-convesse o molto irregolari
- Complessità di installazione: installazioni personalizzate richiedono un compilatore C++ compatibile per costruire le estensioni
- L’accelerazione GPU dipende da versioni compatibili di TensorFlow, CUDA e cuDNN
- Alcuni utenti segnalano problemi nell’esecuzione del plugin ImageJ a causa della configurazione Java
Domande Frequenti
StarDist funziona con diversi tipi di immagini, inclusi fluorescenza, campo chiaro e istopatologia (es. H&E), grazie ai suoi modelli pre-addestrati flessibili e all’adattabilità a diverse modalità di imaging.
Sì — StarDist supporta la segmentazione delle istanze 3D usando poliedri star-convessi per dati volumetrici, estendendo le capacità 2D all’analisi completa in 3D.
Non necessariamente. Sono disponibili modelli pre-addestrati che spesso funzionano bene subito. Tuttavia, per dati specializzati o nuovi, annotare e addestrare modelli personalizzati migliora significativamente l’accuratezza.
StarDist si integra con napari, ImageJ/Fiji e QuPath, permettendo di eseguire la segmentazione da GUI senza programmare. Supporta anche l’uso diretto tramite API Python per flussi di lavoro avanzati.
StarDist fornisce funzioni integrate per calcolare metriche comuni di segmentazione delle istanze, inclusi precisione, richiamo, F1 score e qualità panottica per valutare le prestazioni della segmentazione.
SAM
Informazioni sull'applicazione
| Sviluppatore | Meta AI Research (FAIR) |
| Dispositivi supportati |
|
| Lingua e disponibilità | Modello di base open-source disponibile a livello globale; documentazione in inglese |
| Prezzo | Gratuito — open-source sotto licenza Meta tramite GitHub e integrazione MIB |
Panoramica generale
SAM (Segment Anything Model) è un potente modello di intelligenza artificiale di base creato da Meta che consente la segmentazione interattiva e automatica di praticamente qualsiasi oggetto nelle immagini. Utilizzando prompt come punti, riquadri di delimitazione o maschere approssimative, SAM genera maschere di segmentazione senza richiedere un riaddestramento specifico per il compito. Nella ricerca in microscopia, la flessibilità di SAM è stata adattata per la segmentazione cellulare, il rilevamento di organelli e l’analisi istopatologica, offrendo una soluzione scalabile per i ricercatori che necessitano di uno strumento di segmentazione generalista e promptabile.
Introduzione dettagliata
Originariamente addestrato da Meta su oltre 1 miliardo di maschere distribuite su 11 milioni di immagini, SAM è stato progettato come modello di base promptabile per la segmentazione con prestazioni "zero-shot" su domini nuovi. Nella ricerca di imaging medico, SAM è stato valutato per la segmentazione di intere vetrini patologici, il rilevamento di tumori e l’identificazione di nuclei cellulari. Tuttavia, le sue prestazioni su istanze densamente raggruppate — come i nuclei cellulari — sono variabili: anche con prompt estesi (ad esempio, 20 clic o riquadri), la segmentazione zero-shot può incontrare difficoltà in immagini complesse di microscopia.
Per superare questa limitazione, sono emersi adattamenti specifici per dominio:
- SAMCell — Affinato su grandi dataset di microscopia per una segmentazione zero-shot efficace su diversi tipi cellulari senza riaddestramento per esperimento
- μSAM — Riaddestrato su oltre 17.000 immagini di microscopia annotate manualmente per migliorare la precisione su piccole strutture cellulari
Caratteristiche principali
Interazione flessibile usando punti, riquadri e maschere per un controllo preciso.
Esegue la segmentazione senza affinamento su nuovi domini di immagini.
Adattabile per microscopia e istopatologia tramite riaddestramento few-shot o basato su prompt.
Disponibile in Microscopy Image Browser (MIB) con supporto per segmentazione 3D e interpolata.
IDCC-SAM consente il conteggio automatico delle cellule in immunocitochimica senza annotazioni manuali.
Download o accesso
Guida utente
- Aprire Microscopy Image Browser e navigare al pannello di segmentazione SAM
- Configurare l’interprete Python e selezionare tra i modelli SAM-1 o SAM-2
- Per l’accelerazione GPU, selezionare "cuda" nell’ambiente di esecuzione (consigliato per prestazioni ottimali)
- Prompt puntuali: Cliccare su un oggetto per definire un seme positivo; usare Shift + clic per espandere e Ctrl + clic per semi negativi
- Stack 3D: Usare la modalità interattiva 3D — cliccare su una fetta, shift-scroll e interpolare semi tra le fette
- Modalità di regolazione: Sostituire, aggiungere, sottrarre maschere o creare un nuovo livello secondo necessità
- Usare l’opzione "Automatic everything" di MIB nel pannello SAM-2 per segmentare tutti gli oggetti visibili in una regione
- Revisionare e perfezionare le maschere dopo la segmentazione secondo necessità
- Usare pipeline di affinamento basate su prompt (es. "All-in-SAM") per generare annotazioni a livello di pixel da prompt utente sparsi
- Per il conteggio cellulare, applicare IDCC-SAM, che utilizza SAM in una pipeline zero-shot con post-elaborazione
- Per segmentazione cellulare ad alta precisione, usare SAMCell, affinato su immagini di cellule in microscopia
Limitazioni e considerazioni
- Prestazioni zero-shot incoerenti su strutture dense o sovrapposte senza affinamento di dominio
- La qualità della segmentazione dipende molto dalla progettazione e strategia del prompt
- GPU fortemente consigliata; l’inferenza su CPU è molto lenta
- Difficoltà con immagini a risoluzione molto alta di interi vetrini e strutture tissutali multi-scala
- L’affinamento o l’adattamento di SAM per microscopia può richiedere competenze di machine learning
Domande frequenti
Sì — tramite adattamenti come SAMCell, che affina SAM su dataset di microscopia specifici per compiti di segmentazione cellulare.
Non sempre. Con IDCC-SAM, è possibile effettuare il conteggio cellulare zero-shot senza annotazioni manuali.
Usi l’affinamento basato su prompt (es. "All-in-SAM") o versioni pre-addestrate per microscopia come μSAM, addestrato su oltre 17.000 immagini di microscopia annotate.
Anche se possibile su CPU, la GPU è fortemente raccomandata per una velocità di inferenza pratica e segmentazione interattiva in tempo reale.
Sì — l’integrazione SAM-2 di MIB supporta la segmentazione 3D con interpolazione di semi tra le fette per analisi volumetriche.
AxonDeepSeg
| Sviluppatore | NeuroPoly Lab presso Polytechnique Montréal e Université de Montréal |
| Piattaforme supportate |
|
| Lingua | Documentazione in inglese; strumento open source utilizzato a livello globale |
| Prezzo | Gratuito e open source |
Panoramica
AxonDeepSeg è uno strumento basato su intelligenza artificiale per la segmentazione automatica di assoni e mielina in immagini microscopiche. Utilizzando reti neurali convoluzionali, fornisce una segmentazione precisa a tre classi (assone, mielina, sfondo) su diverse modalità di imaging, tra cui TEM, SEM e microscopia a campo chiaro. Automatizzando le misurazioni morfometriche come diametro dell’assone, rapporto g e spessore della mielina, AxonDeepSeg semplifica l’analisi quantitativa nella ricerca neuroscientifica, riducendo significativamente il tempo di annotazione manuale e migliorando la riproducibilità.
Caratteristiche principali
Modelli pronti all’uso ottimizzati per le modalità di microscopia TEM, SEM e a campo chiaro.
Classificazione precisa delle regioni di assone, mielina e sfondo nelle immagini microscopiche.
Calcolo automatico di diametro dell’assone, rapporto g, spessore della mielina e metriche di densità.
L’integrazione con l’interfaccia Napari consente la rifinitura manuale delle maschere di segmentazione per una maggiore accuratezza.
Si integra perfettamente in pipeline personalizzate per l’analisi su larga scala del tessuto neurale.
Script di test completi garantiscono la riproducibilità e risultati affidabili nella segmentazione.
Dettagli tecnici
Sviluppato dal NeuroPoly Lab, AxonDeepSeg sfrutta il deep learning per fornire segmentazioni ad alta precisione per applicazioni neuroscientifiche. Sono disponibili modelli pre-addestrati per diverse modalità di microscopia, garantendo versatilità tra le tecniche di imaging. Lo strumento si integra con Napari, permettendo correzioni interattive delle maschere di segmentazione, migliorando l’accuratezza su dataset complessi. AxonDeepSeg calcola metriche morfometriche chiave, supportando studi ad alto rendimento sulla struttura e patologia del tessuto neurale. Il suo framework basato su Python consente l’integrazione in pipeline personalizzate per analisi su larga scala della morfologia di assoni e mielina.
Download o Accesso
Installazione e configurazione
Assicurarsi che Python 3.8 o versione successiva sia installato, quindi installare AxonDeepSeg e Napari usando pip:
pip install axondeepseg napariEseguire gli script di test forniti per confermare che tutti i componenti siano installati correttamente e funzionanti.
Importare immagini microscopiche (TEM, SEM o a campo chiaro) in Napari o nel proprio ambiente Python.
Scegliere il modello pre-addestrato appropriato per la modalità di imaging e avviare la segmentazione per generare maschere di assone e mielina.
Calcolare automaticamente le misurazioni morfometriche tra cui diametro dell’assone, rapporto g, densità e spessore della mielina, quindi esportare i risultati in formato CSV.
Utilizzare l’interfaccia Napari per regolare manualmente le maschere di segmentazione dove necessario, unendo o eliminando maschere per migliorare l’accuratezza.
Considerazioni importanti
- Le prestazioni possono diminuire su modalità di imaging nuove o non addestrate
- Potrebbero essere necessarie correzioni manuali per regioni complesse o difficili
- GPU consigliata per elaborazione più veloce di grandi dataset; supportata anche l’elaborazione su CPU
Domande frequenti
AxonDeepSeg supporta TEM (Microscopia Elettronica a Trasmissione), SEM (Microscopia Elettronica a Scansione) e microscopia a campo chiaro con modelli pre-addestrati ottimizzati per ciascuna modalità.
Sì, AxonDeepSeg è completamente gratuito e open source, disponibile per uso accademico e commerciale.
Sì, AxonDeepSeg calcola automaticamente diametro dell’assone, rapporto g, spessore della mielina e metriche di densità dalle immagini segmentate.
La GPU è consigliata per una segmentazione più veloce di grandi dataset, ma è supportata anche l’elaborazione su CPU per analisi di dimensioni minori.
Sì, l’integrazione con l’interfaccia Napari consente correzioni interattive e rifiniture delle maschere di segmentazione per una maggiore accuratezza in regioni complesse.
Ilastik
| Sviluppatore | Team Ilastik presso l’European Molecular Biology Laboratory (EMBL) e partner accademici associati |
| Piattaforme supportate |
|
| Lingua | Inglese |
| Prezzo | Gratuito e open source |
Panoramica
Ilastik è uno strumento potente, basato su intelligenza artificiale, per la segmentazione interattiva, classificazione e analisi di dati microscopici. Utilizzando tecniche di machine learning come i classificatori Random Forest, consente ai ricercatori di segmentare pixel, classificare oggetti, tracciare cellule nel tempo e effettuare conteggi di densità in dataset 2D e 3D. Con la sua interfaccia intuitiva e il feedback in tempo reale, Ilastik è accessibile a scienziati senza competenze di programmazione ed è ampiamente adottato in biologia cellulare, neuroscienze e imaging biomedico.
Caratteristiche principali
Feedback in tempo reale mentre si annotano regioni rappresentative per risultati di segmentazione immediati.
Categorizzazione delle strutture segmentate basata su caratteristiche morfologiche e di intensità.
Traccia il movimento e la divisione cellulare in esperimenti di microscopia temporale 2D e 3D.
Quantifica regioni affollate senza segmentazione esplicita di singoli oggetti.
Segmentazione semi-automatica per volumi 3D complessi con interazione intuitiva.
Elabora automaticamente più immagini usando la modalità headless da linea di comando.
Download
Guida introduttiva
Scarichi Ilastik per il suo sistema operativo dal sito ufficiale. Il pacchetto include tutte le dipendenze Python necessarie, quindi segua le istruzioni di installazione per la sua piattaforma.
Apra Ilastik e scelga il flusso di lavoro di analisi: Classificazione dei pixel, Classificazione degli oggetti, Tracciamento o Conteggio di densità. Carichi il suo dataset di immagini, che può includere immagini multicanale, 3D o temporali.
Etichetti alcuni pixel o oggetti rappresentativi nelle sue immagini. Il classificatore Random Forest di Ilastik apprende da queste annotazioni e predice automaticamente le etichette su tutto il dataset.
Applichi il modello addestrato per segmentare o classificare l’intero dataset. Esporti i risultati come immagini etichettate, mappe di probabilità o tabelle quantitative per analisi e visualizzazioni successive.
Utilizzi la modalità headless di Ilastik per elaborare automaticamente più immagini senza intervento manuale, ideale per pipeline di analisi su larga scala.
Limitazioni e considerazioni
- L’etichettatura interattiva può richiedere molto tempo per dataset molto grandi
- La precisione dipende dalla qualità e rappresentatività delle annotazioni dell’utente
- Requisiti di memoria — dataset ad altissima risoluzione o multi-gigabyte possono richiedere molta RAM
- Dati complessi — i classificatori Random Forest possono avere prestazioni inferiori rispetto a reti neurali profonde su dati di imaging altamente variabili o complessi
Domande frequenti
Sì, Ilastik supporta completamente volumi 3D e esperimenti temporali per segmentazione, tracciamento e analisi quantitativa su più punti temporali.
Sì, Ilastik è completamente gratuito e open source, disponibile per tutti gli utenti senza restrizioni di licenza.
No, Ilastik offre un’interfaccia grafica intuitiva con feedback in tempo reale, rendendolo accessibile a ricercatori senza competenze di programmazione. Gli utenti avanzati possono anche utilizzare l’elaborazione batch da linea di comando.
Sì, il flusso di lavoro dedicato al tracciamento consente l’analisi del movimento e della divisione cellulare in dataset temporali 2D e 3D con tracciamento automatico della linea cellulare.
I risultati della segmentazione possono essere esportati come immagini etichettate, mappe di probabilità o tabelle quantitative, permettendo un’integrazione fluida con strumenti di analisi e software di visualizzazione successivi.
Questi strumenti coprono livelli da principiante a esperto. Molti sono gratuiti e open-source, facilitando flussi di lavoro di IA riproducibili e condivisibili nella comunità scientifica.
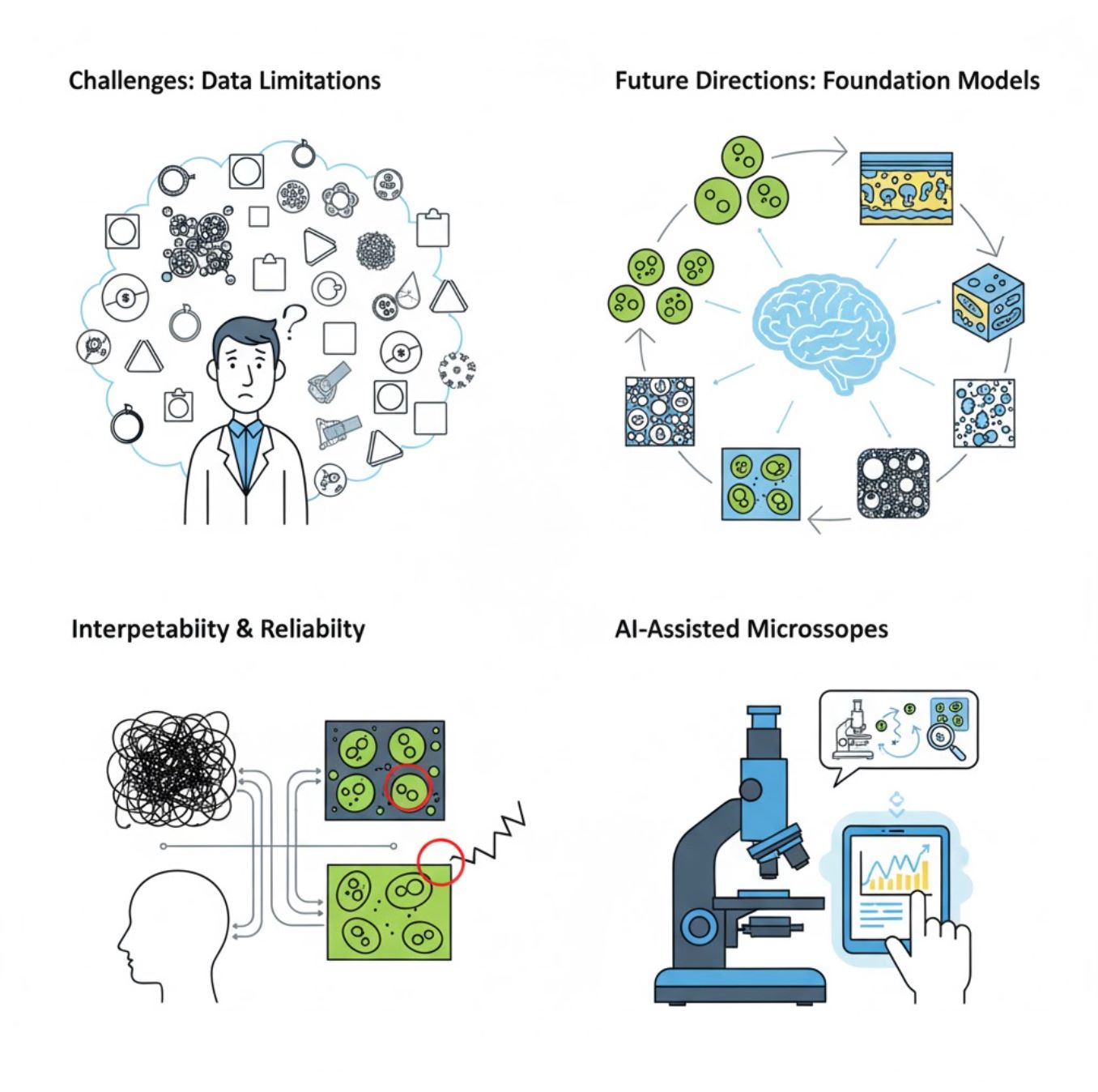
Sfide e Direzioni Future
Sfide Attuali
Tendenze Emergenti
Modelli Foundation per la Visione
I sistemi di IA di nuova generazione promettono di ridurre la necessità di addestramento specifico per compito.
- Modelli come SAM e sistemi basati su CLIP
- Un'unica IA gestisce molteplici compiti di microscopia
- Distribuzione e adattamento più rapidi
Microscopi Assistiti da IA
Sistemi di microscopia completamente autonomi e intelligenti stanno diventando realtà.
- Controllo tramite linguaggio naturale con LLM
- Loop di feedback completamente automatizzati
- Democratizza l'accesso alla microscopia avanzata
Punti Chiave
- L'IA sta rapidamente trasformando l'elaborazione delle immagini al microscopio con maggiore precisione e automazione
- Il deep learning supera il machine learning tradizionale su immagini microscopiche complesse e variabili
- Le CNN apprendono automaticamente caratteristiche gerarchiche dai pixel grezzi per un'analisi robusta
- Applicazioni chiave includono segmentazione, classificazione, tracciamento, riduzione del rumore e acquisizione automatizzata
- Il successo dipende da dati di qualità e da una valida convalida da parte di esperti
- I modelli foundation per la visione e i microscopi assistiti da IA rappresentano il futuro del settore
Con continui progressi e sforzi comunitari (strumenti open-source, dataset condivisi), l'IA diventerà sempre più parte integrante dell'"occhio" del microscopio, aiutando gli scienziati a vedere ciò che è invisibile.







No comments yet. Be the first to comment!