IA en el procesamiento de imágenes de microscopio
La IA está revolucionando el procesamiento de imágenes de microscopio con capacidades potentes como segmentación precisa, reducción de ruido, superresolución y adquisición automática de imágenes. Este artículo destaca herramientas esenciales de IA y tendencias emergentes en la investigación científica.
Las técnicas de IA están revolucionando la microscopía al optimizar la adquisición de imágenes y automatizar el análisis. En los microscopios inteligentes modernos, los módulos de IA pueden ajustar parámetros de imagen en tiempo real (por ejemplo, enfoque, iluminación) para minimizar el fotoblanqueo y mejorar la señal. Mientras tanto, los algoritmos de aprendizaje profundo pueden analizar datos complejos de imágenes para extraer conocimientos biológicos ocultos e incluso vincular imágenes con otros datos (por ejemplo, genómica).
Métodos de IA: Aprendizaje Automático vs Aprendizaje Profundo
Los métodos de IA van desde el clásico aprendizaje automático (ML) hasta el moderno aprendizaje profundo (DL). Cada enfoque tiene fortalezas y limitaciones distintas:
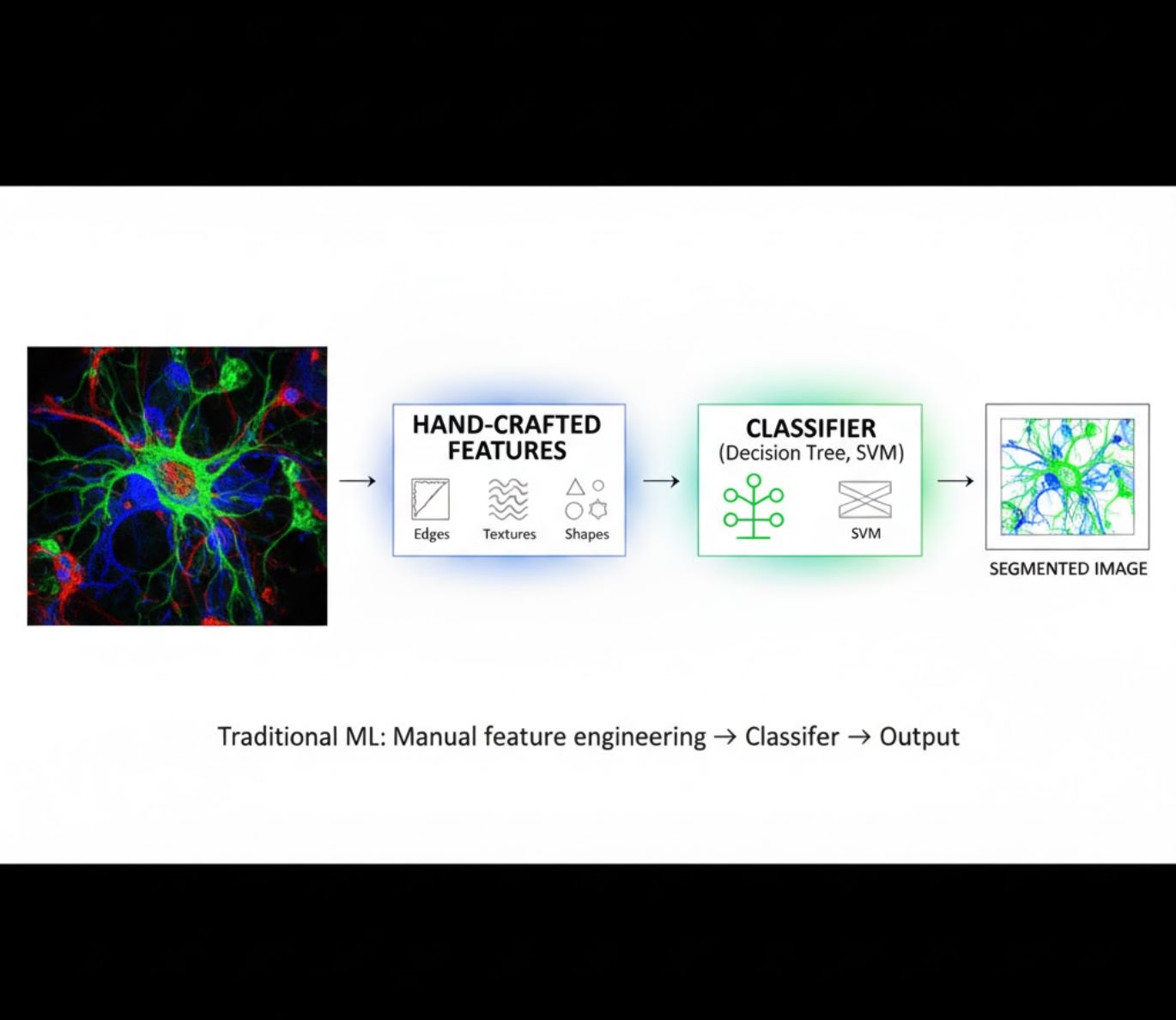
Características Diseñadas Manualmente
- Los investigadores diseñan manualmente características de imagen (bordes, texturas, formas)
- Las características se alimentan a clasificadores (árboles de decisión, SVM)
- Rápido de entrenar
- Dificultad con imágenes complejas o ruidosas
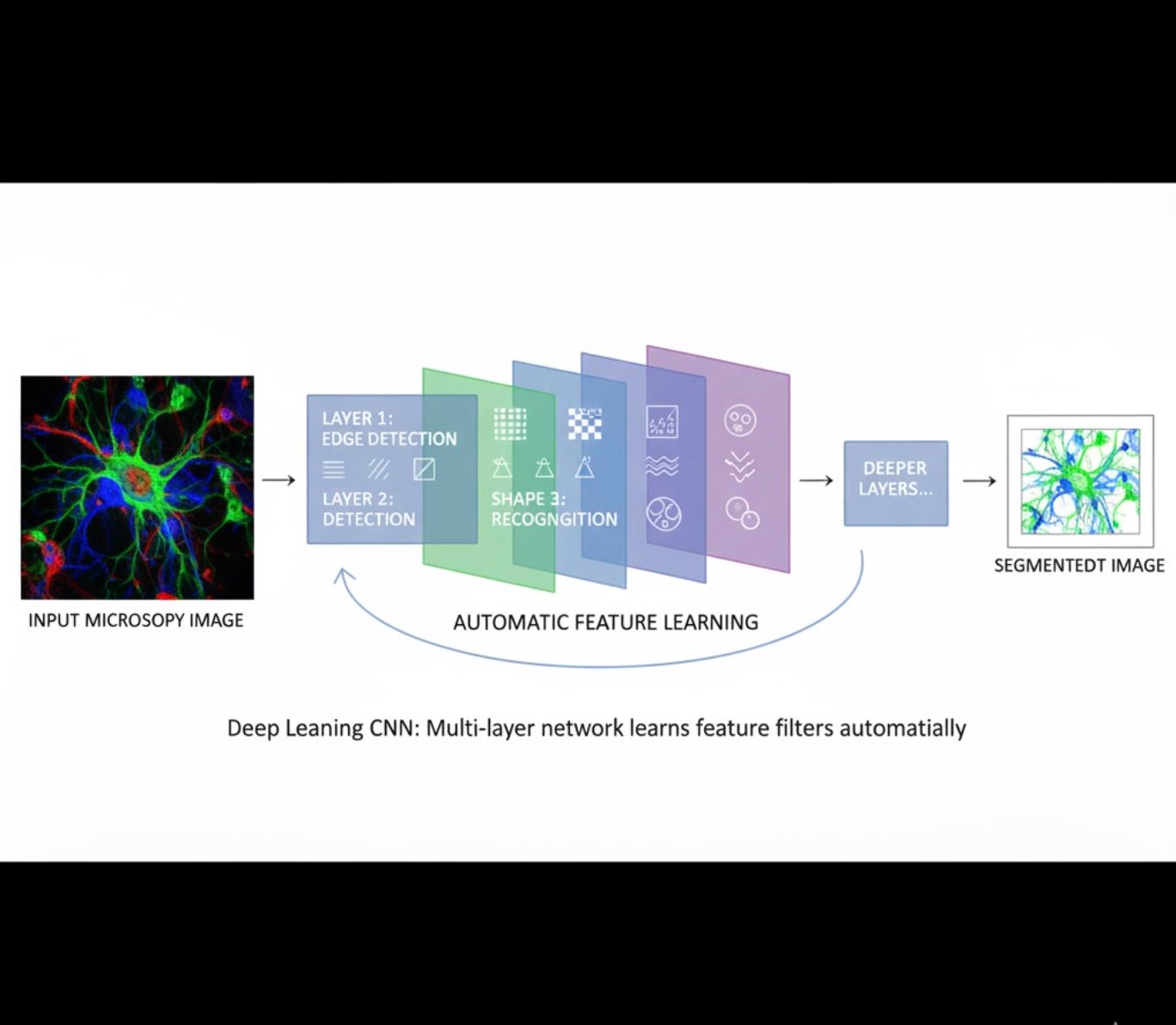
Aprendizaje Automático de Características
- Redes neuronales multicapa (CNN) aprenden características automáticamente
- Aprendizaje de extremo a extremo desde píxeles en bruto
- Mucho más robusto a variaciones
- Captura texturas y estructuras intrincadas de forma confiable
Cómo funcionan las CNN: Las redes neuronales convolucionales aplican filtros sucesivos a imágenes de microscopía, aprendiendo a detectar patrones simples (bordes) en capas iniciales y estructuras complejas (formas celulares, texturas) en capas profundas. Este aprendizaje jerárquico hace que el DL sea excepcionalmente robusto incluso cuando los perfiles de intensidad varían significativamente.
Comparación Visual: Flujos de ML vs DL
Aplicaciones Clave de IA en Microscopía
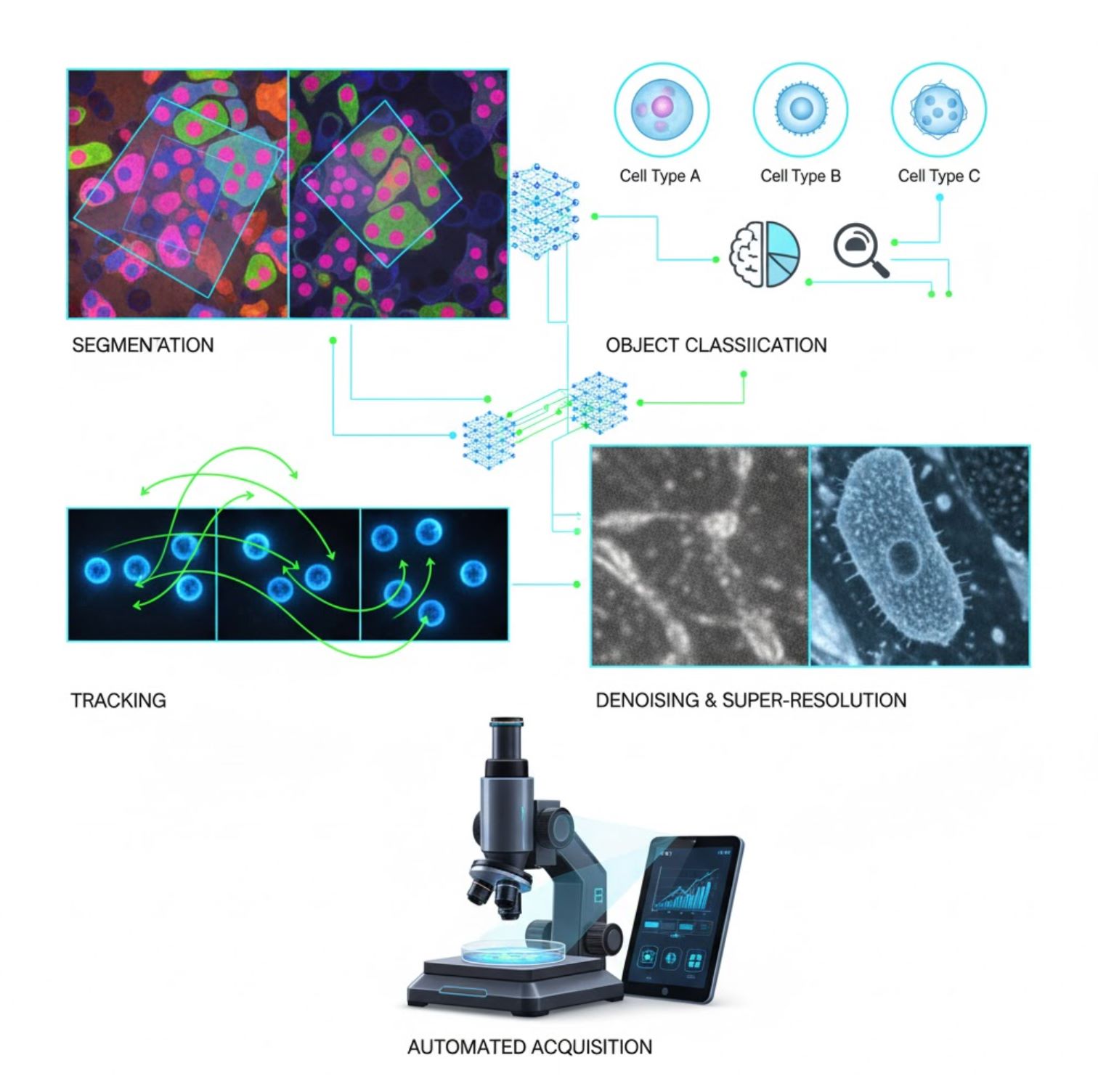
La IA está ahora integrada en muchas tareas de procesamiento de imágenes a lo largo del flujo de trabajo en microscopía:
Segmentación
Dividir imágenes en regiones (por ejemplo, identificar cada célula o núcleo). Redes profundas como U-Net sobresalen en esta tarea.
- Segmentación semántica: Etiquetas de clase por píxel
- Segmentación de instancias: Separar objetos individuales
- Alta precisión en imágenes saturadas o tenues
- Modelos base de visión (por ejemplo, μSAM) ahora adaptados para microscopía
Clasificación de Objetos
Después de la segmentación, la IA clasifica cada objeto con alta precisión.
- Identificación del tipo celular
- Determinación de la etapa mitótica
- Detección de indicadores patológicos
- Distingue fenotipos sutiles difíciles de cuantificar manualmente
Seguimiento
En microscopía de lapso de tiempo, la IA rastrea células o partículas a través de fotogramas con precisión sin precedentes.
- El aprendizaje profundo mejora dramáticamente la precisión del seguimiento
- Permite análisis confiables de células en movimiento
- Captura procesos biológicos dinámicos
Reducción de Ruido y Superresolución
Los modelos de IA mejoran la calidad de imagen eliminando ruido y desenfoque.
- Modelos profundos informados por física aprenden la óptica del microscopio
- Reconstruyen imágenes más nítidas y sin artefactos
- Mayor resolución con menos artefactos comparado con métodos tradicionales
Adquisición Automatizada
La IA guía el microscopio en tiempo real.
- Analiza imágenes en vivo para tomar decisiones inteligentes
- Ajusta automáticamente el enfoque y escanea áreas de interés
- Reduce la fototoxicidad y ahorra tiempo
- Permite experimentos de imagen de alto rendimiento y adaptativos
Herramientas Populares de IA en el Procesamiento de Imágenes de Microscopio
Un ecosistema completo de herramientas que soportan la inteligencia artificial en microscopía. Los investigadores han desarrollado tanto software de uso general como especializado, muchos de ellos de código abierto:
Cellpose
| Desarrolladores | Carsen Stringer y Marius Pachitariu (grupo de investigación MouseLand) |
| Plataformas Soportadas |
Requiere Python (instalación con pip/conda). GUI disponible solo en escritorio. |
| Soporte de Idioma | Documentación en inglés; adoptado globalmente en laboratorios de investigación en todo el mundo |
| Modelo de Precio | Gratis y de código abierto bajo licencia BSD-3-Clause |
Resumen
Cellpose es una herramienta avanzada de segmentación basada en aprendizaje profundo diseñada para imágenes de microscopía. Como algoritmo generalista, segmenta con precisión diversos tipos celulares (núcleos, citoplasma, etc.) en diferentes modalidades de imagen sin requerir reentrenamiento del modelo. Con capacidades de intervención humana, los investigadores pueden refinar resultados, adaptar el modelo a sus datos y aplicar el sistema tanto en flujos de trabajo 2D como 3D.
Características Clave
Funciona listo para usar con una amplia variedad de tipos celulares, tinciones y modalidades de imagen sin entrenamiento personalizado.
Soporta pilas 3D completas usando un enfoque "2.5D" que reutiliza modelos 2D para datos volumétricos.
Corrige manualmente los resultados de segmentación y reentrena el modelo con tus datos personalizados para mayor precisión.
Acceso vía API de Python, interfaz de línea de comandos o interfaz gráfica para flujos de trabajo flexibles.
Capacidades de reducción de ruido, desenfoque y aumento de resolución para mejorar la calidad de imagen antes de la segmentación.
Descargar o Acceder
Antecedentes Técnicos
Cellpose fue presentado en un estudio seminal por Stringer, Wang, Michaelos y Pachitariu, entrenado con un conjunto de datos grande y muy variado que contiene más de 70,000 objetos segmentados. Esta diversidad permite que el modelo generalice a través de formas, tamaños celulares y configuraciones de microscopía, reduciendo significativamente la necesidad de entrenamiento personalizado en la mayoría de los casos. Para datos 3D, Cellpose reutiliza inteligentemente su modelo 2D en un modo "2.5D", evitando la necesidad de datos de entrenamiento completamente anotados en 3D, mientras entrega segmentación volumétrica. Cellpose 2.0 introdujo el reentrenamiento con intervención humana, permitiendo a los usuarios corregir manualmente predicciones y reentrenar con sus propias imágenes para mejorar el rendimiento en conjuntos de datos específicos.
Instalación y Configuración
Configura un entorno Python usando conda:
conda create -n cellpose python=3.10Activa el entorno e instala Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposePrimeros Pasos
Modo GUI
- Inicia la GUI ejecutando:
python -m cellpose - Arrastra y suelta archivos de imagen (
.tif,.png, etc.) en la interfaz - Selecciona el tipo de modelo (por ejemplo, "cyto" para citoplasma o "nuclei" para núcleos)
- Establece el diámetro celular estimado o deja que Cellpose lo calibre automáticamente
- Haz clic para iniciar la segmentación y ver los resultados
Modo API Python
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Refinar y Reentrenar
- Después de generar máscaras, corrige la segmentación en la GUI fusionando o eliminando máscaras manualmente
- Usa las funciones integradas de entrenamiento para reentrenar con los ejemplos corregidos
- Mejora el rendimiento del modelo en tu conjunto de datos específico
Procesar Datos 3D
- Carga un TIFF multi-Z o pila volumétrica
- Usa la bandera
--Zstacken la GUI o API para procesar como 3D - Opcionalmente, refina los flujos 3D mediante suavizado o parámetros especializados para mejor segmentación
Limitaciones y Consideraciones
- Compromiso en la Generalidad del Modelo: Aunque el modelo generalista funciona ampliamente, formas celulares muy inusuales o condiciones de imagen pueden requerir reentrenamiento.
- Esfuerzo de Anotación: El entrenamiento con intervención humana requiere correcciones manuales, lo que puede ser laborioso para conjuntos de datos grandes.
- Complejidad de Instalación: La instalación de la GUI puede requerir uso de línea de comandos, entornos conda y gestión de dependencias Python — no siempre sencillo para usuarios sin experiencia en programación.
- Solo Escritorio: Cellpose está diseñado para uso en escritorio; no hay aplicaciones nativas para Android o iOS.
Preguntas Frecuentes
No — Cellpose ofrece modelos generalistas preentrenados que suelen funcionar bien sin reentrenamiento. Sin embargo, para resultados óptimos en datos especiales o inusuales, puedes anotar y reentrenar usando las funciones de intervención humana.
Sí — soporta 3D reutilizando su modelo 2D (llamado "2.5D"), y puedes procesar pilas volumétricas mediante la GUI o API.
Se recomienda una GPU para inferencia y entrenamiento más rápidos, especialmente en conjuntos de datos grandes o 3D, pero Cellpose puede funcionar en máquinas solo con CPU con rendimiento más lento.
En la GUI, establece manualmente el diámetro celular estimado o deja que Cellpose lo calibre automáticamente. Puedes refinar resultados y reentrenar si la segmentación no es óptima.
Sí — las versiones más recientes (Cellpose 3) incluyen modelos de restauración de imágenes para reducción de ruido, desenfoque y aumento de resolución para mejorar la calidad de segmentación antes del procesamiento.
StarDist
| Desarrolladores | Uwe Schmidt, Martin Weigert, Coleman Broaddus y Gene Myers |
| Plataformas Soportadas |
|
| Soporte de Idioma | Proyecto de código abierto con documentación y comunidad principalmente en inglés |
| Modelo de Precio | Gratis y de código abierto. Licenciado bajo BSD-3-Clause |
Resumen
StarDist es una herramienta de aprendizaje profundo para la segmentación de instancias en imágenes de microscopía. Representa cada objeto (como núcleos celulares) como un polígono estrellado convexo en 2D o un poliedro en 3D, permitiendo la detección y separación precisa de objetos densamente agrupados o superpuestos. Con su arquitectura robusta, StarDist es ampliamente utilizado para la segmentación automática de células y núcleos en microscopía de fluorescencia, histopatología y otras aplicaciones de análisis bioimagen.
Características Clave
Segmentación de instancias altamente precisa usando polígonos estrellados convexos (2D) y poliedros (3D) para una detección confiable de objetos.
Modelos dedicados para imágenes 2D y datos volumétricos 3D para un análisis microscópico integral.
Modelos listos para usar para núcleos fluorescentes, histología teñida con H&E y otros escenarios comunes de imagen.
Clasifica los objetos detectados en clases distintas (por ejemplo, diferentes tipos celulares) en una sola ejecución de segmentación.
Integración fluida con ImageJ/Fiji, QuPath y napari para flujos de trabajo accesibles basados en GUI.
Evaluación completa de segmentación de instancias incluyendo precisión, recall, puntuación F1 y calidad panóptica.
Antecedentes Técnicos
Introducido originalmente en un artículo MICCAI 2018, la innovación central de StarDist es la predicción de distancias radiales a lo largo de rayos fijos combinada con la probabilidad de objeto para cada píxel, lo que permite la reconstrucción precisa de formas estrelladas convexas. Este enfoque segmenta de manera confiable objetos muy cercanos que son difíciles de separar usando métodos tradicionales basados en píxeles o cajas delimitadoras.
Desarrollos recientes han ampliado StarDist a imágenes de histopatología, permitiendo no solo la segmentación de núcleos sino también la clasificación multiclase de objetos detectados. El método alcanzó un rendimiento destacado en desafíos como CoNIC (Identificación y Conteo de Núcleos de Colon).
Descargar o Acceder
Instalación y Configuración
Instale TensorFlow (versión 1.x o 2.x) como requisito previo para StarDist.
Use pip para instalar el paquete Python de StarDist:
pip install stardistPara napari:
pip install stardist-napariPara QuPath: Instale la extensión StarDist arrastrando el archivo .jar dentro de QuPath.
Para ImageJ/Fiji: Use el gestor de plugins incorporado o la instalación manual desde el menú de plugins.
Ejecutar Segmentación
Cargue un modelo preentrenado, normalice su imagen y ejecute la predicción:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Abra su imagen en napari, seleccione el plugin StarDist, elija un modelo preentrenado o personalizado y ejecute la predicción directamente desde la GUI.
Use el plugin StarDist desde el menú Plugins para aplicar un modelo a su pila de imágenes con una interfaz intuitiva.
Después de instalar la extensión, ejecute la detección StarDist mediante la consola de scripts o la interfaz gráfica de QuPath para análisis histopatológico.
Entrenamiento y Ajuste Fino
Genere imágenes de etiquetas de verdad de terreno donde cada objeto esté etiquetado de forma única. Use herramientas de anotación como LabKit, QuPath o Fiji para preparar su conjunto de datos.
Use la API de Python de StarDist para entrenar un modelo nuevo o ajustar uno existente con sus datos anotados personalizados.
Opciones de Post-Procesamiento
- Aplique supresión de no máximos (NMS) para eliminar formas candidatas redundantes
- Use StarDist OPP (Post-Procesamiento de Objetos) para fusionar máscaras en formas no estrelladas convexas
Limitaciones y Consideraciones
- La suposición estrellada convexa puede no modelar perfectamente formas muy no convexas o irregulares
- Complejidad de instalación: instalaciones personalizadas requieren un compilador C++ compatible para construir extensiones
- La aceleración por GPU depende de versiones compatibles de TensorFlow, CUDA y cuDNN
- Algunos usuarios reportan problemas al ejecutar el plugin de ImageJ debido a la configuración de Java
Preguntas Frecuentes
StarDist funciona con una variedad de tipos de imagen incluyendo fluorescencia, campo claro e histopatología (por ejemplo, H&E), gracias a sus modelos preentrenados flexibles y su adaptabilidad a diferentes modalidades de imagen.
Sí — StarDist soporta segmentación de instancias 3D usando poliedros estrellados convexos para datos volumétricos, extendiendo las capacidades 2D al análisis completo en 3D.
No necesariamente. Hay modelos preentrenados disponibles que suelen funcionar bien desde el inicio. Sin embargo, para datos especializados o novedosos, anotar y entrenar modelos personalizados mejora significativamente la precisión.
StarDist se integra con napari, ImageJ/Fiji y QuPath, permitiendo ejecutar segmentación desde una GUI sin necesidad de programar. También soporta uso directo de la API Python para flujos avanzados.
StarDist ofrece funciones incorporadas para calcular métricas comunes de segmentación de instancias, incluyendo precisión, recall, puntuación F1 y calidad panóptica para evaluar el rendimiento de la segmentación.
SAM
Información de la Aplicación
| Desarrollador | Meta AI Research (FAIR) |
| Dispositivos Compatibles |
|
| Idioma y Disponibilidad | Modelo base de código abierto disponible globalmente; documentación en inglés |
| Precio | Gratis — código abierto bajo licencia de Meta vía GitHub e integración en MIB |
Visión General
SAM (Segment Anything Model) es un potente modelo base de IA creado por Meta que permite la segmentación interactiva y automática de prácticamente cualquier objeto en imágenes. Usando indicaciones como puntos, cuadros delimitadores o máscaras aproximadas, SAM genera máscaras de segmentación sin requerir reentrenamiento específico para la tarea. En la investigación en microscopía, la flexibilidad de SAM ha sido adaptada para segmentación celular, detección de orgánulos y análisis histopatológico, ofreciendo una solución escalable para investigadores que necesitan una herramienta de segmentación general y basada en indicaciones.
Introducción Detallada
Originalmente entrenado por Meta con más de 1 mil millones de máscaras en 11 millones de imágenes, SAM fue diseñado como un modelo base basado en indicaciones para segmentación con rendimiento "zero-shot" en dominios nuevos. En investigación de imágenes médicas, SAM ha sido evaluado para segmentación de patología en portaobjetos completos, detección de tumores e identificación de núcleos celulares. Sin embargo, su rendimiento en instancias densamente agrupadas —como núcleos celulares— es variable: incluso con indicaciones extensas (por ejemplo, 20 clics o cuadros), la segmentación zero-shot puede tener dificultades en imágenes microscópicas complejas.
Para abordar esta limitación, han surgido adaptaciones específicas del dominio:
- SAMCell — Ajustado finamente en grandes conjuntos de datos de microscopía para segmentación zero-shot robusta en diversos tipos celulares sin reentrenamiento por experimento
- μSAM — Reentrenado con más de 17,000 imágenes de microscopía anotadas manualmente para mejorar la precisión en estructuras celulares pequeñas
Características Clave
Interacción flexible usando puntos, cuadros y máscaras para control preciso.
Realiza segmentación sin ajuste fino en nuevos dominios de imágenes.
Adaptable para microscopía e histopatología mediante reentrenamiento few-shot o basado en indicaciones.
Disponible en Microscopy Image Browser (MIB) con soporte para segmentación 3D e interpolada.
IDCC-SAM permite conteo automático de células en inmunocitoquímica sin anotación manual.
Descargar o Acceder
Guía del Usuario
- Abra Microscopy Image Browser y navegue al panel de segmentación SAM
- Configure el intérprete de Python y seleccione entre los modelos SAM-1 o SAM-2
- Para aceleración por GPU, seleccione "cuda" en el entorno de ejecución (recomendado para rendimiento óptimo)
- Indicaciones por puntos: Haga clic en un objeto para definir una semilla positiva; use Shift + clic para expandir y Ctrl + clic para semillas negativas
- Pilas 3D: Use el modo interactivo 3D—haga clic en una rebanada, desplácese con shift y realice interpolación de semillas entre rebanadas
- Modo de ajuste: Reemplace, agregue, reste máscaras o cree una nueva capa según sea necesario
- Use la opción "Automatic everything" de MIB en el panel SAM-2 para segmentar todos los objetos visibles en una región
- Revise y refine las máscaras después de la segmentación según sea necesario
- Use pipelines de ajuste fino basados en indicaciones (por ejemplo, "All-in-SAM") para generar anotaciones a nivel de píxel a partir de indicaciones de usuario dispersas
- Para conteo celular, aplique IDCC-SAM, que usa SAM en un pipeline zero-shot con posprocesamiento
- Para segmentación celular de alta precisión, use SAMCell, ajustado finamente en imágenes de células de microscopía
Limitaciones y Consideraciones
- Rendimiento zero-shot inconsistente en estructuras densas o superpuestas sin ajuste de dominio
- La calidad de la segmentación depende mucho del diseño y estrategia de indicaciones
- Se recomienda encarecidamente GPU; la inferencia en CPU es muy lenta
- Dificultades con imágenes de portaobjetos completos de muy alta resolución y estructuras tisulares multiescala
- El ajuste fino o adaptación de SAM para microscopía puede requerir conocimientos en aprendizaje automático
Preguntas Frecuentes
Sí—mediante adaptaciones como SAMCell, que ajusta finamente SAM en conjuntos de datos de microscopía específicamente para tareas de segmentación celular.
No siempre. Con IDCC-SAM, puede realizar conteo celular zero-shot sin anotaciones manuales.
Use ajuste fino basado en indicaciones (por ejemplo, "All-in-SAM") o versiones preentrenadas para microscopía como μSAM, entrenado con más de 17,000 imágenes de microscopía anotadas.
Aunque es posible en CPU, se recomienda encarecidamente GPU para velocidad de inferencia práctica y segmentación interactiva en tiempo real.
Sí—la integración SAM-2 de MIB soporta segmentación 3D con interpolación de semillas entre rebanadas para análisis volumétrico.
AxonDeepSeg
| Desarrollador | Laboratorio NeuroPoly en Polytechnique Montréal y Université de Montréal |
| Plataformas compatibles |
|
| Idioma | Documentación en inglés; herramienta de código abierto usada globalmente |
| Precio | Gratis y de código abierto |
Resumen
AxonDeepSeg es una herramienta impulsada por IA para la segmentación automática de axones y mielina en imágenes de microscopía. Utilizando redes neuronales convolucionales, ofrece una segmentación precisa de tres clases (axón, mielina, fondo) en múltiples modalidades de imagen, incluyendo TEM, SEM y microscopía de campo claro. Al automatizar mediciones morfométricas como diámetro del axón, relación g y grosor de la mielina, AxonDeepSeg agiliza el análisis cuantitativo en la investigación en neurociencia, reduciendo significativamente el tiempo de anotación manual y mejorando la reproducibilidad.
Características clave
Modelos listos para usar optimizados para modalidades de microscopía TEM, SEM y campo claro.
Clasificación precisa de regiones de axón, mielina y fondo en imágenes de microscopía.
Cálculo automático de diámetro del axón, relación g, grosor de mielina y métricas de densidad.
La integración con la interfaz Napari permite el refinamiento manual de máscaras de segmentación para mayor precisión.
Se integra fácilmente en flujos de trabajo personalizados para análisis a gran escala de tejido neural.
Scripts de prueba completos garantizan reproducibilidad y resultados confiables de segmentación.
Detalles técnicos
Desarrollado por el Laboratorio NeuroPoly, AxonDeepSeg aprovecha el aprendizaje profundo para ofrecer segmentación de alta precisión en aplicaciones neurocientíficas. Se disponen de modelos preentrenados para diferentes modalidades de microscopía, asegurando versatilidad en técnicas de imagen. La herramienta se integra con Napari, permitiendo correcciones interactivas de máscaras de segmentación, lo que mejora la precisión en conjuntos de datos complejos. AxonDeepSeg calcula métricas morfométricas clave, apoyando estudios de alto rendimiento sobre la estructura y patología del tejido neural. Su framework basado en Python facilita la integración en flujos personalizados para análisis a gran escala de la morfología de axones y mielina.
Descargar o acceder
Instalación y configuración
Asegúrese de tener Python 3.8 o superior instalado, luego instale AxonDeepSeg y Napari usando pip:
pip install axondeepseg napariEjecute los scripts de prueba proporcionados para confirmar que todos los componentes están correctamente instalados y funcionando.
Importe imágenes de microscopía (TEM, SEM o campo claro) en Napari o en su entorno Python.
Elija el modelo preentrenado adecuado para su modalidad de imagen y ejecute la segmentación para generar máscaras de axón y mielina.
Calcule automáticamente mediciones morfométricas como diámetro del axón, relación g, densidad y grosor de mielina, luego exporte los resultados en formato CSV.
Use la interfaz Napari para ajustar manualmente las máscaras de segmentación donde sea necesario, fusionando o eliminando máscaras para mejorar la precisión.
Consideraciones importantes
- El rendimiento puede disminuir en modalidades de imagen novedosas o no entrenadas
- Puede ser necesaria la corrección manual en regiones complejas o difíciles
- Se recomienda GPU para procesamiento más rápido de grandes conjuntos de datos; también se soporta CPU
Preguntas frecuentes
AxonDeepSeg soporta TEM (Microscopía Electrónica de Transmisión), SEM (Microscopía Electrónica de Barrido) y microscopía de campo claro con modelos preentrenados optimizados para cada modalidad.
Sí, AxonDeepSeg es completamente gratuito y de código abierto, disponible para uso académico y comercial.
Sí, AxonDeepSeg calcula automáticamente el diámetro del axón, relación g, grosor de mielina y métricas de densidad a partir de imágenes segmentadas.
Se recomienda GPU para una segmentación más rápida de grandes conjuntos de datos, pero también se soporta procesamiento en CPU para análisis más pequeños.
Sí, la integración con la interfaz Napari permite correcciones interactivas y refinamiento de máscaras de segmentación para mayor precisión en regiones difíciles.
Ilastik
| Desarrollador | Equipo Ilastik del Laboratorio Europeo de Biología Molecular (EMBL) y socios académicos asociados |
| Plataformas compatibles |
|
| Idioma | Inglés |
| Precio | Gratis y de código abierto |
Resumen
Ilastik es una herramienta potente impulsada por IA para la segmentación interactiva, clasificación y análisis de imágenes de microscopía. Usando técnicas de aprendizaje automático como clasificadores Random Forest, permite a los investigadores segmentar píxeles, clasificar objetos, seguir células a lo largo del tiempo y realizar conteo de densidad en conjuntos de datos 2D y 3D. Con su interfaz intuitiva y retroalimentación en tiempo real, Ilastik es accesible para científicos sin experiencia en programación y es ampliamente adoptado en biología celular, neurociencia e imagen biomédica.
Características clave
Retroalimentación en tiempo real mientras anotas regiones representativas para obtener resultados instantáneos de segmentación.
Categoriza estructuras segmentadas según características morfológicas e intensidad.
Sigue el movimiento y la división celular en experimentos de microscopía de lapso de tiempo en 2D y 3D.
Cuantifica regiones densamente pobladas sin segmentación explícita de objetos individuales.
Segmentación semiautomática para volúmenes 3D complejos con interacción intuitiva.
Procesa múltiples imágenes automáticamente usando modo sin interfaz gráfica (headless).
Descarga
Guía para comenzar
Descarga Ilastik para tu sistema operativo desde el sitio web oficial. El paquete incluye todas las dependencias de Python necesarias, así que sigue las instrucciones de instalación para tu plataforma.
Abre Ilastik y elige tu flujo de trabajo de análisis: Clasificación de píxeles, Clasificación de objetos, Seguimiento o Conteo de densidad. Carga tu conjunto de imágenes, que puede incluir imágenes multicanal, 3D o de lapso de tiempo.
Etiqueta algunos píxeles u objetos representativos en tus imágenes. El clasificador Random Forest de Ilastik aprende de estas anotaciones y predice automáticamente etiquetas en todo tu conjunto de datos.
Aplica el modelo entrenado para segmentar o clasificar todo tu conjunto de datos. Exporta los resultados como imágenes etiquetadas, mapas de probabilidad o tablas cuantitativas para análisis y visualización posteriores.
Usa el modo sin interfaz gráfica de Ilastik para procesar automáticamente múltiples imágenes sin intervención manual, ideal para pipelines de análisis a gran escala.
Limitaciones y consideraciones
- La etiquetación interactiva puede ser lenta para conjuntos de datos muy grandes
- La precisión depende de la calidad y representatividad de las anotaciones del usuario
- Requisitos de memoria — conjuntos de datos de muy alta resolución o de varios gigabytes pueden requerir mucha RAM
- Datos complejos — los clasificadores Random Forest pueden rendir menos que redes neuronales profundas en datos de imagen altamente variables o complejos
Preguntas frecuentes
Sí, Ilastik soporta completamente volúmenes 3D y experimentos de lapso de tiempo para segmentación, seguimiento y análisis cuantitativo a través de múltiples puntos temporales.
Sí, Ilastik es completamente gratuito y de código abierto, disponible para todos los usuarios sin restricciones de licencia.
No, Ilastik ofrece una interfaz gráfica intuitiva con retroalimentación en tiempo real, haciéndolo accesible para investigadores sin experiencia en programación. Los usuarios avanzados también pueden usar procesamiento por lotes desde línea de comandos.
Sí, el flujo de trabajo dedicado al seguimiento permite analizar el movimiento y la división celular en conjuntos de datos de lapso de tiempo en 2D y 3D con seguimiento automático de linajes.
Los resultados de segmentación pueden exportarse como imágenes etiquetadas, mapas de probabilidad o tablas cuantitativas, permitiendo una integración fluida con herramientas de análisis y software de visualización posteriores.
Estas herramientas abarcan desde niveles novatos hasta expertos. Muchas son gratuitas y de código abierto, facilitando flujos de trabajo de IA reproducibles y compartibles en la comunidad investigadora.
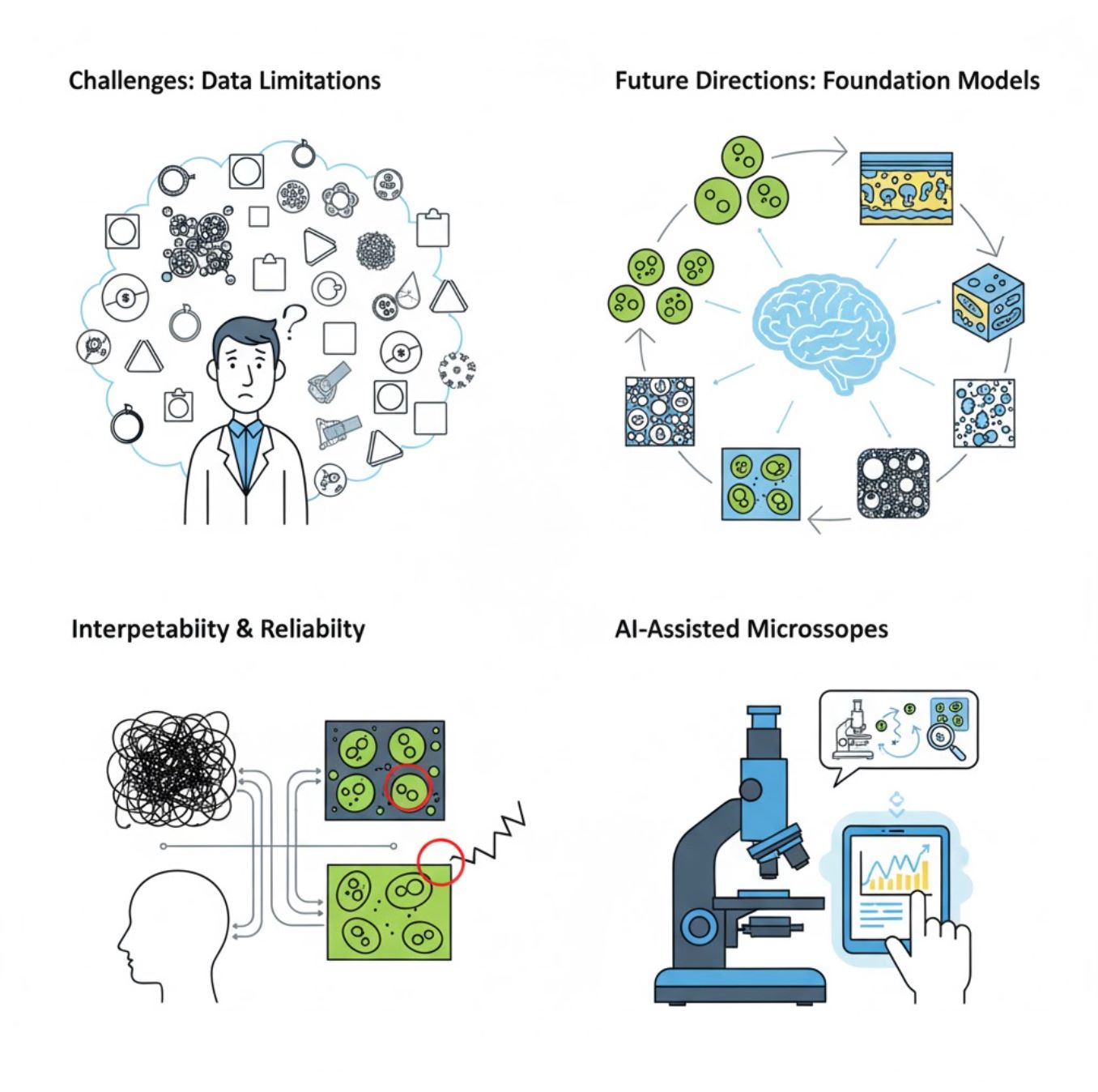
Desafíos y Direcciones Futuras
Desafíos Actuales
Tendencias Emergentes
Modelos Base de Visión
Los sistemas de IA de próxima generación prometen reducir la necesidad de entrenamiento específico para cada tarea.
- Modelos como SAM y sistemas basados en CLIP
- Una IA que maneja muchas tareas de microscopía
- Despliegue y adaptación más rápidos
Microscopios Asistidos por IA
Sistemas de microscopía totalmente autónomos e inteligentes están convirtiéndose en realidad.
- Control por lenguaje natural mediante LLMs
- Bucle de retroalimentación totalmente automatizado
- Democratiza el acceso a microscopía avanzada
Conclusiones Clave
- La IA está transformando rápidamente el procesamiento de imágenes de microscopio con mayor precisión y automatización
- El aprendizaje profundo supera al aprendizaje automático tradicional en imágenes de microscopía complejas y variables
- Las CNN aprenden automáticamente características jerárquicas desde píxeles en bruto para un análisis robusto
- Las aplicaciones clave incluyen segmentación, clasificación, seguimiento, reducción de ruido y adquisición automatizada
- El éxito depende de datos de calidad y validación cuidadosa por expertos
- Los modelos base de visión y los microscopios asistidos por IA representan el futuro del campo
Con avances continuos y esfuerzos comunitarios (herramientas de código abierto, conjuntos de datos compartidos), la IA será cada vez más parte esencial del "ojo" del microscopio, ayudando a los científicos a ver lo invisible.







No comments yet. Be the first to comment!