Штучний інтелект у обробці зображень мікроскопа
Штучний інтелект революціонізує обробку зображень мікроскопа завдяки потужним можливостям, таким як точна сегментація, зниження шуму, суперроздільна здатність та автоматизоване отримання зображень. У цій статті висвітлено основні інструменти ШІ та нові тенденції у наукових дослідженнях.
Методи штучного інтелекту революціонізують мікроскопію, оптимізуючи отримання зображень та автоматизуючи аналіз. У сучасних розумних мікроскопах модулі ШІ можуть у реальному часі налаштовувати параметри зйомки (наприклад, фокус, освітлення), щоб мінімізувати фотоблідинг і покращити сигнал. Тим часом алгоритми глибокого навчання можуть аналізувати складні дані зображень, щоб виявляти приховані біологічні закономірності та навіть пов’язувати зображення з іншими даними (наприклад, геномікою).
Методи ШІ: машинне навчання проти глибокого навчання
Методи ШІ варіюються від класичного машинного навчання (ML) до сучасного глибокого навчання (DL). Кожен підхід має свої сильні та слабкі сторони:
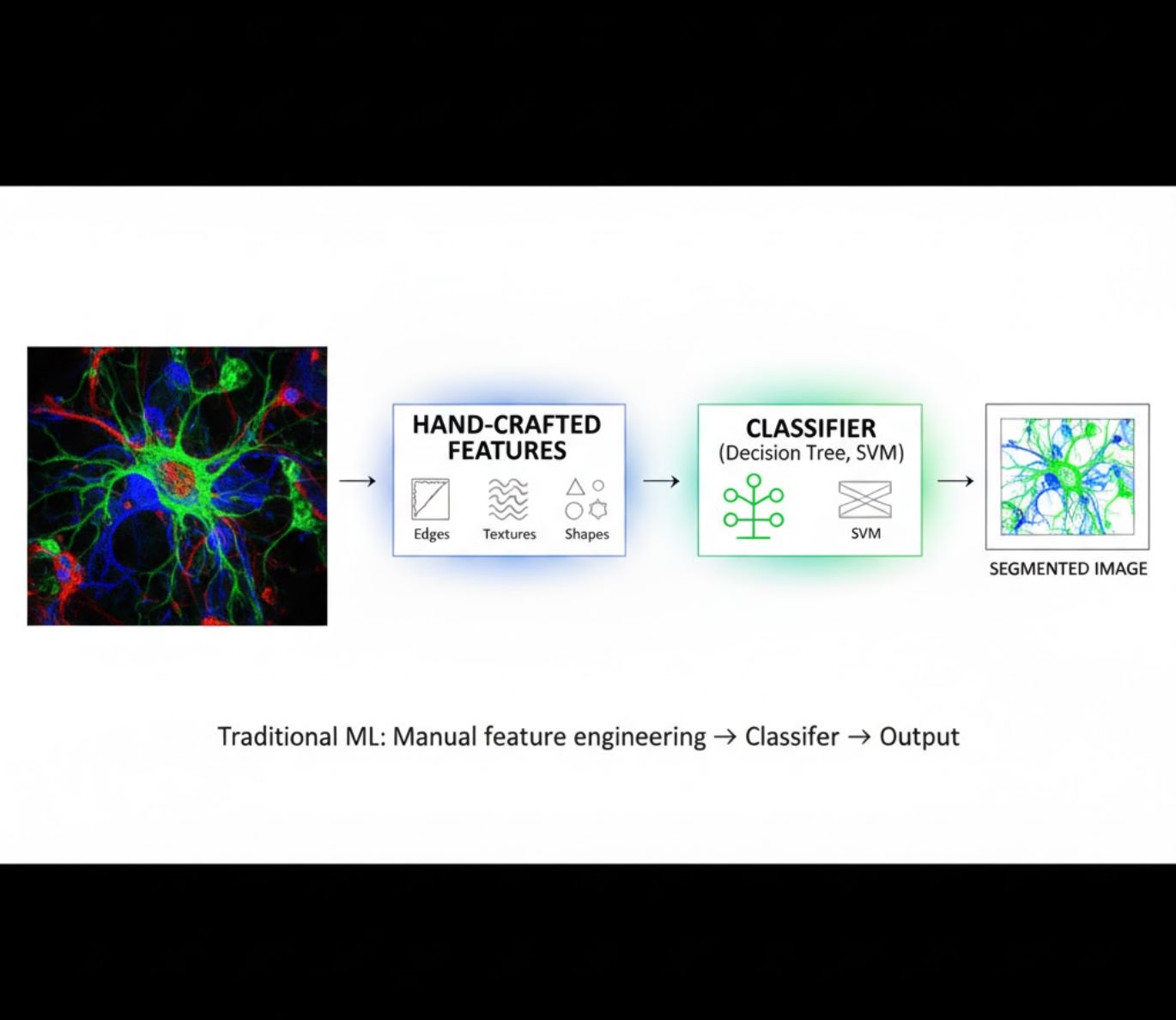
Ручне створення ознак
- Дослідники вручну створюють ознаки зображень (краї, текстури, форми)
- Ознаки подаються на класифікатори (дерева рішень, SVM)
- Швидке навчання
- Проблеми з обробкою складних або зашумлених зображень
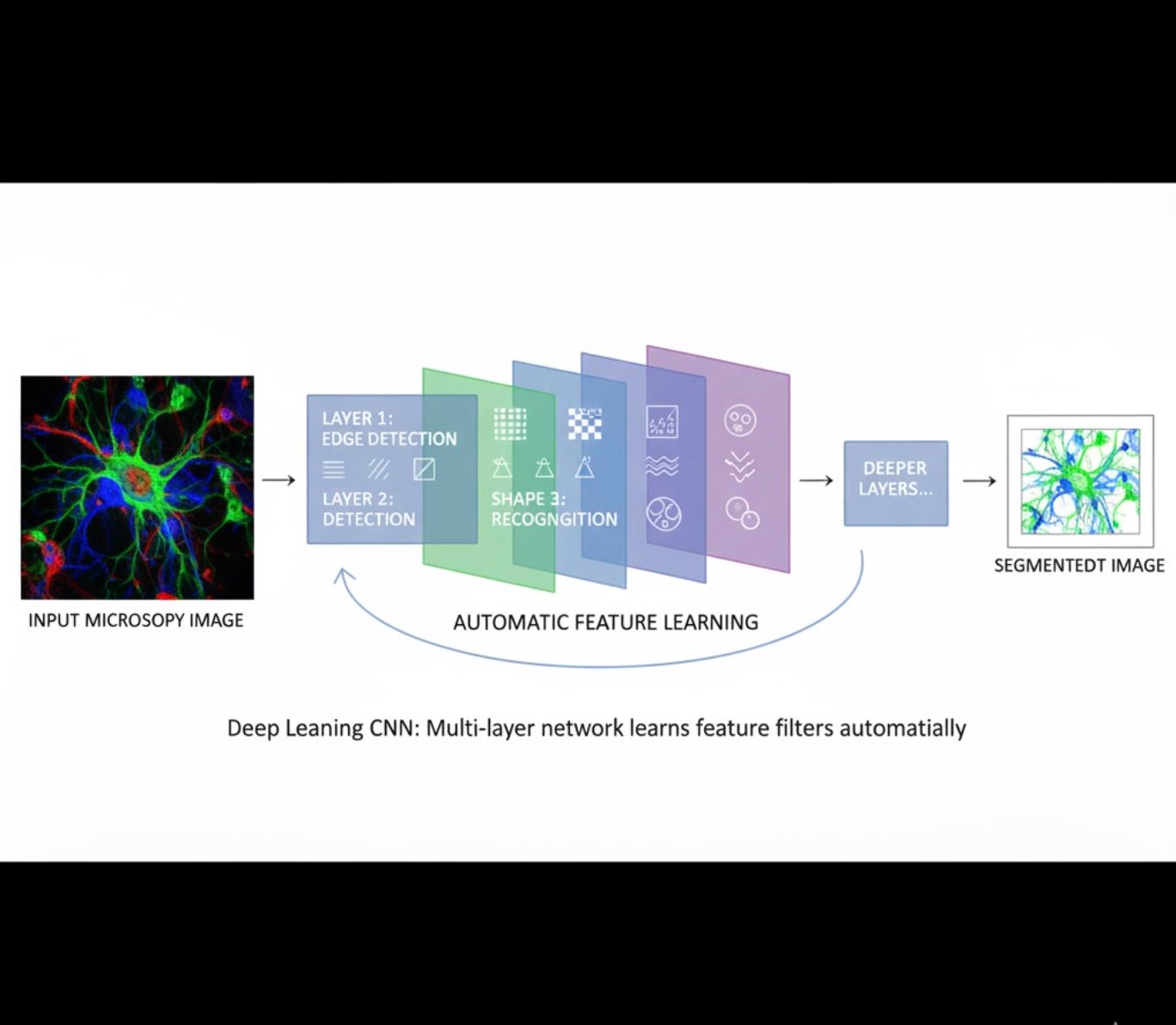
Автоматичне навчання ознак
- Багатошарові нейронні мережі (CNN) автоматично вивчають ознаки
- Навчання від сирих пікселів до кінцевого результату
- Набагато стійкіше до варіацій
- Надійно захоплює складні текстури та структури
Як працюють CNN: Згорткові нейронні мережі застосовують послідовні фільтри до мікроскопічних зображень, навчаючись виявляти прості патерни (краї) на ранніх шарах і складні структури (форми клітин, текстури) на глибших. Така ієрархічна модель робить глибоке навчання надзвичайно стійким навіть при значних варіаціях інтенсивності.
Візуальне порівняння: ML проти DL конвеєрів
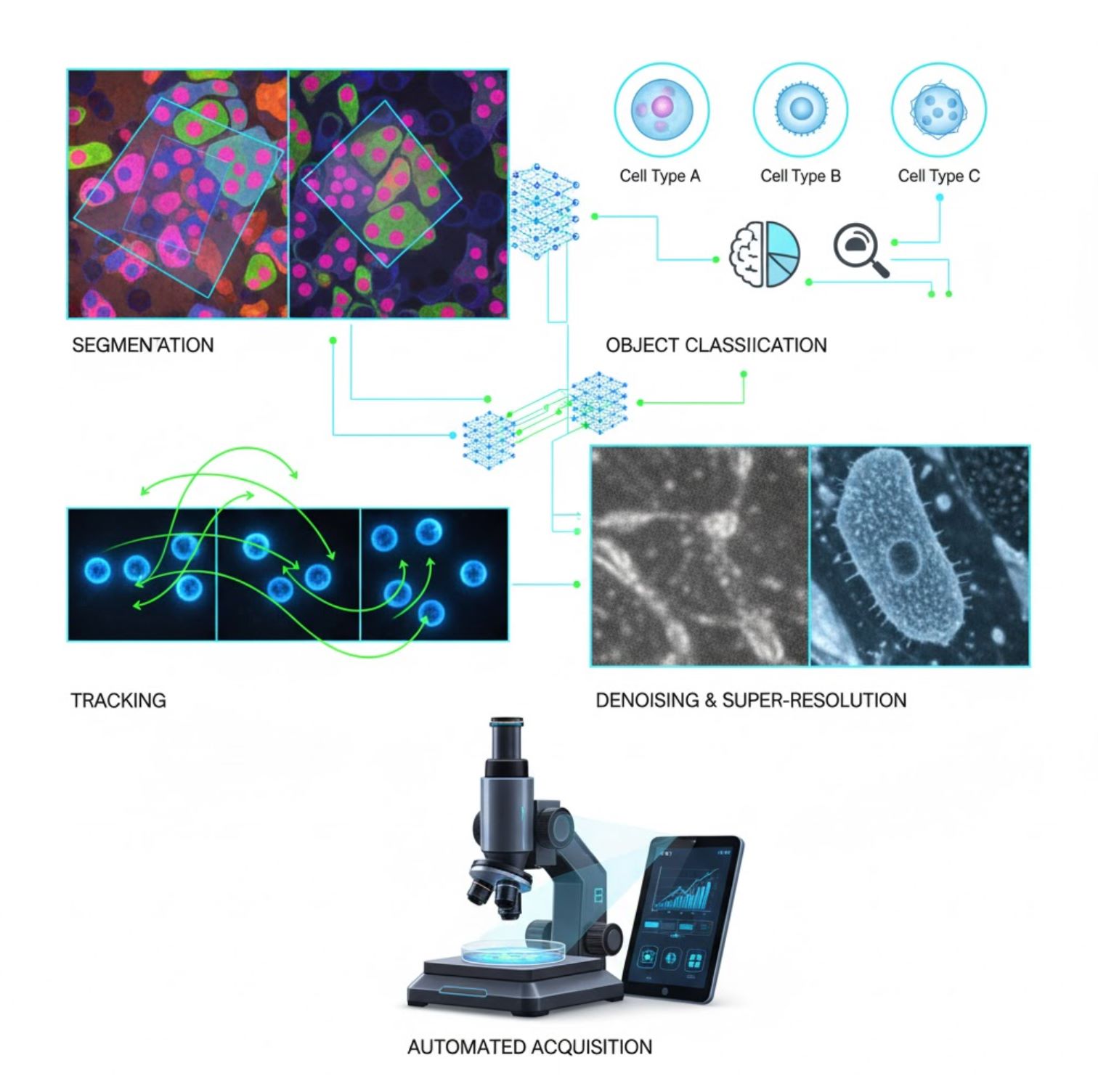
Ключові застосування ШІ в мікроскопії
ШІ вже інтегровано у багато завдань обробки зображень у робочому процесі мікроскопії:
Сегментація
Розподіл зображень на області (наприклад, ідентифікація кожної клітини або ядра). Глибокі мережі, такі як U-Net, відмінно справляються з цим завданням.
- Семантична сегментація: позначення класів для кожного пікселя
- Інстанс-сегментація: розділення окремих об’єктів
- Висока точність на густих або тьмяних зображеннях
- Фундаційні моделі зору (наприклад, μSAM) тепер адаптовані для мікроскопії
Класифікація об’єктів
Після сегментації ШІ класифікує кожен об’єкт із високою точністю.
- Ідентифікація типу клітин
- Визначення стадії мітозу
- Виявлення патологічних індикаторів
- Розрізняє тонкі фенотипи, які важко кількісно оцінити вручну
Відстеження
У часовій мікроскопії ШІ відстежує клітини або частинки між кадрами з безпрецедентною точністю.
- Глибоке навчання значно покращує точність відстеження
- Дозволяє надійно аналізувати рухомі клітини
- Фіксує динамічні біологічні процеси
Зниження шуму та суперроздільна здатність
Моделі ШІ покращують якість зображень, усуваючи шум і розмиття.
- Глибокі моделі з урахуванням фізики вивчають оптику мікроскопа
- Відновлюють чіткіші, безартефактні зображення
- Вища роздільна здатність із меншими артефактами порівняно з традиційними методами
Автоматизоване отримання зображень
ШІ керує самим мікроскопом у реальному часі.
- Аналізує живі зображення для прийняття розумних рішень
- Автоматично налаштовує фокус і сканує цікаві області
- Знижує фототоксичність і економить час
- Забезпечує високопродуктивні та адаптивні експерименти з візуалізацією
Популярні інструменти ШІ для обробки зображень мікроскопа
Багата екосистема інструментів підтримує штучний інтелект у мікроскопії. Дослідники створили як універсальні, так і спеціалізовані програми, багато з яких є відкритим кодом:
Cellpose
| Розробники | Карсен Стрінгер та Маріус Пачитаріу (дослідницька група MouseLand) |
| Підтримувані платформи |
Потрібен Python (встановлення через pip/conda). GUI доступний лише на настільних ПК. |
| Підтримка мов | Документація англійською; широко використовується в дослідницьких лабораторіях по всьому світу |
| Модель ліцензування | Безкоштовна та з відкритим кодом під ліцензією BSD-3-Clause |
Огляд
Cellpose — це сучасний інструмент сегментації на основі глибокого навчання, призначений для мікроскопічних зображень. Як універсальний алгоритм, він точно сегментує різноманітні типи клітин (ядра, цитоплазма тощо) у різних режимах візуалізації без необхідності перенавчання моделі. Завдяки можливості навчання з участю користувача дослідники можуть покращувати результати, адаптувати модель під свої дані та застосовувати систему як для 2D, так і для 3D робочих процесів.
Ключові особливості
Працює «з коробки» для широкого спектра типів клітин, барвників і методів візуалізації без потреби в індивідуальному навчанні.
Підтримує повні 3D стекові зображення за допомогою підходу «2.5D», що повторно використовує 2D моделі для об’ємних даних.
Ручне виправлення результатів сегментації та перенавчання моделі на власних даних для підвищення точності.
Доступ через Python API, командний рядок або графічний інтерфейс для гнучких робочих процесів.
Можливості шумозаглушення, розмиття та збільшення роздільної здатності для покращення якості зображень перед сегментацією.
Завантаження або доступ
Технічний опис
Cellpose було представлено у фундаментальному дослідженні Стрінгера, Ванга, Майклоса та Пачитаріу, навчено на великому та дуже різноманітному наборі даних, що містить понад 70 000 сегментованих об’єктів. Ця різноманітність дозволяє моделі узагальнюватися на різні форми, розміри клітин та умови мікроскопії, суттєво зменшуючи потребу в індивідуальному навчанні у більшості випадків. Для 3D даних Cellpose розумно повторно використовує свою 2D модель у режимі «2.5D», уникаючи необхідності повністю 3D-розмічених навчальних даних, при цьому забезпечуючи об’ємну сегментацію. Cellpose 2.0 представив навчання з участю користувача, що дозволяє вручну коригувати передбачення та перенавчати модель на власних зображеннях для покращення продуктивності на конкретних наборах даних.
Встановлення та налаштування
Налаштуйте середовище Python за допомогою conda:
conda create -n cellpose python=3.10Активуйте середовище та встановіть Cellpose:
# For GUI support
pip install cellpose[gui]
# For minimal setup (API/CLI only)
pip install cellposeПочаток роботи
Режим GUI
- Запустіть GUI, виконавши:
python -m cellpose - Перетягніть файли зображень (
.tif,.pngтощо) у інтерфейс - Виберіть тип моделі (наприклад, «cyto» для цитоплазми або «nuclei» для ядер)
- Встановіть приблизний діаметр клітин або дозвольте Cellpose автоматично калібрувати
- Натисніть, щоб почати сегментацію та переглянути результати
Режим Python API
from cellpose import models
# Load model
model = models.Cellpose(model_type='cyto')
# Segment images
masks, flows = model.eval(images, diameter=30)Уточнення та перенавчання
- Після створення масок вручну виправте сегментацію в GUI, об’єднуючи або видаляючи маски
- Використовуйте вбудовані функції навчання для перенавчання на виправлених прикладах
- Покращена продуктивність моделі на вашому конкретному наборі даних
Обробка 3D даних
- Завантажте багатошаровий TIFF або об’ємний стек
- Використовуйте прапорець
--Zstackу GUI або API для обробки як 3D - За бажанням уточнюйте 3D потоки за допомогою згладжування або спеціалізованих параметрів для кращої сегментації
Обмеження та зауваження
- Компроміс універсальності моделі: Хоча універсальна модель працює широко, дуже незвичні форми клітин або умови візуалізації можуть вимагати перенавчання.
- Зусилля з анотації: Навчання з участю користувача вимагає ручних виправлень, що може бути трудомістким для великих наборів даних.
- Складність встановлення: Встановлення GUI може вимагати використання командного рядка, середовищ conda та управління залежностями Python — не завжди просто для непрофесіоналів.
- Лише для настільних ПК: Cellpose розроблено для використання на настільних комп’ютерах; немає рідних додатків для Android або iOS.
Часті запитання
Ні — Cellpose надає попередньо навчені універсальні моделі, які часто добре працюють без перенавчання. Однак для оптимальних результатів на спеціальних або незвичних даних ви можете анотувати та перенавчати модель за допомогою функцій навчання з участю користувача.
Так — він підтримує 3D, повторно використовуючи свою 2D модель (так званий «2.5D»), і ви можете обробляти об’ємні стеки через GUI або API.
GPU настійно рекомендується для швидшої обробки та навчання, особливо на великих або 3D наборах даних, але Cellpose може працювати і на машинах лише з CPU з повільнішою продуктивністю.
У GUI встановіть приблизний діаметр клітин вручну або дозвольте Cellpose автоматично його калібрувати. Ви можете уточнювати результати та перенавчати модель, якщо сегментація не є оптимальною.
Так — новіші версії (Cellpose 3) включають моделі відновлення зображень для шумозаглушення, розмиття та збільшення роздільної здатності, щоб покращити якість сегментації перед обробкою.
StarDist
| Розробники | Уве Шмідт, Мартін Вайгерт, Коулмен Броддус та Джин Майєрс |
| Підтримувані платформи |
|
| Підтримка мов | Проєкт з відкритим кодом з документацією та спільнотою переважно англійською мовою |
| Модель ліцензування | Безкоштовно та з відкритим кодом. Ліцензія BSD-3-Clause |
Огляд
StarDist — це інструмент глибокого навчання для сегментації окремих об’єктів на мікроскопічних зображеннях. Він представляє кожен об’єкт (наприклад, ядра клітин) як зіркоподібний опуклий багатокутник у 2D або багатогранник у 3D, що дозволяє точно виявляти та розділяти щільно розташовані або перекриваючіся об’єкти. Завдяки надійній архітектурі StarDist широко використовується для автоматизованої сегментації клітин і ядер у флуоресцентній мікроскопії, гістопатології та інших застосуваннях біообробки зображень.
Ключові особливості
Високоточна сегментація окремих об’єктів за допомогою зіркоподібних опуклих багатокутників (2D) і багатогранників (3D) для надійного виявлення об’єктів.
Спеціалізовані моделі для 2D-зображень і 3D-об’ємних даних для комплексного аналізу мікроскопії.
Готові до використання моделі для флуоресцентних ядер, гістології з фарбуванням H&E та інших поширених сценаріїв візуалізації.
Класифікація виявлених об’єктів за різними класами (наприклад, різні типи клітин) в одному запуску сегментації.
Безшовна інтеграція з ImageJ/Fiji, QuPath та napari для зручних робочих процесів із графічним інтерфейсом.
Комплексна оцінка сегментації окремих об’єктів, включно з точністю, повнотою, F1-метрикою та паноптичною якістю.
Технічний фон
Спочатку представлений у статті MICCAI 2018 року, основною інновацією StarDist є прогнозування радіальних відстаней вздовж фіксованих променів у поєднанні з ймовірністю об’єкта для кожного пікселя, що дозволяє точно відтворювати зіркоподібні опуклі форми. Цей підхід надійно сегментує щільно розташовані об’єкти, які важко розділити традиційними методами на основі пікселів або обмежувальних рамок.
Останні розробки розширили StarDist на гістопатологічні зображення, що дозволяє не лише сегментацію ядер, а й багатокласову класифікацію виявлених об’єктів. Метод досяг найкращих результатів у змаганнях, таких як CoNIC (Colon Nuclei Identification and Counting).
Завантаження або доступ
Встановлення та налаштування
Встановіть TensorFlow (версії 1.x або 2.x) як передумову для StarDist.
Використайте pip для встановлення пакету StarDist для Python:
pip install stardistДля napari:
pip install stardist-napariДля QuPath: Встановіть розширення StarDist, перетягнувши файл .jar у QuPath.
Для ImageJ/Fiji: Використовуйте вбудований менеджер плагінів або встановлення вручну через меню плагінів.
Запуск сегментації
Завантажте попередньо навчену модель, нормалізуйте зображення та виконайте прогноз:
from stardist.models import StarDist2D
model = StarDist2D.from_pretrained('2D_versatile_fluo')
labels, details = model.predict_instances(image)Відкрийте зображення в napari, оберіть плагін StarDist, виберіть попередньо навчену або власну модель і виконайте прогноз безпосередньо через GUI.
Використовуйте плагін StarDist у меню Плагіни для застосування моделі до стеку зображень через інтуїтивний інтерфейс.
Після встановлення розширення виконуйте виявлення StarDist через консоль скриптів або графічний інтерфейс QuPath для гістопатологічного аналізу.
Навчання та донавчання
Створіть еталонні зображення з унікальним маркуванням кожного об’єкта. Використовуйте інструменти анотації, такі як LabKit, QuPath або Fiji, для підготовки набору даних.
Використовуйте Python API StarDist для навчання нової моделі або донавчання існуючої на власних анотованих даних.
Варіанти постобробки
- Застосування придушення максимумів (NMS) для усунення зайвих кандидатів на форми
- Використання StarDist OPP (постобробка об’єктів) для об’єднання масок не зіркоподібних опуклих форм
Обмеження та зауваження
- Припущення зіркоподібної опуклості може не ідеально описувати дуже неопуклі або нерегулярні форми
- Складність встановлення: для кастомних інсталяцій потрібен сумісний компілятор C++ для збірки розширень
- Прискорення на GPU залежить від сумісних версій TensorFlow, CUDA та cuDNN
- Деякі користувачі повідомляють про проблеми з запуском плагіна ImageJ через налаштування Java
Часті запитання
StarDist працює з різними типами зображень, включно з флуоресцентними, світловими (brightfield) та гістопатологічними (наприклад, H&E), завдяки гнучким попередньо навченим моделям і адаптивності до різних методів візуалізації.
Так — StarDist підтримує 3D сегментацію окремих об’єктів за допомогою зіркоподібних опуклих багатогранників для об’ємних даних, розширюючи можливості 2D до повного 3D аналізу.
Не обов’язково. Попередньо навчені моделі доступні і часто добре працюють одразу. Однак для спеціалізованих або нових даних анотація та навчання власних моделей значно підвищує точність.
StarDist інтегрується з napari, ImageJ/Fiji та QuPath, що дозволяє запускати сегментацію через графічний інтерфейс без програмування. Також підтримується пряме використання Python API для складніших робочих процесів.
StarDist надає вбудовані функції для обчислення поширених метрик сегментації окремих об’єктів, включно з точністю, повнотою, F1-метрикою та паноптичною якістю для оцінки продуктивності сегментації.
SAM
Інформація про застосунок
| Розробник | Meta AI Research (FAIR) |
| Підтримувані пристрої |
|
| Мова та доступність | Відкрита базова модель, доступна глобально; документація англійською |
| Вартість | Безкоштовно — відкритий код за ліцензією Meta через GitHub та інтеграцію в MIB |
Загальний огляд
SAM (Segment Anything Model) — потужна базова модель штучного інтелекту, створена Meta, яка забезпечує інтерактивну та автоматичну сегментацію практично будь-якого об’єкта на зображеннях. Використовуючи підказки у вигляді точок, обмежувальних рамок або приблизних масок, SAM генерує сегментаційні маски без необхідності донавчання для конкретних завдань. У мікроскопічних дослідженнях гнучкість SAM адаптована для сегментації клітин, виявлення органел та аналізу гістопатології, пропонуючи масштабоване рішення для дослідників, які потребують універсального інструменту сегментації з підтримкою підказок.
Детальний вступ
Спочатку навчена Meta на понад 1 мільярді масок на 11 мільйонах зображень, SAM була розроблена як базова модель сегментації з підтримкою підказок і «zero-shot» продуктивністю на нових доменах. У медичних дослідженнях SAM оцінювали для сегментації цілих слайдів патології, виявлення пухлин і ідентифікації ядер клітин. Однак її продуктивність на щільно розташованих об’єктах — таких як ядра клітин — є неоднозначною: навіть при великій кількості підказок (наприклад, 20 кліків або рамок) сегментація в режимі zero-shot може бути складною на складних мікроскопічних зображеннях.
Для подолання цього обмеження з’явилися доменно-специфічні адаптації:
- SAMCell — донавчена на великих мікроскопічних наборах даних для сильної zero-shot сегментації різних типів клітин без необхідності донавчання для кожного експерименту
- μSAM — донавчена на понад 17 000 вручну анотованих мікроскопічних зображень для підвищення точності на дрібних клітинних структурах
Ключові особливості
Гнучка взаємодія за допомогою точок, рамок і масок для точного контролю.
Виконує сегментацію без донавчання на нових доменах зображень.
Адаптується для мікроскопії та гістопатології через few-shot або підказкове донавчання.
Доступна в Microscopy Image Browser (MIB) з підтримкою 3D та інтерпольованої сегментації.
IDCC-SAM забезпечує автоматичний підрахунок клітин в імуногістохімії без ручного анотування.
Завантаження або доступ
Керівництво користувача
- Відкрийте Microscopy Image Browser і перейдіть до панелі сегментації SAM
- Налаштуйте інтерпретатор Python і виберіть між моделями SAM-1 або SAM-2
- Для прискорення на GPU виберіть "cuda" у середовищі виконання (рекомендується для оптимальної продуктивності)
- Підказки точками: Клацніть на об’єкті, щоб визначити позитивне насіння; використовуйте Shift + клацання для розширення та Ctrl + клацання для негативних насінин
- 3D стекі: Використовуйте інтерактивний 3D режим — клацніть на одному зрізі, прокручуйте з Shift і інтерполюйте насіння між зрізами
- Режим корекції: Замінюйте, додавайте, віднімайте маски або створюйте новий шар за потреби
- Використовуйте опцію «Автоматично все» в панелі SAM-2 MIB для сегментації всіх видимих об’єктів у регіоні
- Переглядайте та коригуйте маски після сегментації за потреби
- Використовуйте підказкові конвеєри донавчання (наприклад, «All-in-SAM») для створення піксельних анотацій із розріджених підказок користувача
- Для підрахунку клітин застосовуйте IDCC-SAM, який використовує SAM у zero-shot конвеєрі з подальшою обробкою
- Для високоточних сегментацій клітин використовуйте SAMCell, донавчену на мікроскопічних зображеннях клітин
Обмеження та зауваження
- Zero-shot продуктивність нестабільна на щільних або накладених структурах без доменної адаптації
- Якість сегментації сильно залежить від дизайну та стратегії підказок
- Настійно рекомендується GPU; робота на CPU дуже повільна
- Має труднощі з дуже високою роздільною здатністю цілих слайдів і багатомасштабними тканинними структурами
- Донавчання або адаптація SAM для мікроскопії може вимагати знань у машинному навчанні
Часті запитання
Так — через адаптації, як-от SAMCell, яка донавчає SAM на мікроскопічних наборах даних спеціально для задач сегментації клітин.
Не завжди. За допомогою IDCC-SAM можна виконувати підрахунок клітин у режимі zero-shot без ручних анотацій.
Використовуйте підказкове донавчання (наприклад, «All-in-SAM») або попередньо навчені версії для мікроскопії, як-от μSAM, навчена на понад 17 000 анотованих мікроскопічних зображеннях.
Хоча можливо запускати на CPU, GPU настійно рекомендується для практичної швидкості інференсу та інтерактивної сегментації в реальному часі.
Так — інтеграція SAM-2 у MIB підтримує 3D сегментацію з інтерполяцією насінин між зрізами для об’ємного аналізу.
AxonDeepSeg
| Розробник | Лабораторія NeuroPoly при Політехнічній школі Монреаля та Університеті Монреаля |
| Підтримувані платформи |
|
| Мова | Документація англійською; інструмент з відкритим кодом, що використовується у всьому світі |
| Вартість | Безкоштовно та з відкритим кодом |
Огляд
AxonDeepSeg — це інструмент на базі штучного інтелекту для автоматичної сегментації аксонів і мієліну на мікроскопічних зображеннях. Використовуючи згорткові нейронні мережі, він забезпечує точну сегментацію з трьома класами (аксон, мієлін, фон) для різних методів візуалізації, включно з TEM, SEM та світловою мікроскопією. Автоматизація морфометричних вимірювань, таких як діаметр аксона, g-коефіцієнт і товщина мієліну, дозволяє AxonDeepSeg оптимізувати кількісний аналіз у нейронаукових дослідженнях, значно скорочуючи час ручної розмітки та підвищуючи відтворюваність.
Ключові особливості
Готові до використання моделі, оптимізовані для TEM, SEM та світлової мікроскопії.
Точна класифікація аксонів, мієліну та фону на мікроскопічних зображеннях.
Автоматичне обчислення діаметра аксона, g-коефіцієнта, товщини мієліну та щільності.
Інтеграція з Napari GUI дозволяє вручну уточнювати маски сегментації для підвищення точності.
Безшовна інтеграція у власні конвеєри для масштабного аналізу нервової тканини.
Комплексні тестові скрипти забезпечують відтворюваність і надійність результатів сегментації.
Технічні деталі
Розроблений лабораторією NeuroPoly, AxonDeepSeg використовує глибоке навчання для високоточних сегментацій у нейронаукових застосуваннях. Попередньо навчені моделі доступні для різних методів мікроскопії, що забезпечує універсальність для різних технік візуалізації. Інструмент інтегрується з Napari, що дозволяє інтерактивно коригувати маски сегментації, підвищуючи точність на складних наборах даних. AxonDeepSeg обчислює ключові морфометричні показники, підтримуючи високопродуктивні дослідження структури та патології нервової тканини. Його фреймворк на Python дозволяє інтегрувати інструмент у власні конвеєри для масштабного аналізу морфології аксонів і мієліну.
Завантаження або доступ
Встановлення та налаштування
Переконайтеся, що встановлено Python 3.8 або новішої версії, потім встановіть AxonDeepSeg і Napari за допомогою pip:
pip install axondeepseg napariЗапустіть надані тестові скрипти, щоб підтвердити правильність встановлення та функціонування всіх компонентів.
Імпортуйте мікроскопічні зображення (TEM, SEM або світлові) у Napari або у ваше Python-середовище.
Обирайте відповідну попередньо навчену модель для вашої модальності зображень і запускайте сегментацію для створення масок аксонів і мієліну.
Автоматично обчислюйте морфометричні показники, включно з діаметром аксона, g-коефіцієнтом, щільністю та товщиною мієліну, а потім експортуйте результати у форматі CSV.
Використовуйте графічний інтерфейс Napari для ручного коригування масок сегментації, об’єднуючи або видаляючи маски для підвищення точності.
Важливі зауваження
- Продуктивність може знижуватися на нових або не навчених методах візуалізації
- Для складних або проблемних ділянок може знадобитися ручна корекція
- Рекомендується GPU для швидшої обробки великих наборів даних; також підтримується обробка на CPU
Часті запитання
AxonDeepSeg підтримує TEM (трансмісійна електронна мікроскопія), SEM (скануюча електронна мікроскопія) та світлову мікроскопію з попередньо навченими моделями, оптимізованими для кожної модальності.
Так, AxonDeepSeg повністю безкоштовний і з відкритим кодом, доступний для академічного та комерційного використання.
Так, AxonDeepSeg автоматично розраховує діаметр аксона, g-коефіцієнт, товщину мієліну та показники щільності на основі сегментованих зображень.
GPU рекомендується для швидшої сегментації великих наборів даних, але обробка на CPU також підтримується для менших аналізів.
Так, інтеграція з графічним інтерфейсом Napari дозволяє інтерактивно коригувати та уточнювати маски сегментації для підвищення точності на складних ділянках.
Ilastik
| Розробник | Команда Ilastik у Європейській лабораторії молекулярної біології (EMBL) та пов’язані академічні партнери |
| Підтримувані платформи |
|
| Мова | Англійська |
| Ціноутворення | Безкоштовно та з відкритим кодом |
Огляд
Ilastik — потужний інструмент на основі штучного інтелекту для інтерактивної сегментації зображень, класифікації та аналізу мікроскопічних даних. Використовуючи методи машинного навчання, такі як класифікатори Random Forest, він дозволяє дослідникам сегментувати пікселі, класифікувати об’єкти, відстежувати клітини в часі та виконувати підрахунок густоти як у 2D, так і в 3D наборах даних. Завдяки інтуїтивному інтерфейсу та зворотному зв’язку в реальному часі Ilastik доступний для науковців без навичок програмування і широко використовується в клітинній біології, нейронауці та біомедичній візуалізації.
Ключові функції
Зворотний зв’язок у реальному часі під час анотування представницьких областей для миттєвих результатів сегментації.
Категоризація сегментованих структур на основі морфологічних та інтенсивнісних ознак.
Відстеження руху та поділу клітин у 2D та 3D часових мікроскопічних експериментах.
Кількісна оцінка скупчених областей без явної сегментації окремих об’єктів.
Напівавтоматична сегментація складних 3D об’ємів з інтуїтивною взаємодією.
Автоматична обробка кількох зображень у безголовому режимі командного рядка.
Завантаження
Посібник для початку роботи
Завантажте Ilastik для вашої операційної системи з офіційного сайту. Пакет містить усі необхідні залежності Python, тому дотримуйтесь інструкцій з встановлення для вашої платформи.
Відкрийте Ilastik і оберіть робочий процес аналізу: класифікація пікселів, класифікація об’єктів, відстеження або підрахунок густоти. Завантажте ваш набір зображень, який може включати багатоканальні, 3D або часові зображення.
Позначте кілька представницьких пікселів або об’єктів на ваших зображеннях. Класифікатор Random Forest Ilastik навчається на цих анотаціях і автоматично прогнозує мітки для всього набору даних.
Застосуйте навчану модель для сегментації або класифікації всього набору даних. Експортуйте результати у вигляді позначених зображень, карт ймовірностей або кількісних таблиць для подальшого аналізу та візуалізації.
Використовуйте безголовий режим Ilastik для автоматичної обробки кількох зображень без ручного втручання, що ідеально підходить для масштабних аналітичних конвеєрів.
Обмеження та зауваження
- Інтерактивне маркування може займати багато часу для дуже великих наборів даних
- Точність залежить від якості та репрезентативності анотацій користувача
- Вимоги до пам’яті — дуже високороздільні або багатогігабайтні набори даних можуть потребувати значного обсягу оперативної пам’яті
- Складні дані — класифікатори Random Forest можуть поступатися глибоким нейронним мережам на дуже варіабельних або складних зображеннях
Часті запитання
Так, Ilastik повністю підтримує 3D об’єми та часові експерименти для сегментації, відстеження та кількісного аналізу на різних часових точках.
Так, Ilastik є повністю безкоштовним і з відкритим кодом, доступним для всіх користувачів без ліцензійних обмежень.
Ні, Ilastik має інтуїтивний графічний інтерфейс із зворотним зв’язком у реальному часі, що робить його доступним для дослідників без навичок програмування. Досвідчені користувачі також можуть використовувати пакетну обробку через командний рядок.
Так, спеціалізований робочий процес відстеження дозволяє аналізувати рух і поділ клітин у 2D та 3D часових наборах даних з автоматичним відстеженням ліній спадковості.
Результати сегментації можна експортувати у вигляді позначених зображень, карт ймовірностей або кількісних таблиць, що забезпечує безшовну інтеграцію з інструментами подальшого аналізу та програмним забезпеченням для візуалізації.
Ці інструменти підходять як для початківців, так і для експертів. Багато з них безкоштовні та з відкритим кодом, що сприяє відтворюваності та спільному використанню робочих процесів ШІ у науковій спільноті.
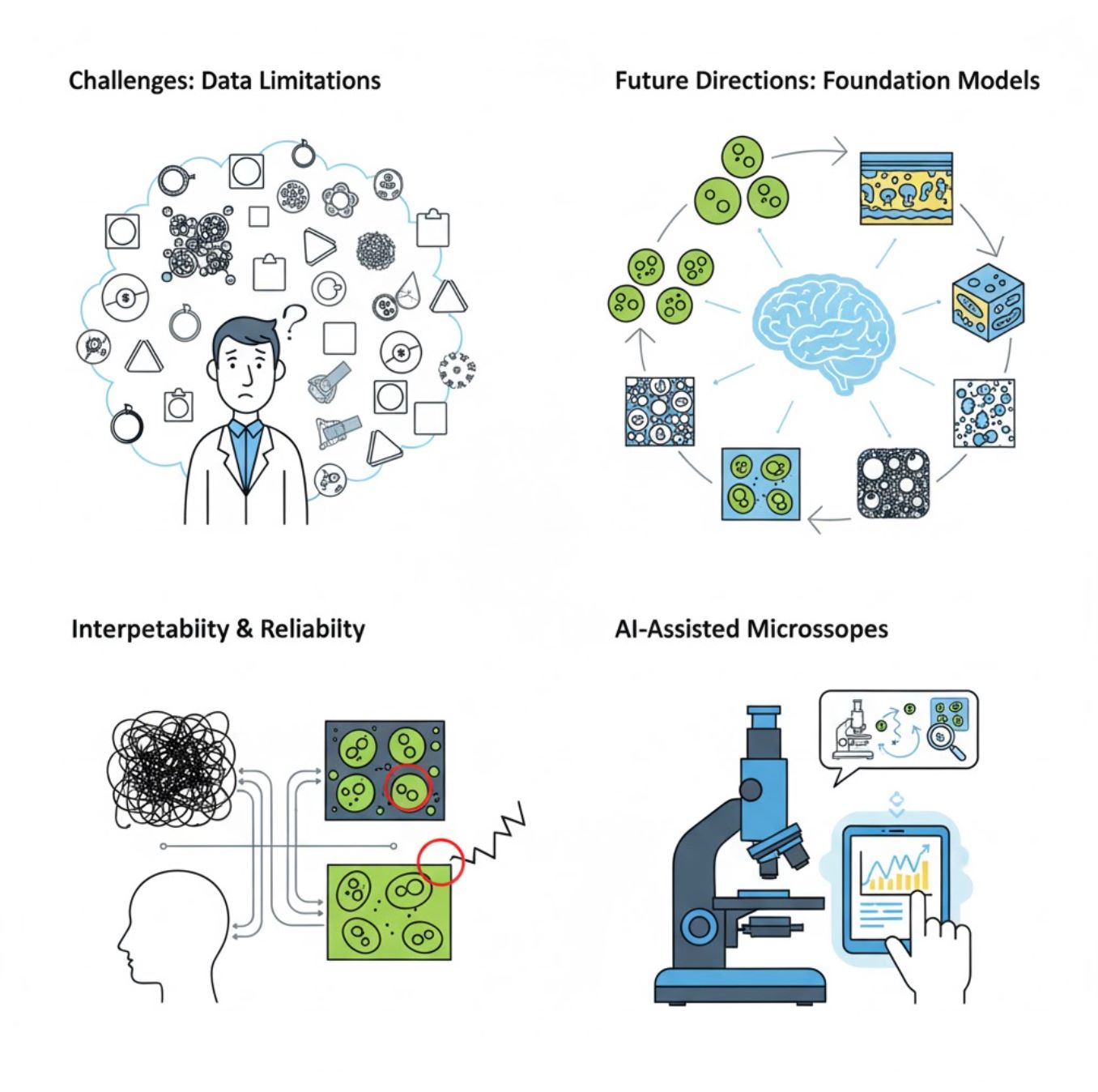
Виклики та майбутні напрямки
Поточні виклики
Нові тенденції
Фундаційні моделі зору
Системи ШІ наступного покоління обіцяють зменшити потребу у навчанні для конкретних завдань.
- Моделі на кшталт SAM та системи на основі CLIP
- Один ШІ для багатьох завдань мікроскопії
- Швидше впровадження та адаптація
Мікроскопи з підтримкою ШІ
Повністю автономні та інтелектуальні системи мікроскопії стають реальністю.
- Керування природною мовою через великі мовні моделі (LLM)
- Повністю автоматизовані цикли зворотного зв’язку
- Демократизація доступу до передової мікроскопії
Основні висновки
- ШІ швидко трансформує обробку зображень мікроскопа, підвищуючи точність і автоматизацію
- Глибоке навчання перевершує традиційне машинне навчання на складних, варіабельних мікроскопічних зображеннях
- CNN автоматично вивчають ієрархічні ознаки з сирих пікселів для надійного аналізу
- Ключові застосування включають сегментацію, класифікацію, відстеження, зниження шуму та автоматизоване отримання
- Успіх залежить від якісних даних і ретельної перевірки експертами
- Фундаційні моделі зору та мікроскопи з підтримкою ШІ — це майбутнє галузі
З подальшим розвитком і спільними зусиллями (інструменти з відкритим кодом, спільні набори даних) ШІ дедалі більше стане основним «оком» мікроскопа, допомагаючи вченим бачити невидиме.







No comments yet. Be the first to comment!